Статья на конкурс «био/мол/текст»: Микробиом человеческого кишечника представляет собой уникальную совокупность микроорганизмов. Его незримое присутствие опосредует целый ряд важных процессов: от метаболических и иммунных до когнитивных, а отклонение его состава от нормы приводит к развитию разнообразных патологических состояний: аллергических и аутоиммунных заболеваний, сахарного диабета, ожирения и др. Качественный и количественный состав микробиома, от которого во многом зависит будущее здоровье человека, определяется во младенчестве. Процессам его формирования и будет посвящена эта статья.
В настоящее время понятие «микрофлора человека» утратило свою актуальность: мы несем в своем организме не просто набор бактерий, а настоящий биом — микробиом . Но если биом с точки зрения экологов представляет собой крупную экосистему, то наше тело — это место обитания многочисленной популяции микроорганизмов, своего рода микробная экосистема, характеризующаяся своей генетической регуляцией и сложными взаимодействиями и реагирующая на влияние факторов внешней и внутренней среды. Она настолько уникальна, что в мире не найдется двух людей с идентичным микробиомом. Различия в микробном составе зависят от таких факторов среды, как набор питательных веществ, рН, влажность и температура. Те или иные их значения способствуют размножению бактерий и опосредуют пользу, которую они могут принести хозяину — человеку [1], [2].
Утрачивает актуальность оно еще и потому, что современный уровень знаний позволяет выражаться более корректно, и всё чаще «микрофлору» заменяют термином «микробиота» (наряду с «микробиомом»), подчеркивая, что бактерии и грибы (ну и вирусы с простейшими тоже) никак к «флоре», то есть миру растений, не относятся. Однако многие микробиологи «микрофлору» спокойно помещают в научные статьи и не кидаются друг в друга тухлыми помидорами: ничего особо ужасного в этом традиционном термине нет. — Ред.
Микробиом распределен в нашем организме неравномерно, по его топографии и видовому составу принято различать микробиом кожи, полости рта, дыхательных путей, урогенитального тракта и кишечника. Наиболее крупным микробиомом нашего тела является, несомненно, кишечный. Он может состоять из сотен видов различных микроорганизмов, но у взрослого человека преобладают бактерии двух типов: Firmicutes и Bacteroidetes [3]. Кишечный микробиом изучен лучше других бактериальных сообществ человека, и многолетние исследования, о которых будет рассказано ниже, показали, что именно он в большей степени влияет на здоровье своего носителя. Давайте же совершим путешествие в прошлое и разберем, как формируется микробиом нашего кишечника.
Микробиом: начало
Еще до недавнего времени считалось, что плод в утробе матери полностью огражден от контакта с миром микроорганизмов, то есть человек рождается полностью стерильным, а его заселение бактериями происходит позже. Но появились данные о том, что первые колонизаторы осваивают организм человека еще до его рождения. В ряде исследований было выявлено, что в плаценте, околоплодных водах, пуповинной крови и первичном кале — меконии — присутствуют бактерии родов Enterococcus, Escherichia, Leuconostoc, Lactococcus и Streptococcus, а у недоношенных младенцев — следы Enterobacter, Enterococcus (в меньшей степени, чем у доношенных), Lactobacillus, Photorhabdus и Tannerella [4–14]. Также в одной из работ были получены доказательства внутриутробной бактериальной транслокации — проникновения бактерий из кишечника матери к плоду [13]. Исследователи предполагают, что это происходит посредством кровотока: механизмом, сходным с «энтеромаммарной осью», о которой поговорим чуть позже. Эту гипотезу поддерживают данные другого эксперимента, в котором беременные мыши перорально получали меченых Enterococcus faecium, после чего эти бактерии «заявляли о себе» в плаценте и даже меконии еще не рожденных мышат [13].
Но всё же по-настоящему серьезный контакт с миром микроорганизмов происходит после рождения, и во многом от того, как пройдет эта встреча, зависит будущее здоровье человека. Колонизация кишечника у здоровых детей укладывается в четыре последовательные временны́е фазы. Первая длится от момента рождения до двух недель. Микробная популяция в этот период представлена в основном стрептококками и кишечной палочкой. В зависимости от вида вскармливания — грудного или искусственного — через некоторое время присоединяются бифидо- или лактобактерии соответственно. В небольших количествах обнаруживаются и представители родов Clostridium и Bacteroides. Через две недели начинается вторая фаза, которая продолжается до введения в рацион прикорма. В это время увеличивается численность представителей рода Bacteroides. С момента введения прикорма начинается третья фаза, длящаяся до завершения грудного вскармливания. В эту фазу окончательно формируется микробиом ребенка: постепенно, по мере увеличения в рационе доли твердой пищи и снижения доли грудного молока, растет количество бактероидов и анаэробных грамположительных кокков (пептококков и пептострептококков). Окончание грудного вскармливания знаменует переход к четвертой фазе. Она характеризуется относительной стабильностью микробного состава, который сохраняется в течение всей жизни индивида [15].
А теперь рассмотрим подробнее, как протекают процессы колонизации и становления иммунного ответа во время и после рождения.
Родоразрешение
Как оказалось, даже характер родоразрешения (ребенок может появиться на свет естественным путем, а может и оперативным, с помощью кесарева сечения) влияет на состав микробиома младенца.
Первое, с чем сталкивается ребенок, — микробный мир родовых путей его матери. У небеременных женщин идентифицировано шесть видов лактобактерий, исходя из соотношения которых выделяют по крайней мере пять качественно различных типов микробиома влагалища [16]. В четырех из них — характерных, как правило, для представительниц европейских и азиатских народов — преобладают следующие представители рода Lactobacillus:
- L. crispatus (I тип микробиома);
- L. gasseri (II тип микробиома);
- L. iners (III тип микробиома);
- L. jensenii (V тип микробиома).
IV тип микробиома часто встречается у чернокожих и латиноамериканских женщин и характеризуется низким уровнем Lactobacillus spp. и большим числом анаэробных бактерий.
При беременности из-за изменения pH влагалища бактериальное разнообразие уменьшается, но повышается стабильность состава микробиоты. Как правило, в этот период в микробиоме преобладают Lactobacillus crispatus и Lactobacillus iners. Количественное превосходство этих видов подчеркивает их важность для поддержания здоровой среды родовых путей. У европейских и азиатских женщин во время беременности могут происходить сдвиги между типами микробиомов, но, как правило, они редко переходят к IV типу [18]. Таким образом, в зависимости от особенностей микробиоты влагалища матери ребенок начинает свою жизнь со «знакомства» с определенным видовым набором микроорганизмов, что подчеркивает важность исследований микробиомов различных групп населения.
Но во что же выливается это знакомство? Почему вообще заселение микробами так важно для ребенка? Дело в том, что бактерии за счет симбиотических взаимоотношений с эпителиальными и иммунными структурами кишечника фактически активируют иммунную систему хозяина (рис. 1) [21].
Покидая родовые пути естественным образом, доношенный новорожденный в небольших количествах заглатывает представителей вагинальной и кишечной микробиоты матери. В основном это бактерии родов Prevotella, Sneathia и Lactobacillus [15]. Если же родоразрешение происходит путем кесарева сечения, одними из первых колонизируют организм новорожденного представители кожных микробиомов матери и медицинского персонала, в основном — бактерии родов Propionibacterium, Corynebacterium и Streptococcus [19], [20]. У таких младенцев отмечают замедление заселения кишечника филой Bacteroidetes и низкое бактериальное разнообразие в течение первых двух лет жизни [22]. Однако с четырех месяцев различия в бактериальном разнообразии с естественно рожденными детьми начинают стираться, и к 12 месяцам практически исчезают [19].
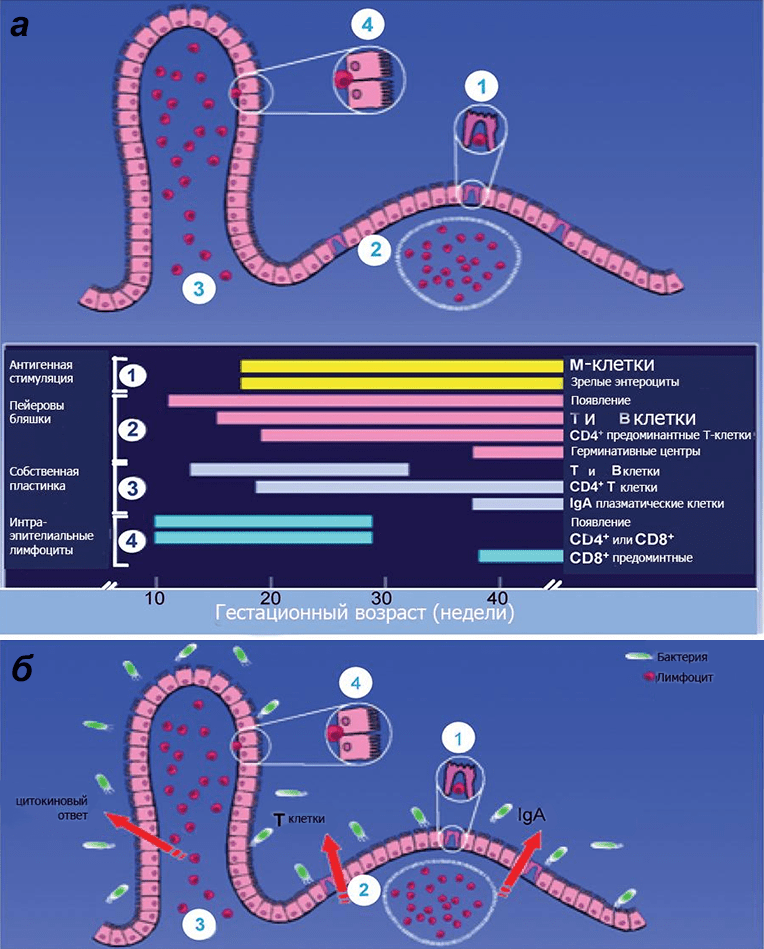
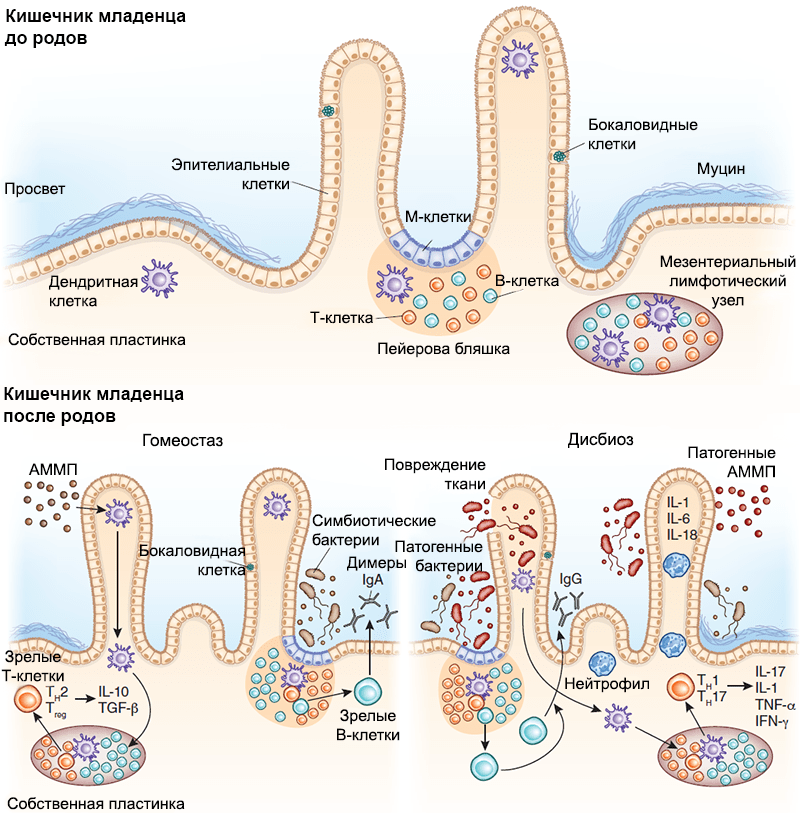
Рисунок 1. Схема иммунной защиты слизистой оболочки кишечника плода в зависимости от гестационного возраста. а — У младенца, рожденного в срок, все компоненты иммунной защиты слизистой оболочки являются зрелыми. б — Однако чтобы иммунная система приобрела работоспособность, должна произойти ее стимуляция первичными колонизаторами. Иммунная защита кишечника включает в себя: специализированный эпителий, или М-клетки (1), пейеровы бляшки (2), интерстициальные (3) и интраэпителиальные (4) лимфоциты, опосредующие развитие иммунных реакций. Среди лимфоидных образований кишечника выделяют одиночные лимфатические узлы (расположены преимущественно в дистальных отделах кишечника) и пейеровы бляшки (расположены в основном в подвздошной кишке). Последние образованы сгруппированными лимфатическими фолликулами, выпячивающими эпителий в просвет кишки в виде купола, и межузелковыми скоплениями лимфоидной ткани. Эпителий слизистой оболочки в области бляшки содержит до 10% специфических микроскладчатых (М-) клеток, обеспечивающих трансэпителиальный транспорт, механизм которого — трансцитоз: благодаря тонкому гликокаликсу они активно поглощают апикальной поверхностью макромолекулы (в том числе антигены) из просвета кишки, в составе эндосом перемещают их через свою цитоплазму и посредством экзоцитоза передают иммунокомпетентным клеткам пейеровых бляшек. Для ускорения процесса М-клетки снизу образуют «карманы», заполняющиеся «иммунным винегретом» из В-клеток, плазмоцитов, Т-клеток, макрофагов и антигенпрезентирующих дендритных клеток.
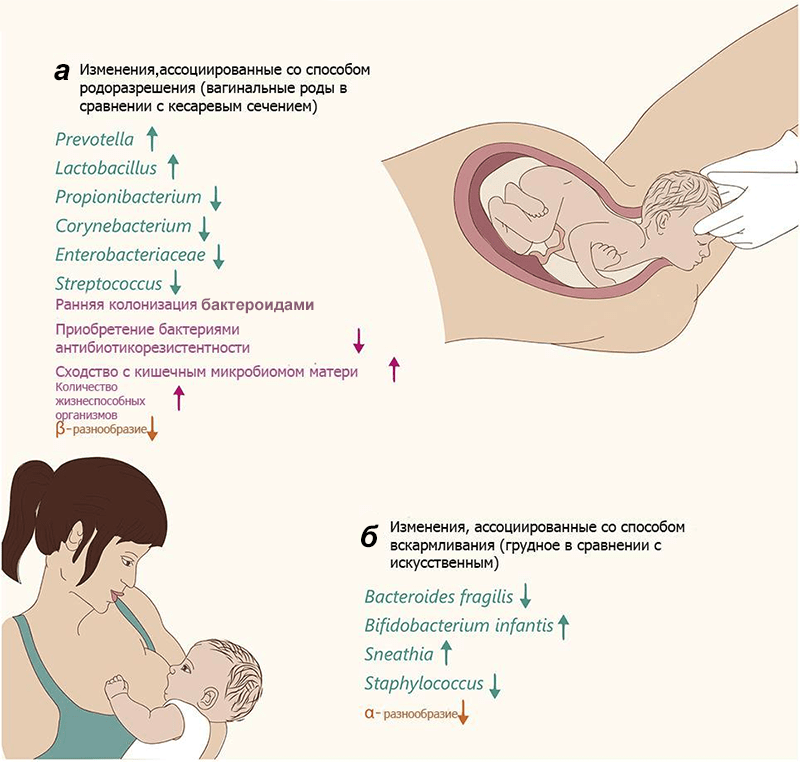
Тем не менее очевидно, что характер родоразрешения влияет на микробиом новорожденного (рис. 2). Правда, пока неясно, сказываются ли эти различия на здоровье взрослого индивида: хотя некоторые эпидемиологические исследования демонстрируют связь между кесаревым сечением и различными заболеваниями (табл. 1), причина их развития окончательно не выяснена.
Рисунок 2. Изменения микробиома у новорожденных. Цвет текста и стрелки обозначают изменения специфических разновидностей (зеленый), общие изменения (розовый) и разнообразие сообщества (оранжевый). а — Изменения у младенцев, родившихся естественным путем, относительно извлеченных хирургически. б — Изменения у младенцев, находящихся на грудном вскармливании, относительно «искусственников». α-разнообразие — видовое разнообразие внутри исследуемого сообщества, β-разнообразие — видовое разнообразие между сообществами данной области (по Р. Уиттекеру).
Грудное вскармливание
Не менее важным фактором в формировании микробиома новорожденного является характер питания. Грудное молоко — оптимально сбалансированная пища для младенца, обеспечивающая его нормальное развитие [23]. Как известно, в первые дни жизни именно оно защищает ребенка от инфекционных болезней и способствует снижению смертности от них [24] за счет содержания множества иммунных факторов: Т- и В-лимфоцитов, плазматических клеток, иммуноглобулинов (в первую очередь IgА) и антимикробных ферментов (лизоцима и лактоферрина). Установлено, что грудное вскармливание в какой-то мере предотвращает развитие таких хронических заболеваний, как сахарный диабет [25] и ожирение [26]. И, несомненно, грудное молоко способствует формированию «здорового» микробиома. Первые данные об обнаружении бактерий в женском молоке были получены в 1950 году, но исследователей тех лет интересовали лишь патогенные микроорганизмы [27]. И хотя уже в 70-х годах в грудном молоке были найдены первые бактерии-симбионты, ученым не удалось их идентифицировать [27]. Лишь в 2003 году, когда в молоке, на коже груди и ареолах сосков восьми кормящих матерей и в ротовой полости и фекалиях их детей были обнаружены родственные лактобациллы, ученые предположили, что эти бактерии не занесены случайно извне, а имеют эндогенное происхождение [28].
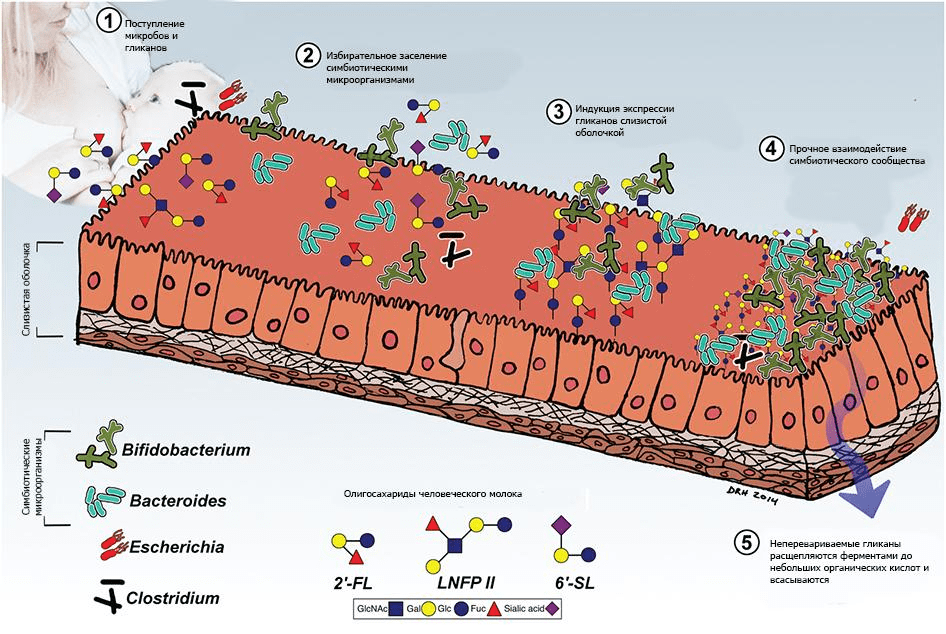
Независимо от того, материнское или донорское грудное молоко используется для вскармливания, у каждого младенца формируется свой особый микробиом. Заслуга в этом принадлежит олигосахаридам молока человека (ОМЧ) [29], [30]. В составе женского молока около 8% отведено перевариваемым ОМЧ — пребиотикам, поддерживающим рост Bifidobacterium longum subsp. infantis [31]. При этом профиль ОМЧ у каждой женщины уникален, что обеспечивает, в свою очередь, индивидуальность младенческого микробиома (рис. 3) [32].
Рисунок 3. Взаимодействие гликанов человеческого молока и микробиоты. Взаимодействие новорожденного с материнскими и содержащимися в окружающей среде микроорганизмами опосредовано потреблением молозива и содержащихся в нём гликанов (олигосахаридов молока человека, ОМЧ). ОМЧ обладают пребиотической, антиадгезионной и противовоспалительной активностью, облегчают экспансию симбионтов — в особенности Bacteroides и Bifidobacterium — и ингибируют рост и адгезию оппортунистических и облигатных патогенов.
Другим опосредующим формирование микробиома фактором является так называемая энтеромаммарная ось — система, которая обеспечивает транспорт бактерий из кишечника (будущей) матери в молочные железы. Ее первичным звеном служат дендритные клетки кишечника, которые захватывают бактерий и транспортируют их в местные лимфоидные фолликулы [1]. Там осуществляется выработка специфического иммуноглобулина А. Эти дендритные клетки и секретирующие иммуноглобулин лимфоциты циркулируют в крови, но могут избирательно возвращаться в кишечник за счет взаимодействия между β7-интегринами и выделяемыми эндотелиоцитами молекулами адгезии (адрессинами, MAdCAM-1). Клетки эндотелия молочных желез синтезируют молекулы MAdCAM-1 во время беременности, обеспечивая избирательное поступление в железу «запрограммированных» дендритных клеток, содержащих кишечные бактерии [33]. Помимо бактерий, в молозиве и молоке матери содержатся Т-клетки, производящие β7-интегрины, и плазматические клетки, вырабатывающие специфический IgА [34]. Также в молоке находятся цитокины, состав которых зависит от иммунологического опыта матери, приобретенного в течение жизни.
Существует теория, предполагающая передачу микроорганизмов из полости рта младенца в молочную железу матери с последующей выработкой в ее организме специфических антител и их поступлением в ЖКТ ребенка [34]. Предположение подтверждается тем фактом, что в ротовой полости новорожденного и в грудном молоке обнаруживаются идентичные бактерии родов Gemella, Veillonella, Staphylococcus и Streptococcus [35], [36]. Хотя есть данные, что они присутствуют в молозиве еще до начала грудного вскармливания (даже после тщательной гигиенической обработки железы образцы сцеженного молока содержат бактерии кожного и кишечного микробиома матери [37]), это всё же не отменяет возможность реализации механизма обратного заноса.
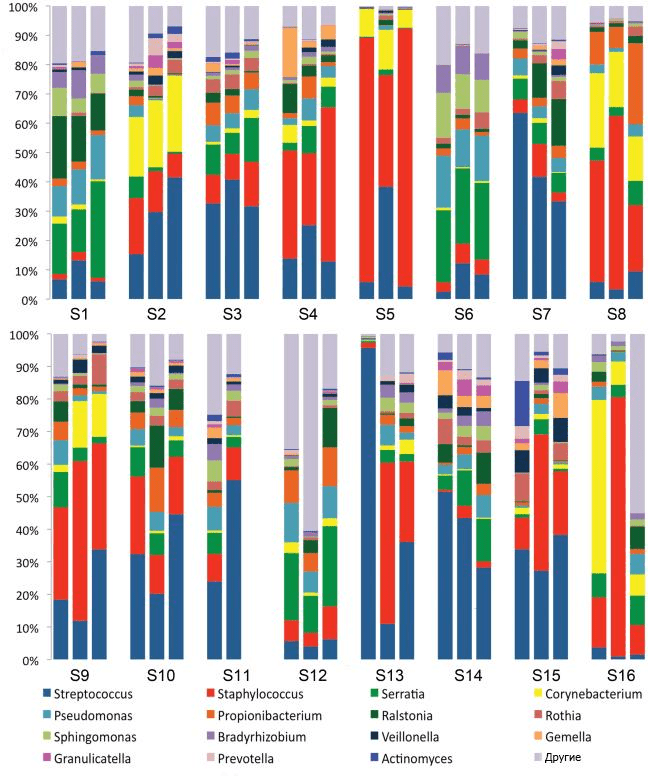
В 2011 году ученые из США обнаружили девять таксономических единиц бактерий в образцах молока от 16 женщин. Установив тем самым, что микробный состав молока чрезвычайно разнообразен, они предложили концепцию «ядра микробиома женского молока» (рис. 4) [35]. В дальнейшем стало ясно, что микробиом изменяется в течение всего периода лактации. Так, в молозиве преобладают бактерии родов Weissella, Leuconostoc, Staphylococcus, Streptococcus и Lactococcus, а в молоке, вырабатываемом в первые шесть месяцев после родов, видимо, из-за частого контакта с обитателями полости рта младенца происходит «перекос» в сторону Veillonella, Leptotrichia и Prevotella — что опять же подтверждает теорию обратного заноса [37].
Рисунок 4. Девять бактериальных таксономических единиц, обнаруженных в образцах молока от 16 женщин в 2011 году. Наблюдаемые сообщества оказались достаточно сложными, и при относительном постоянстве состава у одной группы испытуемых у другой с течением времени отмечались изменения.
Здоровье матери также играет важную роль в формировании микробного состава молока. В первый месяц лактации в молоке страдающих ожирением женщин преобладают Lactobacillus, однако через полгода их сменяют представители рода Staphylococcus [37], которые, как показывают исследования, начинают преобладать и в кишечнике тучных младенцев, в связи с чем ученые предположили существование связи между особенностями микробиома женского молока и паратрофией у детей.
Постепенное отнятие младенца от груди и переход на твердую пищу также играют важную роль в формировании бактериального разнообразия. После завершения грудного вскармливания в микробиоме детского кишечника появляются характерные для взрослого бактерии — представители типов Bacteroidetes, Firmicutes и класса Clostridia: Clostridium, Ruminococcus, Faecalibacterium, Roseburia, и Anaerostipes [19], [38].
Иммунитет
Недавние исследования подтвердили важную роль кишечной микрофлоры в развитии иммунной защиты. В Nature Reviews Immunology был опубликован обзор, подчеркнувший важный вклад бактерий-комменсалов в функционирование эпителиального барьера кишечника (рис. 5).
Рисунок 5. Эпителиальный барьер кишечника. Простой цилиндрический эпителий обладает механизмами физической и биохимической адаптации к микробной колонизации, поддерживающими целостность барьера. К ним относятся: богатые актином микроворсинки; плотные контакты эпителиальных клеток (а); муцины, которые формируют «сито» для просеивания молекул — гликокаликс; продукция различных антимикробных пептидов. М-клетки, покрывающие пейеровы бляшки и одиночные лимфоидные фолликулы, обеспечивают транслокацию молекул из просвета кишки под эпителий, к антигенпредставляющим клеткам. Дендриты специализированных дендритных клеток (ДК) могут проникать в просвет кишечника через плотные контакты (б).
Ученые предположили, что бактерии кишечника не только стимулируют его лимфоидные элементы, но и оказывают влияние на кишечный слизистый барьер, стимулируя образование микроворсинок [39], [40] и плотных контактов [41].
Рисунок 6. Взаимодействие между иммунной системой кишечника и микробиомом младенца. Развитие вторичных лимфоидных структур, в том числе пейеровых бляшек и одиночных лимфоузлов, происходит внутриутробно, задолго до начала бактериальной колонизации. С ее началом настраиваются механизмы взаимодействия иммунной системы хозяина и бактерий-симбионтов. М-клетки путем трансцитоза передают бактериальные антигены дендритным клеткам, те их презентируют, опосредуя Т-зависимое созревание В-лимфоцитов и способствуя секреции плазматическими клетками IgA, который играет важную роль в защите от патогенов. Бактерии могут транслоцироваться также через дендритные клетки и презентироваться Т-клеткам лимфоузла, индуцируя их дифференцировку. Нижняя левая панель — АММП — ассоциированный с микроорганизмами молекулярный паттерн. В условиях гомеостаза АММП, ассоциированные с бактериями-симбионтами, стимулируют продукцию регуляторных цитокинов (IL-25, IL-33, тимусного стромального лимфопоэтина и трансформирующего фактора роста, TGF-β). Трансдукция сигнала на дендритные клетки стимулирует развитие регуляторных Т-клеток и способствует секреции IL-10. Нижняя правая панель — В состоянии дисбиоза снижение количества бактерий-симбионтов приводит к размножению патогенов. Патогенные АММП индуцируют секрецию провоспалительных цитокинов (IL-1, IL-6 и IL-18), способствуя размножению эффекторных Т-клеток. Эти Т-клетки дифференцируются в CD4+ Th1 и Th17 и секретируют IL-17, фактор некроза опухоли (TNF-α) и интерферон-γ (IFN-γ), которые привлекают в очаг воспаления нейтрофилы, защищая организм хозяина от патогенов.
Исследуя влияние бактерий на защитные механизмы кишечника (рис. 6), ученые искусственно колонизировали бактерией Bacteroides thetaiotaomicron кишечники безмикробных мышей. Затем РНК кишечного эпителия анализировали на предмет изменения экспрессии генов [42]. Была отмечена обширная активация генов эпителиоцитов, которые регулировали функцию эпителиального барьера и способствовали повышению продукции рецептора к IgА. Это исследование прекрасно отражает влияние бактериальной колонизации на кишечник новорожденного, ведь он в данном контексте такой же, практически безбактериальный, организм.
Всё новые работы подчеркивают важную роль бактериальной колонизации в формировании и поддержании здоровья млекопитающих. В недавнем эксперименте, посвященном изучению функций Toll-подобных рецепторов, нокаутировали ген важного компонента врожденного иммунитета — рецептора TLR5, расположенного на базолатеральной поверхности энтероцитов мышей. Это повлекло за собой следующее: мыши начали регулярно объедаться и в конце концов развили метаболический синдром, сопровождавшийся изменением состава кишечной микробиоты. Возникло предположение, что микробиом может служить индикатором развития многих заболеваний. Но ученые пошли дальше и пересадили «патологическую» микробиоту из TLR5-дефицитных особей безмикробным мышам с нормальным рецептором, и у тех тоже проявились признаки метаболического синдрома. То есть микробиом, возможно, может служить не только индикатором системных проблем, но и непосредственно участвовать в их возникновении. Интересно, что ограничение питания TLR5-дефицитных мышей предотвращало развитие ожирения, но не резистентности к инсулину [43].
Вообще, связь колонизации бактериями-симбионтами с развитием как приобретенного, так и врожденного иммунитета, демонстрировали неоднократно. Было установлено, что взаимодействие рецепторов энтероцитов и иммунных клеток кишечника с антигенами микроорганизмов вызывает естественную, самоограничивающуюся воспалительную реакцию. Таким способом механизмы врожденного иммунного ответа позволяют предотвратить проникновение патогенов через эпителиальный барьер кишечника, при этом отличая их от безвредных симбионтов (рис. 6) [44], [45]. Когда ребенок покидает утробу матери, происходит контакт с огромным количеством бактерий. И для того чтобы избежать непрерывной воспалительной реакции в ответ на колонизацию кишечника, снижается экспрессия упомянутых рецепторов, в частности TLR2 и TLR4 [46]. К сожалению, у детей, появившихся на свет раньше срока, описанные механизмы еще незрелые, что часто приводит к развитию некротического энтероколита [47].
Исследования последних лет показали, что кишечные бактерии могут вносить свой вклад и в развитие иммунной толерантности, то есть защиты от избыточных иммунных реакций. Эти наблюдения чрезвычайно актуальны для разработки возможных терапевтических вмешательств, ведь многие бактериальные виды имеют количественное преимущество лишь на ранних этапах колонизации, во время грудного вскармливания (см. главу «Микробиом и грудное вскармливание»). Как же симбионтов связали с иммунотолерантностью? Например, полисахарид А капсулы Bacteroides fragilis может взаимодействовать с TLR2-рецепторами дендритных клеток кишечника, активируя продукцию противовоспалительных цитокинов, которые создают для бактерий специфическую микросреду [48]. Некоторые виды клостридий подобным образом увеличивают количество регуляторных Т-клеток — контролеров, подавляющих иммунный ответ, если Т-эффекторы неоправданно разбушевались, — предотвращая тем самым IgE-опосредованные заболевания [49]. Таким образом, конкретные микроорганизмы, представленные в материнском молоке, еще во младенчестве могут предопределить степень «аллергичности» индивида: упомянутые механизмы позволяют выработать толерантность к «полезным» антигенам и бактериям, предотвращая в дальнейшем развитие аллергий и аутоиммунных заболеваний .
Дополнительно о связях микробиома с иммунитетом, питанием, болезнями и препаратами для их лечения можно прочитать на «биомолекуле»: «Зоопарк в моем животе» [50].
И другие…
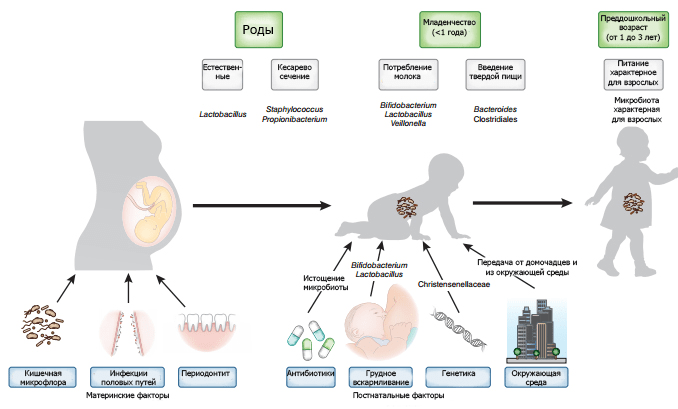
Помимо генетических факторов, характера родоразрешения и вскармливания, на формирование микробиома и иммунитета у новорожденного в той или иной мере оказывают влияние особенности питания, опыт приема антибиотиков и факторы окружающей среды (рис. 7).
Рисунок 7. Факторы, обеспечивающие формирование микробиома младенца. Инфекции половых путей женщины могут привести к бактериальному загрязнению матки. Микрофлора кишечника и ротовой полости может транспортироваться с кровью к плоду. Характер родоразрешения формирует первичную микрофлору. Генетика и постнатальные факторы, такие как режим питания, использование антибиотиков и воздействие окружающей среды оказывают дополнительное влияние на микробиом.
Антибиотики — одни из наиболее часто выписываемых детям препаратов. Назначение их матери в послеродовом периоде или же новорожденному может нарушить хрупкие процессы, которые лежат в основе формирования микробиома, и стать причиной ряда заболеваний (табл. 1). Исследования последних лет регулярно подчеркивают важность понимания процессов, ведущих к неонатальному дисбиозу и развитию в дальнейшем таких патологий, как диабет II типа, воспалительные заболевания кишечника или аллергическая реакция на компоненты молока [51–56]. Изменения в микробиоме, провоцируемые антибиотиками, зависят от способа введения, мишени, типа и дозировки препарата. Всё это пока плохо изучено у младенцев, что затрудняет понимание влияния антибиотикотерапии на формирование нормальной микрофлоры.
| Фактор, вызывающий дисбаланс | Характеристика когорты | Исходы |
|---|---|---|
| Кесарево сечение | 1,9 млн датских детей в возрасте 0–15 лет | Астма, системные заболевания соединительной ткани, ювенильный ревматоидный артрит, воспалительные заболевания кишечника, иммунодефициты и лейкозы |
| 1255 трехлетних детей из США | Ожирение, высокий ИМТ | |
| 2803 норвежских ребенка 0–3 лет | Аллергическая реакция на куриные яйца, рыбу или орехи | |
| Антибиотикотерапия | 1401 ребенок 0–6 месяцев из США | Астма и аллергия |
| 5780 британских детей 0–2 лет | Астма и экзема | |
| 12 062 финских ребенка 0–2 лет | Избыточный вес и ожирение | |
| 162820 детей 2–18 лет из США | Избыточный вес | |
| 9 млн британских детей | Воспалительные заболевания кишечника | |
| Пробиотики | 215 испанских детей 0–6 месяцев | Снижение частоты инфекций ЖКТ и верхних дыхательных путей |
| Европейское общество специалистов в области детской гастроэнтерологии, гепатологии и питания, комиссия по вопросам питания | Снижение частоты неспецифических желудочно-кишечных инфекций | |
| Пищевые добавки | 139 африканских детей 6–14 лет | С большей частотой — воспалительные заболевания кишечника, с меньшей — колики |
| Гигиена | 184 ребенка 0–3 лет (исследование чистоты пустышек) | Чистота пустышек снижала риск развития астмы, аллергии и сенсибилизации |
| Домашние животные | 3143 финских ребенка 0–1 года | Снижение риска развития диабета I типа |
Отдельно стоит сказать о пробиотиках и пребиотиках, которыми повсеместно обогащены смеси для искусственного вскармливания, несмотря на недоказанность их эффективности [57]. Пробиотики представляют собой живые микроорганизмы, которые, предположительно, должны участвовать в формировании микробиома, а пребиотики — это вещества, которые способствуют росту полезных микроорганизмов. Использование пробиотиков в педиатрической практике — до сих пор спорный момент, хотя их влияние на различные детские заболевания изучалось достаточно широко. Так, некоторые мета-анализы обнаружили их эффективность в терапии атопического дерматита, в то время как другие не выявили существенного влияния на детей младше 12 месяцев [58–61]. Была проведена оценка воздействия самых популярных пробиотических добавок в детском питании (Lactobacillus и Bifidobacterium spp., L. reuteri) на колики у младенцев. После трех недель их применения в кишечнике новорожденных увеличивалось количество лактобацилл и уменьшалось содержание кишечной палочки [62]. Однако на сегодняшний день большинство исследований показывает, что пре- или пробиотики значительно не влияют на качественный и количественный состав микробиома кишечника.
Окружающая среда не стерильна, и предметы быта, с которыми сталкивается новорожденный, тоже служат источниками микроорганизмов, участвующих в формировании микробиома. К примеру, вероятность обмена бактериями посредством предметов быта и воздуха в помещении увеличивается соразмерно количеству людей, проживающих в доме. Исследование 60 семей из США выявило, что члены одной семьи (домовладения) имеют более схожие микробиомы, чем члены разных семей [63]. Особенно показательно максимальное сходство микробиоты кожи у супругов, а также обмен поверхностными бактериальными сообществами между хозяевами и их собаками. Частый контакт с компонентами домашней пыли и большая семья в первые два месяца жизни могут привести к изменениям в микробиоме, ассоциированным с возникновением аллергии. Они заключаются в увеличении у младенцев количества бифидобактерий (за исключением B. adolescentis) и снижении численности Lactobacillus spp., Bifidobacterium adolescentis и Clostridium difficile [64].
Вероятно, частый контакт с животными, а соответственно, и с их микробиотой, на первом году жизни оказывает защитное действие, повышая иммунную толерантность. Например, взаимодействие с домашними животными с раннего возраста снижает риск развития аллергических состояний и астмы, но механизмы этого явления пока в полной мере не выявлены [65]. Ну и после всего сказанного, конечно, не станет сюрпризом вывод ученых о том, что бактериальное разнообразие гораздо выше у сельских детей, чем у городских [66]. В заключение стόит отметить, что ряд бактериальных штаммов обнаруживается одновременно у матерей и их взрослых дочерей, из чего следует, что одни бактерии временно «арендуют» жилплощадь, а другие получают в макроорганизме пожизненную «прописку».
Коррекция
В недавно проведенных исследованиях изучались терапевтические вмешательства, которые могли бы изменять микробом и профилактировать микробный дисбаланс еще в раннем детстве.
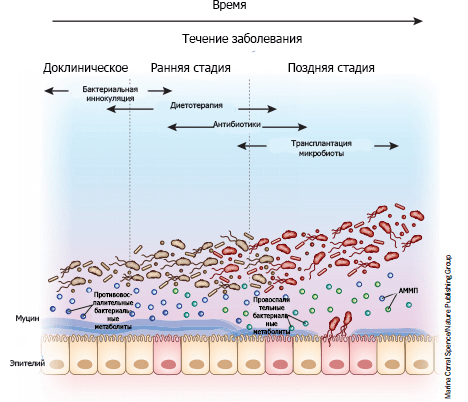
Подходы к модификации микробиома, как правило, подразделяются на три основные группы: очищение от микроорганизмов, их модуляция и замена, восстановление микробиоты. К примеру, антибиотики из-за их способности эффективно очищать кишечник от бактерий часто используют для лечения состояний, вызванных неспецифической патогенной микробиотой (рис. 8).
Рисунок 8. «Микробная терапия» в зависимости от стадии заболевания. На доклинических стадиях болезнь еще не проявляется в полной мере: ее симптомы (если вообще они есть) минимальны и неспецифичны, но тонкие биологические изменения уже происходят. Использование культур определенных бактериальных сообществ на ранних стадиях болезни может с максимальной эффективностью предотвратить дисбиоз и развитие патологии. По мере прогрессирования заболевания происходит обогащение микробиома патогенами (изображены красным), продуцирующими провоспалительные метаболиты и тем самым активирующими воспалительные пути (рис. 6). Слизистый слой, защищенный эпителием, истончается по мере накопления патогенов, утяжеляя течение заболевания. Диетотерапия и антибиотики на этом этапе могут использоваться в качестве радикальной меры, изменяющей количественный и качественный состав бактерий. На поздних стадиях продолжающееся истончение слизистого слоя позволяет бактериям прорваться через эпителиальный барьер. Тогда агрессивная антибиотикотерапия в сочетании с трансплантацией микробиоты может помочь восстановить микробный баланс. АММП — ассоциированный с микроорганизмами молекулярный паттерн.
Однако у детей раннего возраста длительное применение антибиотиков чревато значительным риском осложнений. Состав микробиоты можно изменить и посредством диетотерапии, употребляя продукты, которые способствуют росту «полезных» микроорганизмов. Одним из примеров подобной диеты является особое энтеральное питание, применяемое для симптоматической терапии болезни Крона у детей. Оно представляет собой специфическую смесь всех необходимых микро- и макронутриентов и потому в течение длительного времени может служить единственным источником питательных веществ. Оно подается исключительно в жидком виде — перорально либо через зонд — и способствует достижению клинической ремиссии за счет создания своеобразного «режима покоя»: снижения функциональной нагрузки на воспаленный кишечник и уменьшения его травматизации [67].
Исследования в области дефицита питания продемонстрировали, что на сегодняшний день невозможно, используя только диетотерапию, существенно повлиять на состав кишечного микробиома, хотя изучение влияния характера питания на него продолжается.
Учитывая то, какое значительное воздействие на здоровье оказывает микробиота в раннем детстве, весьма актуальными выглядят методы, направленные на своевременное, профилактическое, восстановление микробного баланса. Недавно удалось показать, что микробиом новорожденных, появившихся на свет посредством кесарева сечения, можно восстановить до состояния, схожего с младенцами, родившимися естественным путем. Обтирание таких детей тампонами, которые были введены за час до кесарева сечения во влагалище матери, приводило к значительному обогащению их микробиома представителями Lactobacillus и Bacteroides. Правда, пока не выяснены возможные последствия такой процедуры для здоровья [68].
Микробиом кишечника можно рассматривать как целый отдельный орган нашего тела. Мы приобретаем его при рождении, и то, каким он будет, зависит от множества факторов. Но одно можно сказать наверняка: он будет непохожим ни на какой другой микробиом. Это практически такой же уникальный признак, как папиллярные линии и сосудистый рисунок сетчатки глаза. И подобно тому, как отпечатки пальцев могут рассказать следователям криминальную биографию преступника, микробиом кишечника может предъявить ученым вехи онтогенеза своего хозяина. И как нельзя лучше процесс бактериальной колонизации и его значимость для организма отражает знаменитая пословица: «Что посеешь, то и пожнешь». Берегите свой микробиом и будьте здоровы!
Литература
- Hooper L.V., Littman D.R., Macpherson A.J. (2012). Interactions between the microbiota and the immune system. Science. 336, 1268–1273;
- Mayer E.A., Knight R., Mazmanian S.K., Cryan J.F., Tillisch K. (2014). Gut microbes and the brain: paradigm shift in neuroscience. J. Neurosci. 34, 15490–15496;
- Rajilic-Stojanovic M., Heilig H.G., Molenaar D., Kajander K., Surakka A., Smidt H., de Vos W.M. (2009). Development and application of the human intestinal tract chip, a phylogenetic microarray: analysis of universally conserved phylotypes in the abundant microbiota of young and elderly adults. Environ. Microbiol. 11, 1736–1751;
- Ardissone A.N., de la Cruz D.M., Davis-Richardson A.G., Rechcigl K.T., Li N., Drew J.C. et al. (2014). Meconium microbiome analysis identifies bacteria correlated with premature birth. PLoS One. 9, e90784;
- Moles L., Gomez M., Heilig H., Bustos G., Fuentes S., de Vos W. et al. (2013). Bacterial diversity in meconium of preterm neonates and evolution of their fecal microbiota during the first month of life. PLoS One. 8, e66986;
- Rautava S., Kainonen E., Salminen S., Isolauri E. (2012). Maternal probiotic supplementation during pregnancy and breast-feeding reduces the risk of eczema in the infant. J. Allergy Clin. Immunol. 130, 1355–1360;
- Dominguez-Bello M.G., Costello E.K., Contreras M., Magris M., Hidalgo G., Fierer N., Knight R. (2010). Delivery mode shapes the acquisition and structure of the initial microbiota across multiple body habitats in newborns. Proc. Natl. Acad. Sci. USA. 107, 11971–11975;
- Satokari R., Grönroos T., Laitinen K., Salminen S., Isolauri E. (2009). Bifidobacterium and Lactobacillus DNA in the human placenta. Lett. Appl. Microbiol. 48, 8–12;
- Aagaard K., Ma J., Antony K.M., Ganu R., Petrosino J., Versalovic J. (2014). The placenta harbors a unique microbiome. Sci. Transl. Med. 6, 237ra265;
- Oh K.J., Lee S.E., Jung H., Kim G., Romero R., Yoon B.H. (2010). Detection of ureaplasmas by the polymerase chain reaction in the amniotic fluid of patients with cervical insufficiency. J. Perinat. Med. 38, 261–268;
- DiGiulio D.B., Romero R., Amogan H.P., Kusanovic J.P., Bik E.M., Gotsch F. et al. (2008). Microbial prevalence, diversity and abundance in amniotic fluid during preterm labor: a molecular and culture-based investigation. PLoS One. 3, e3056;
- Jiménez E., Fernández L., Marín M.L., Martín R., Odriozola J.M., Nueno-Palop C. et al. (2005). Isolation of commensal bacteria from umbilical cord blood of healthy neonates born by cesarean section. Curr. Microbiol. 51, 270–274;
- Jimenez E., Marin M.L., Martin R., Odriozola J.M., Olivares M., Xaus J. et al. (2008). Is meconium from healthy newborns actually sterile? Res. Microbiol. 159, 187–193;
- Hu J., Nomura Y., Bashir A., Fernandez-Hernandez H., Itzkowitz S., Pei Z. et al. (2013). Diversified microbiota of meconium is affected by maternal diabetes status. PLoS One. 8, e78257;
- Mackie R.I., Sghir A., Gaskins H.R. (1999). Developmental microbial ecology of the neonatal gastrointestinal tract. Am. J. Clin. Nutr. 69, 1035S—1045S;
- Ravel J., Gajer P., Abdo Z., Schneider G.M., Koenig S.S., McCulle S.L. et al. (2011). Vaginal microbiome of reproductive-age women. Proc. Natl. Acad. Sci. USA. 108 Suppl 1, 4680–4687;
- Cohen J. (2016). Vaginal bacteria species can raise HIV infection risk and undermine prevention. Science;
- Witkin S.S. and Ledger W.J. (2012). Complexities of the uniquely human vagina. Sci. Transl. Med. 4, 132fs11;
- Backhed F., Roswall J., Peng Y., Feng Q., Jia H., Kovatcheva-Datchary P. et al. (2015). Dynamics and stabilization of the human gut microbiome during the first year of life. Cell Host Microbe. 17, 690–703;
- MacIntyre D.A., Chandiramani M., Lee Y.S., Kindinger L., Smith A., Angelopoulos N. et al. (2015). The vaginal microbiome during pregnancy and the postpartum period in a European population. Sci. Rep. 5, 8988;
- Insoft R.M., Sanderson I.S., Walker W.A. (1996). Development of immune function within the human intestine and its role in neonatal diseases. Pediatr. Clin. North Am. 43, 551–571;
- Jakobsson H.E., Abrahamsson T.R., Jenmalm M.C., Harris K., Quince C., Jernberg C. et al. (2014). Decreased gut microbiota diversity, delayed Bacteroidetes colonisation and reduced Th1 responses in infants delivered by caesarean section. Gut. 63, 559–566;
- Butte N., Lopez-Alarcon M., Garza C. Nutrient adequacy of exclusive breastfeeding for the term infant during the first six months of life. Geneva: World Health Organization, 2002. — 47 p.;
- WHO collaborative study team on the role of breastfeeding on the prevention of infant mortality. (2000). Effect of breastfeeding on infant and child mortality due to infectious diseases in less developed countries: a pooled analysis. Lancet. 355, 451–455;
- Horta B.L. and Victora C.G. Long-term effects of breastfeeding: a systematic review. Geneva: World Health Organization, 2013. — 69 p.;
- Horta B.L., de Mola C.L., Victora C.G. (2015). Long-term consequences of breastfeeding on cholesterol, obesity, systolic blood pressure, and type-2 diabetes: systematic review and meta-analysis. Acta Paediatr. 104, 30–37;
- Dorr H. and Sittel I. (1953). Bacteriological examination of human milk and its relation to mastitis. Zentralbl. Gynakol. 75, 1833–1835;
- Martín R., Langa S., Reviriego C., Jimínez E., Marín M.L., Xaus J. et al. (2003). Human milk is a source of lactic acid bacteria for the infant gut. J. Pediatr. 143, 754–758;
- Azad M.B., Konya T., Maughan H., Guttman D.S., Field C.J., Chari R.S. et al. (2013). Gut microbiota of healthy Canadian infants: profi les by mode of delivery and infant diet at 4 months. CMAJ. 185, 385–394;
- Yatsunenko T., Rey F.E., Manary M.J., Trehan I., Dominguez-Bello M.G., Contreras M. et al. (2012). Human gut microbiome viewed across age and geography. Nature. 486, 222–227;
- Gura T. (2014). Nature’s fi rst functional food. Science. 345, 747–749;
- De Leoz M.L., Kalanetra K.M., Bokulich N.A., Strum J.S., Underwood M.A., German J.B. et al. (2015). Human milk glycomics and gut microbial genomics in infant feces show a correlation between human milk oligosaccharides and gut microbiota: a proof-of-concept study. J. Proteome Res. 14, 491–502;
- Bourges D., Meurens F., Berri M., Chevaleyre C., Zanello G., Levast B. et al. (2008). New insights into the dual recruitment of IgA+ B cells in the developing mammary gland. Mol. Immunol. 45, 3354–3362;
- Latuga M.S., Stuebe A., Seed P.C. (2014). A review of the source and function of microbiota in breast milk. Semin. Reprod. Med. 32, 68–73;
- Hunt K.M., Foster J.A., Forney L.J., Schutte U.M., Beck D.L., Abdo Z. et al. (2011). Characterization of the diversity and temporal stability of bacterial communities in human milk. PLoS One. 6, e21313;
- Lif Holgerson P., Harnevik L., Hernell O., Tanner A.C., Johansson I. (2011). Mode of birth delivery affects oral microbiota in infants. J. Dent. Res. 90, 1183–1188;
- Cabrera-Rubio R., Collado M.C., Laitinen K., Salminen S., Isolauri E., Mira A. (2012). The human milk microbiome changes over lactation and is shaped by maternal weight and mode of delivery. Am. J. Clin. Nutr. 96, 544–551;
- Valles Y., Artacho A., Pascual-Garcia A., Ferrus M.L., Gosalbes M.J., Abellan J.J., Francino M.P. (2014). Microbial succession in the gut: directional trends of taxonomic and functional change in a birth cohort of Spanish infants. PLoS Genet. 10, e1004406;
- Artis D. (2008). Epithelial-cell recognition of commensal bacteria and maintenance of immune homeostasis in the gut. Nat. Rev. Immunol. 8, 411–420;
- McAuley J.L., Linden S.K., Png C.W., King R.M., Pennington H.L., Gendler S.J. et al. (2007). MUC1 cell surface mucin is a critical element of the mucosal barrier to infection. J. Clin. Invest. 117, 2313–2324;
- Shen L. and Turner J.R. (2006). Role of epithelial cells in initiation and propagation of intestinal inflammation. Eliminating the static: tight junction dynamics exposed. Am. J. Physiol. Gastrointest. Liver. Physiol. 290, G577–G582;
- Hooper L.V., Wong M.H., Thelin A., Hansson L., Falk P.G., Gordon J.I. (2001). Molecular analysis of commensal host-microbial relationships in the intestine. Science. 291, 881–884;
- Vijay-Kumar M., Aitken J.D., Carvalho F.A. Cullender T.C., Mwangi S., Srinivasan S. et al. (2010). Metabolic syndrome and altered gut microbiota in mice lacking toll-like receptor 5. Science. 328, 228–231;
- Round J.L. and Mazmanian S.K. (2009). The gut microbiota shapes intestinal responses during health and disease. Nat. Rev. Immunol. 9, 313–323;
- O’Hara A.M. and Shanahan F. (2007). Gut microbiota: mining for therapeutic potential. Clin. Gastroenterol. Hepatol. 5, 274–284;
- Abreu M.T., Fukata M., Arditi M. (2005). TLR signaling in the gut in health and disease. J. Immunol. 174, 4453–4460;
- Claud E.C., Lu L., Anton P.M., Savidge T., Walker W.A., Cherayil B.J. (2004). Developmentally-regulated IκB expression in intestinal epithelium and susceptibility to flagellin-induced inflammation. Proc. Natl. Acad. Sci. USA. 101, 7404–7408;
- Mazmanian S.K. and Kasper D.L. (2006). The love—hate relationship between bacterial polysaccharides and the host immune system. Nat. Rev. Immunol. 6, 849–858;
- Atarashi K., Tanoue T., Shima T., Imaoka A., Kuwahara T., Momose Y. et al. (2011). Induction of colonic regulatory T cells by indigenous Clostridium species. Science. 331, 337–341;
- Зоопарк в моем животе;
- Kozyrskyj A.L., Ernst P., Becker A.B. (2007). Increased risk of childhood asthma from antibiotic use in early life. Chest. 131, 1753–1759;
- Risnes K.R., Belanger K., Murk W., Bracken M.B. (2011). Antibiotic exposure by 6 months, and asthma and allergy at 6 years: findings in a cohort of 1,401 US children. Am. J. Epidemiol. 173, 310–318;
- Hoskin-Parr L., Teyhan A., Blocker A., Henderson A.J. (2013). Antibiotic exposure in the first 2 years of life and development of asthma and other allergic diseases by 7.5 years: a dose-dependent relationship. Pediatr. Allergy Immunol. 24, 762–771;
- Bailey L.C., Forrest C.B., Zhang P., Richards T.M., Livshits A., DeRusso P.A. (2014). Association of antibiotics in infancy with early childhood obesity. JAMA Pediatr. 168, 1063–1069;
- Metsälä J., Lundqvist A., Virta L.J., Kaila M., Gissler M., Virtanen S.M. (2013). Mother’s and offspring’s use of antibiotics, and infant allergy to cow’s milk. Epidemiology. 24, 303–309;
- Kronman M.P., Zaoutis T.E., Haynes K., Feng R., Coffin S.E. (2012). Antibiotic exposure and IBD development among children: a population-based cohort study. Pediatrics. 130, e794–e803;
- Braegger C., Chmielewska A., Decsi T., Kolacek S., Mihatsch W., Moreno L. et al. (2011). Supplementation of infant formula with probiotics and/or prebiotics: a systematic review and comment by the ESPGHAN committee on nutrition. J. Pediatr. Gastroenterol. Nutr. 52, 238–250;
- Panduru M., Panduru N.M., Sălăvăstru C.M., Tiplica G.S. (2015). Probiotics and primary prevention of atopic dermatitis: a meta-analysis of randomized controlled studies. J. Eur. Acad. Dermatol. Venereol. 29, 232–242;
- Foolad N., Brezinski E.A., Chase E.P., Armstrong A.W. (2013). Effect of nutrient supplementation on atopic dermatitis in children: a systematic review of probiotics, prebiotics, formula and fatty acids. JAMA Dermatol. 149, 350–355;
- Doege K., Grajecki D., Zyriax B.C., Detinkina E., Zu Eulenburg C., Buhling K.J. (2012). Impact of maternal supplementation with probiotics during pregnancy on atopic eczema in childhood — a meta-analysis. Br. J. Nutr. 107, 1–6;
- Kim S.O., Ah Y.M., Yu Y.M., Choi K.H., Shin W.G., Lee J.Y. (2014). Effects of probiotics for the treatment of atopic dermatitis: a meta-analysis of randomized controlled trials. Ann. Allergy Asthma Immunol. 113, 217–226;
- Savino F., Cordisco L., Tarasco V., Palumeri E., Calabrese R., Oggero R. et al. (2010). Lactobacillus reuteri DSM 17938 in infantile colic: a randomized, double-blind, placebo-controlled trial. Pediatrics. 126, e526–e533;
- Song S.J., Lauber C., Costello E.K., Lozupone C.A., Humphrey G., Berg-Lyons D. et al. (2013). Cohabiting family members share microbiota with one another and with their dogs. Elife. 2, e00458;
- Sjögren Y.M., Jenmalm M.C., Böttcher M.F., Björkstén B., Sverremark-Ekström E. (2009). Altered early infant gut microbiota in children developing allergy up to 5 years of age. Clin. Exp. Allergy. 39, 518–526;
- Ownby D.R., Johnson C.C., Peterson E.L. (2002). Exposure to dogs and cats in the first year of life and risk of allergic sensitization at 6 to 7 years of age. J. Am. Med. Assoc. 288, 963–972;
- Normand A.C., Sudre B., Vacheyrou M., Depner M., Wouters I.M., Noss I. et al. (2011). Airborne cultivable microflora and microbial transfer in farm buildings and rural dwellings. Occup. Environ. Med. 68, 849–855;
- Ткаченко Е.И., Иванов С.В., Жигалова Т.Н., Ситкин С.И. (2008). Энтеральное питание при язвенном колите. Лечащий Врач. 6;
- Dominguez-Bello M.G., De Jesus-Laboy K.M., Shen N., Cox L.M., Amir A., Gonzalez A. et al. (2016). Partial restoration of the microbiota of cesarean-born infants via vaginal microbial transfer. Nat. Med. 22, 250–253;
- Nuriel-Ohayon M., Neuman H., Koren O. (2016). Microbial changes during pregnancy, birth, and infancy. Front. Microbiol. 7, 1031;
- Newburg D.S. and Morelli L. (2015). Human milk and infant intestinal mucosal glycans guide succession of the neonatal intestinal microbiota. Pediatr. Res. 77, 115–120;
- Tamburini S., Shen N., Wu H.C., Clemente J.C. (2016). The microbiome in early life: implications for health outcomes. Nat. Med. 22, 713–722.
Graphic depicting the human skin microbiota, with relative prevalences of various classes of bacteria
The human microbiome is the aggregate of all microbiota that reside on or within human tissues and biofluids along with the corresponding anatomical sites in which they reside,[1] including the skin, mammary glands, seminal fluid, uterus, ovarian follicles, lung, saliva, oral mucosa, conjunctiva, biliary tract, and gastrointestinal tract. Types of human microbiota include bacteria, archaea, fungi, protists, and viruses. Though micro-animals can also live on the human body, they are typically excluded from this definition. In the context of genomics, the term human microbiome is sometimes used to refer to the collective genomes of resident microorganisms;[2] however, the term human metagenome has the same meaning.[1]
Humans are colonized by many microorganisms, with approximately the same order of magnitude of non-human cells as human cells.[3] Some microorganisms that colonize humans are commensal, meaning they co-exist without harming humans; others have a mutualistic relationship with their human hosts.[2]: 700 [4] Conversely, some non-pathogenic microorganisms can harm human hosts via the metabolites they produce, like trimethylamine, which the human body converts to trimethylamine N-oxide via FMO3-mediated oxidation.[5][6] Certain microorganisms perform tasks that are known to be useful to the human host, but the role of most of them is not well understood. Those that are expected to be present, and that under normal circumstances do not cause disease, are sometimes deemed normal flora or normal microbiota.[2]
The Human Microbiome Project (HMP) took on the project of sequencing the genome of the human microbiota, focusing particularly on the microbiota that normally inhabit the skin, mouth, nose, digestive tract, and vagina.[2] It reached a milestone in 2012 when it published its initial results.[7]
Terminology[edit]
Though widely known as flora or microflora, this is a misnomer in technical terms, since the word root flora pertains to plants, and biota refers to the total collection of organisms in a particular ecosystem. Recently, the more appropriate term microbiota is applied, though its use has not eclipsed the entrenched use and recognition of flora with regard to bacteria and other microorganisms. Both terms are being used in different literature.[4]
Relative numbers[edit]
As of 2014, it was reported in popular media and in the scientific literature that there are about 10 times as many microbial cells in the human body as there are human cells; this figure was based on estimates that the human microbiome includes around 100 trillion bacterial cells and that an adult human typically has around 10 trillion human cells.[8] In 2014, the American Academy of Microbiology published a FAQ that emphasized that the number of microbial cells and the number of human cells are both estimates, and noted that recent research had arrived at a new estimate of the number of human cells – approximately 37.2 trillion, meaning that the ratio of microbial-to-human cells, if the original estimate of 100 trillion bacterial cells is correct, is closer to 3:1.[8][9] In 2016, another group published a new estimate of the ratio being roughly 1:1 (1.3:1, with «an uncertainty of 25% and a variation of 53% over the population of standard 70-kg [150 lb] males»).[10][3]
A more recent estimate is a ratio of 1.3:1 bacterial cells for every human cell, whereas the number of phages and viruses outnumber bacterial cells by at least an order of magnitude more. The number of bacterial genes (assuming 1000 bacterial species in the gut with 2000 genes per species) is estimated to be 2,000,000 genes, 100 times the number of approximately 20,000 human genes.[11]
Study[edit]
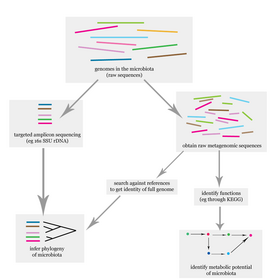
Flowchart illustrating how the human microbiome is studied on the DNA level.
The problem of elucidating the human microbiome is essentially identifying the members of a microbial community, which includes bacteria, eukaryotes, and viruses.[12] This is done primarily using deoxyribonucleic acid (DNA)-based studies, though ribonucleic acid (RNA), protein and metabolite based studies are also performed.[12][13] DNA-based microbiome studies typically can be categorized as either targeted amplicon studies or, more recently, shotgun metagenomic studies. The former focuses on specific known marker genes and is primarily informative taxonomically, while the latter is an entire metagenomic approach which can also be used to study the functional potential of the community.[12] One of the challenges that is present in human microbiome studies, but not in other metagenomic studies, is to avoid including the host DNA in the study.[14]
Aside from simply elucidating the composition of the human microbiome, one of the major questions involving the human microbiome is whether there is a «core», that is, whether there is a subset of the community that is shared among most humans.[15][16] If there is a core, then it would be possible to associate certain community compositions with disease states, which is one of the goals of the HMP. It is known that the human microbiome (such as the gut microbiota) is highly variable both within a single subject and among different individuals, a phenomenon which is also observed in mice.[4]
On 13 June 2012, a major milestone of the HMP was announced by the National Institutes of Health (NIH) director Francis Collins.[7] The announcement was accompanied with a series of coordinated articles published in Nature[17][18] and several journals in the Public Library of Science (PLoS) on the same day. By mapping the normal microbial make-up of healthy humans using genome sequencing techniques, the researchers of the HMP have created a reference database and the boundaries of normal microbial variation in humans. From 242 healthy U.S. volunteers, more than 5,000 samples were collected from tissues from 15 (men) to 18 (women) body sites such as mouth, nose, skin, lower intestine (stool), and vagina. All the DNA, human and microbial, were analyzed with DNA sequencing machines. The microbial genome data were extracted by identifying the bacterial specific ribosomal RNA, 16S rRNA. The researchers calculated that more than 10,000 microbial species occupy the human ecosystem, and they have identified 81–99% of the genera.[citation needed]
Analysis after the processing[edit]
The statistical analysis is essential to validate the obtained results (ANOVA can be used to size the differences between the groups); if it is paired with graphical tools, the outcome is easily visualized and understood.[19]
Once a metagenome is assembled, it is possible to infer the functional potential of the microbiome. The computational challenges for this type of analysis are greater than for single genomes, because usually metagenomes assemblers have poorer quality, and many recovered genes are non-complete or fragmented. After the gene identification step, the data can be used to carry out a functional annotation by means of multiple alignment of the target genes against orthologs databases.[20]
Marker gene analysis[edit]
It is a technique that exploits primers to target a specific genetic region and enables to determine the microbial phylogenies. The genetic region is characterized by a highly variable region which can confer detailed identification; it is delimited by conserved regions, which function as binding sites for primers used in PCR. The main gene used to characterize bacteria and archaea is 16S rRNA gene, while fungi identification is based on Internal Transcribed Spacer (ITS). The technique is fast and not so expensive and enables to obtain a low-resolution classification of a microbial sample; it is optimal for samples that may be contaminated by host DNA. Primer affinity varies among all DNA sequences, which may result in biases during the amplification reaction; indeed, low-abundance samples are susceptible to overamplification errors, since the other contaminating microorganisms result to be over-represented in case of increasing the PCR cycles. Therefore, the optimization of primer selection can help to decrease such errors, although it requires complete knowledge of the microorganisms present in the sample, and their relative abundances.[21]
Marker gene analysis can be influenced by the primer choice; in this kind of analysis it’s desirable to use a well-validated protocol (such as the one used in the Earth Microbiome Project). The first thing to do in a marker gene amplicon analysis is to remove sequencing errors; a lot of sequencing platforms are very reliable, but most of the apparent sequence diversity is still due to errors during the sequencing process. To reduce this phenomenon a first approach is to cluster sequences into Operational taxonomic unit (OTUs): this process consolidates similar sequences (a 97% similarity threshold is usually adopted) into a single feature that can be used in further analysis steps; this method however would discard SNPs because they would get clustered into a single OTU. Another approach is Oligotyping, which includes position-specific information from 16s rRNA sequencing to detect small nucleotide variations and from discriminating between closely related distinct taxa. These methods give as an output a table of DNA sequences and counts of the different sequences per sample rather than OTU.[21]
Another important step in the analysis is to assign a taxonomic name to microbial sequences in the data. This can be done using machine learning approaches that can reach an accuracy at genus-level of about 80%. Other popular analysis packages provide support for taxonomic classification using exact matches to reference databases and should provide greater specificity, but poor sensitivity. Unclassified microorganism should be further checked for organelle sequences.[21]
Phylogenetic analysis[edit]
Many methods that exploit phylogenetic inference use the 16SRNA gene for Archea and Bacteria and the 18SRNA gene for Eukaryotes. Phylogenetic comparative methods (PCS) are based on the comparison of multiple traits among microorganisms; the principle is: the closely they are related, the higher number of traits they share. Usually PCS are coupled with phylogenetic generalized least square (PGLS) or other statistical analysis to get more significant results. Ancestral state reconstruction is used in microbiome studies to impute trait values for taxa whose traits are unknown. This is commonly performed with PICRUSt, which relies on available databases. Phylogenetic variables are chosen by researchers according to the type of study: through the selection of some variables with significant biological informations, it is possible to reduce the dimension of the data to analyse.[22]
Phylogenetic aware distance is usually performed with UniFrac or similar tools, such as Soresen’s index or Rao’s D, to quantify the differences between the different communities. All this methods are negatively affected by horizontal gene transmission (HGT), since it can generate errors and lead to the correlation of distant species. There are different ways to reduce the negative impact of HGT: the use of multiple genes or computational tools to assess the probability of putative HGT events.[22]
Ecological Network analysis[edit]
Microbial communities develop in a very complex dynamics which can be viewed and analyzed as an ecosystem. The ecological interactions between microbes govern its change, equilibrium and stability, and can be represented by a population dynamic model.[23]
The ongoing study of ecological features of the microbiome is growing rapidly and allows to understand the fundamental properties of the microbiome. Understanding the underlying rules of microbial community could help with treating diseases related to unstable microbial communities].
A very basic question is if different human, which share different microbial communities, have the same underlying microbial dynamics.[24] Increasing evidence and indications have found that the dynamics is indeed universal.[25] This question is a basic step that will allow scientists to develop treatment strategies, based on the complex dynamics of human microbial communities.
There are more important properties on which considerations should be taken into account for developing interventions strategies for controlling the human microbial dynamics.[26] Controlling the microbial communities could result in solving very bad and harmful diseases.
Types[edit]
Bacteria[edit]
Commensals vs pathogens mechanism. Mechanisms underlying the inflammation in COPD. Airway epithelium has complex structure: consists of at least seven diverse cell types interacting with each other by means of tight junctions. Moreover, epithelial calls can deliver the signals into the underlying tissues taking part in the mechanisms of innate and adaptive immune defence. The key transmitters of the signals are dendritic cells. Once pathogenic bacterium (e.g., S. pneumoniae, P. aeruginosa) has activated particular pattern recognition receptors on/in epithelial cells, the proinflammatory signaling pathways are activated. This results mainly in IL-1, IL-6 and IL-8 production. These cytokines induce the chemotaxis to the site of infection in its target cells (e.g., neutrophils, dendritic cells and macrophages). On the other hand, representatives of standard microbiota cause only weak signaling preventing the inflammation. The mechanism of distinguishing between harmless and harmful bacteria on the molecular as well as on physiological levels is not completely understood.
Populations of microbes (such as bacteria and yeasts) inhabit the skin and mucosal surfaces in various parts of the body. Their role forms part of normal, healthy human physiology, however if microbe numbers grow beyond their typical ranges (often due to a compromised immune system) or if microbes populate (such as through poor hygiene or injury) areas of the body normally not colonized or sterile (such as the blood, or the lower respiratory tract, or the abdominal cavity), disease can result (causing, respectively, bacteremia/sepsis, pneumonia, and peritonitis).[medical citation needed]
The Human Microbiome Project found that individuals host thousands of bacterial types, different body sites having their own distinctive communities. Skin and vaginal sites showed smaller diversity than the mouth and gut, these showing the greatest richness. The bacterial makeup for a given site on a body varies from person to person, not only in type, but also in abundance. Bacteria of the same species found throughout the mouth are of multiple subtypes, preferring to inhabit distinctly different locations in the mouth. Even the enterotypes in the human gut, previously thought to be well understood, are from a broad spectrum of communities with blurred taxon boundaries.[27][28]
It is estimated that 500 to 1,000 species of bacteria live in the human gut but belong to just a few phyla: Bacillota and Bacteroidota dominate but there are also Pseudomonadota, Verrucomicrobiota, Actinobacteriota, Fusobacteriota, and «Cyanobacteria».[29]
A number of types of bacteria, such as Actinomyces viscosus and A. naeslundii, live in the mouth, where they are part of a sticky substance called plaque. If this is not removed by brushing, it hardens into calculus (also called tartar). The same bacteria also secrete acids that dissolve tooth enamel, causing tooth decay.[citation needed]
The vaginal microflora consist mostly of various lactobacillus species. It was long thought that the most common of these species was Lactobacillus acidophilus, but it has later been shown that L. iners is in fact most common, followed by L. crispatus. Other lactobacilli found in the vagina are L. jensenii, L. delbruekii and L. gasseri. Disturbance of the vaginal flora can lead to infections such as bacterial vaginosis and candiadiasis..[citation needed]
Archaea[edit]
Archaea are present in the human gut, but, in contrast to the enormous variety of bacteria in this organ, the numbers of archaeal species are much more limited.[30] The dominant group are the methanogens, particularly Methanobrevibacter smithii and Methanosphaera stadtmanae.[31] However, colonization by methanogens is variable, and only about 50% of humans have easily detectable populations of these organisms.[32]
As of 2007, no clear examples of archaeal pathogens were known,[33][34] although a relationship has been proposed between the presence of some methanogens and human periodontal disease.[35]
Fungi[edit]
Fungi, in particular yeasts, are present in the human gut.[36][37][38][39] The best-studied of these are Candida species due to their ability to become pathogenic in immunocompromised and even in healthy hosts.[37][38][39] Yeasts are also present on the skin,[36] such as Malassezia species, where they consume oils secreted from the sebaceous glands.[40][41]
Viruses[edit]
Viruses, especially bacterial viruses (bacteriophages), colonize various body sites. These colonized sites include the skin,[42] gut,[43] lungs,[44] and oral cavity.[45] Virus communities have been associated with some diseases, and do not simply reflect the bacterial communities.[46][47][48]
Anatomical areas[edit]
Skin[edit]
A study of 20 skin sites on each of ten healthy humans found 205 identified genera in 19 bacterial phyla, with most sequences assigned to four phyla: Actinomycetota (51.8%), Bacillota (24.4%), Pseudomonadota (16.5%), and Bacteroidota (6.3%).[49] A large number of fungal genera are present on healthy human skin, with some variability by region of the body; however, during pathological conditions, certain genera tend to dominate in the affected region.[36] For example, Malassezia is dominant in atopic dermatitis and Acremonium is dominant on dandruff-affected scalps.[36]
The skin acts as a barrier to deter the invasion of pathogenic microbes. The human skin contains microbes that reside either in or on the skin and can be residential or transient. Resident microorganism types vary in relation to skin type on the human body. A majority of microbes reside on superficial cells on the skin or prefer to associate with glands. These glands such as oil or sweat glands provide the microbes with water, amino acids, and fatty acids. In addition, resident bacteria that associated with oil glands are often Gram-positive and can be pathogenic.[2]
Conjunctiva[edit]
A small number of bacteria and fungi are normally present in the conjunctiva.[36][50] Classes of bacteria include Gram-positive cocci (e.g., Staphylococcus and Streptococcus) and Gram-negative rods and cocci (e.g., Haemophilus and Neisseria) are present.[50] Fungal genera include Candida, Aspergillus, and Penicillium.[36] The lachrymal glands continuously secrete, keeping the conjunctiva moist, while intermittent blinking lubricates the conjunctiva and washes away foreign material. Tears contain bactericides such as lysozyme, so that microorganisms have difficulty in surviving the lysozyme and settling on the epithelial surfaces.
Gastrointestinal tract[edit]
In humans, the composition of the gastrointestinal microbiome is established during birth.[55] Birth by Cesarean section or vaginal delivery also influences the gut’s microbial composition. Babies born through the vaginal canal have non-pathogenic, beneficial gut microbiota similar to those found in the mother.[56] However, the gut microbiota of babies delivered by C-section harbors more pathogenic bacteria such as Escherichia coli and Staphylococcus and it takes longer to develop non-pathogenic, beneficial gut microbiota.[57]
The relationship between some gut microbiota and humans is not merely commensal (a non-harmful coexistence), but rather a mutualistic relationship.[2] Some human gut microorganisms benefit the host by fermenting dietary fiber into short-chain fatty acids (SCFAs), such as acetic acid and butyric acid, which are then absorbed by the host.[4][58] Intestinal bacteria also play a role in synthesizing vitamin B and vitamin K as well as metabolizing bile acids, sterols, and xenobiotics.[2][58] The systemic importance of the SCFAs and other compounds they produce are like hormones and the gut flora itself appears to function like an endocrine organ,[58] and dysregulation of the gut flora has been correlated with a host of inflammatory and autoimmune conditions.[4][59]
The composition of human gut microbiota changes over time, when the diet changes, and as overall health changes.[4][59] A systematic review of 15 human randomized controlled trials from July 2016 found that certain commercially available strains of probiotic bacteria from the Bifidobacterium and Lactobacillus genera (B. longum, B. breve, B. infantis, L. helveticus, L. rhamnosus, L. plantarum, and L. casei), when taken by mouth in daily doses of 109–1010 colony forming units (CFU) for 1–2 months, possess treatment efficacy (i.e., improves behavioral outcomes) in certain central nervous system disorders – including anxiety, depression, autism spectrum disorder, and obsessive–compulsive disorder – and improves certain aspects of memory.[60]
Urethra and bladder[edit]
The genitourinary system appears to have a microbiota,[61][62] which is an unexpected finding in light of the long-standing use of standard clinical microbiological culture methods to detect bacteria in urine when people show signs of a urinary tract infection; it is common for these tests to show no bacteria present.[63] It appears that common culture methods do not detect many kinds of bacteria and other microorganisms that are normally present.[63] As of 2017, sequencing methods were used to identify these microorganisms to determine if there are differences in microbiota between people with urinary tract problems and those who are healthy.[61][62][64] To properly assess the microbiome of the bladder as opposed to the genitourinary system, the urine specimen should be collected directly from the bladder, which is often done with a catheter.[65]
Vagina[edit]
Vaginal microbiota refers to those species and genera that colonize the vagina. These organisms play an important role in protecting against infections and maintaining vaginal health.[66] The most abundant vaginal microorganisms found in premenopausal women are from the genus Lactobacillus, which suppress pathogens by producing hydrogen peroxide and lactic acid.[38][66][67] Bacterial species composition and ratios vary depending on the stage of the menstrual cycle.[68][69][needs update] Ethnicity also influences vaginal flora. The occurrence of hydrogen peroxide-producing lactobacilli is lower in African American women and vaginal pH is higher.[70] Other influential factors such as sexual intercourse and antibiotics have been linked to the loss of lactobacilli.[67] Moreover, studies have found that sexual intercourse with a condom does appear to change lactobacilli levels, and does increase the level of Escherichia coli within the vaginal flora.[67] Changes in the normal, healthy vaginal microbiota is an indication of infections,[71] such as candidiasis or bacterial vaginosis.[67] Candida albicans inhibits the growth of Lactobacillus species, while Lactobacillus species which produce hydrogen peroxide inhibit the growth and virulence of Candida albicans in both the vagina and the gut.[36][38][39]
Fungal genera that have been detected in the vagina include Candida, Pichia, Eurotium, Alternaria, Rhodotorula, and Cladosporium, among others.[36]
Placenta[edit]
Until recently the placenta was considered to be a sterile organ but commensal, nonpathogenic bacterial species and genera have been identified that reside in the placental tissue.[72][73][74] However, the existence of a microbiome in the placenta is controversial as criticized in several researches. So called «placental microbiome» is likely derived from contamination of regents because low-biomass samples are easily contaminated.[75][76][77]
Uterus[edit]
Until recently, the upper reproductive tract of women was considered to be a sterile environment. A variety of microorganisms inhabit the uterus of healthy, asymptomatic women of reproductive age. The microbiome of the uterus differs significantly from that of the vagina and gastrointestinal tract.[78]
Oral cavity[edit]
The environment present in the human mouth allows the growth of characteristic microorganisms found there. It provides a source of water and nutrients, as well as a moderate temperature.[2] Resident microbes of the mouth adhere to the teeth and gums to resist mechanical flushing from the mouth to stomach where acid-sensitive microbes are destroyed by hydrochloric acid.[2][38]
Anaerobic bacteria in the oral cavity include: Actinomyces, Arachnia, Bacteroides, Bifidobacterium, Eubacterium, Fusobacterium, Lactobacillus, Leptotrichia, Peptococcus, Peptostreptococcus, Propionibacterium, Selenomonas, Treponema, and Veillonella.[79][needs update] Genera of fungi that are frequently found in the mouth include Candida, Cladosporium, Aspergillus, Fusarium, Glomus, Alternaria, Penicillium, and Cryptococcus, among others.[36]
Bacteria accumulate on both the hard and soft oral tissues in biofilm allowing them to adhere and strive in the oral environment while protected from the environmental factors and antimicrobial agents.[80] Saliva plays a key biofilm homeostatic role allowing recolonization of bacteria for formation and controlling growth by detaching biofilm buildup.[81] It also provides a means of nutrients and temperature regulation. The location of the biofilm determines the type of exposed nutrients it receives.[82]
Oral bacteria have evolved mechanisms to sense their environment and evade or modify the host. However, a highly efficient innate host defense system constantly monitors the bacterial colonization and prevents bacterial invasion of local tissues. A dynamic equilibrium exists between dental plaque bacteria and the innate host defense system.[83]
This dynamic between host oral cavity and oral microbes plays a key role in health and disease as it provides entry into the body.[84]
A healthy equilibrium presents a symbiotic relationship where oral microbes limit growth and adherence of pathogens while the host provides an environment for them to flourish.[84][80] Ecological changes such as change of immune status, shift of resident microbes and nutrient availability shift from a mutual to parasitic relationship resulting in the host being prone to oral and systemic disease.[80] Systemic diseases such as diabetes and cardiovascular diseases has been correlated to poor oral health.[84] Of particular interest is the role of oral microorganisms in the two major dental diseases: dental caries and periodontal disease.[83] Pathogen colonization at the periodontium cause an excessive immune response resulting in a periodontal pocket- a deepened space between the tooth and gingiva.[80] This acts as a protected blood-rich reservoir with nutrients for anaerobic pathogens.[80] Systemic disease at various sites of the body can result from oral microbes entering the blood bypassing periodontal pockets and oral membranes.[84]
Persistent proper oral hygiene is the primary method for preventing oral and systemic disease.[84] It reduces the density of biofilm and overgrowth of potential pathogenic bacteria resulting in disease.[82] However, proper oral hygiene may not be enough as the oral microbiome, genetics, and changes to immune response play a factor in developing chronic infections.[82] Use of antibiotics could treat already spreading infection but ineffective against bacteria within biofilms.[82]
Nasal cavity[edit]
The healthy nasal microbiome is dominated by Corynebacterium, and Staphylococcus. The mucosal microbiome plays a critical role in modulating viral infection.[85]
Lung[edit]
Much like the oral cavity, the upper and lower respiratory system possess mechanical deterrents to remove microbes. Goblet cells produce mucous which traps microbes and moves them out of the respiratory system via continuously moving ciliated epithelial cells.[2] In addition, a bactericidal effect is generated by nasal mucus which contains the enzyme lysozyme.[2] The upper and lower respiratory tract appears to have its own set of microbiota.[86] Pulmonary bacterial microbiota belong to 9 major bacterial genera: Prevotella, Sphingomonas, Pseudomonas, Acinetobacter, Fusobacterium, Megasphaera, Veillonella, Staphylococcus, and Streptococcus. Some of the bacteria considered «normal biota» in the respiratory tract can cause serious disease especially in immunocompromised individuals; these include Streptococcus pyogenes, Haemophilus influenzae, Streptococcus pneumoniae, Neisseria meningitidis, and Staphylococcus aureus.[citation needed] Fungal genera that compose the pulmonary mycobiome include Candida, Malassezia, Neosartorya, Saccharomyces, and Aspergillus, among others.[36]
Unusual distributions of bacterial and fungal genera in the respiratory tract is observed in people with cystic fibrosis.[36][87] Their bacterial flora often contains antibiotic-resistant and slow-growing bacteria, and the frequency of these pathogens changes in relation to age.[87]
Biliary tract[edit]
Traditionally the biliary tract has been considered to be normally sterile, and the presence of microorganisms in bile is a marker of pathological process. This assumption was confirmed by failure in allocation of bacterial strains from the normal bile duct. Papers began emerging in 2013 showing that the normal biliary microbiota is a separate functional layer which protects a biliary tract from colonization by exogenous microorganisms.[88]
Disease and death[edit]
Human bodies rely on the innumerable bacterial genes as the source of essential nutrients.[89] Both metagenomic and epidemiological studies indicate vital roles for the human microbiome in preventing a wide range of diseases, from type 2 diabetes and obesity to inflammatory bowel disease, Parkinson’s disease, and even mental health conditions like depression.[90] A symbiotic relationship between the gut microbiota and different bacteria may influence an individual’s immune response.[91] Metabolites generated by gut microbes appear to be causative factors in type 2 diabetes.[92] Although in its infancy, microbiome-based treatment is also showing promise, most notably for treating drug-resistant C. difficile[dead link] infection[93] and in diabetes treatment.[94]
Clostridioides difficile infection[edit]
An overwhelming presence of the bacteria, C. difficile, leads to an infection of the gastrointestinal tract, normally associated to dysbiosis with the microbiota believed to have been caused by the administration of antibiotics. Use of antibiotics eradicates the beneficial gut flora within the gastrointestinal tract, which normally prevents pathogenic bacteria from establishing dominance.[95] Traditional treatment for C. difficile infections includes an additional regime of antibiotics, however, efficacy rates average between 20 and 30%.[96] Recognizing the importance of healthy gut bacteria, researchers turned to a procedure known as fecal microbiota transplant, where patients experiencing gastrointestinal diseases, such as C. difficile infection, receive fecal content from a healthy individual in hopes of restoring a normal functioning intestinal microbiota.[97] Fecal microbiota transplant is approximately 85–90% effective in people with CDI for whom antibiotics have not worked or in whom the disease recurs following antibiotics.[98][99] Most people with CDI recover with one FMT treatment.[100][95][101]
Cancer[edit]
Although cancer is generally a disease of host genetics and environmental factors, microorganisms are implicated in some 20% of human cancers.[102] Particularly for potential factors in colon cancer, bacterial density is one million times higher than in the small intestine, and approximately 12-fold more cancers occur in the colon compared to the small intestine, possibly establishing a pathogenic role for microbiota in colon and rectal cancers.[103] Microbial density may be used as a prognostic tool in assessment of colorectal cancers.[103]
The microbiota may affect carcinogenesis in three broad ways: (i) altering the balance of tumor cell proliferation and death, (ii) regulating immune system function, and (iii) influencing metabolism of host-produced factors, foods and pharmaceuticals.[102] Tumors arising at boundary surfaces, such as the skin, oropharynx and respiratory, digestive and urogenital tracts, harbor a microbiota. Substantial microbe presence at a tumor site does not establish association or causal links. Instead, microbes may find tumor oxygen tension or nutrient profile supportive. Decreased populations of specific microbes or induced oxidative stress may also increase risks.[102][103] Of the around 1030 microbes on earth, ten are designated by the International Agency for Research on Cancer as human carcinogens.[102] Microbes may secrete proteins or other factors directly drive cell proliferation in the host, or may up- or down-regulate the host immune system including driving acute or chronic inflammation in ways that contribute to carcinogenesis.[102]
Concerning the relationship of immune function and development of inflammation, mucosal surface barriers are subject to environmental risks and must rapidly repair to maintain homeostasis. Compromised host or microbiota resiliency also reduce resistance to malignancy, possibly inducing inflammation and cancer. Once barriers are breached, microbes can elicit proinflammatory or immunosuppressive programs through various pathways.[102] For example, cancer-associated microbes appear to activate NF-κΒ signaling within the tumor microenvironment. Other pattern recognition receptors, such as nucleotide-binding oligomerization domain–like receptor (NLR) family members NOD-2, NLRP3, NLRP6 and NLRP12, may play a role in mediating colorectal cancer.[102] Likewise Helicobacter pylori appears to increase the risk of gastric cancer, due to its driving a chronic inflammatory response in the stomach.[102][103]
Inflammatory bowel disease[edit]
Inflammatory bowel disease consists of two different diseases: ulcerative colitis and Crohn’s disease and both of these diseases present with disruptions in the gut microbiota (also known as dysbiosis). This dysbiosis presents itself in the form of decreased microbial diversity in the gut,[104][105] and is correlated to defects in host genes that changes the innate immune response in individuals.[104]
Human immunodeficiency virus[edit]
The HIV disease progression influences the composition and function of the gut microbiota, with notable differences between HIV-negative, HIV-positive, and post-ART HIV-positive populations.[citation needed] HIV decreases the integrity of the gut epithelial barrier function by affecting tight junctions. This breakdown allows for translocation across the gut epithelium, which is thought to contribute to increases in inflammation seen in people with HIV.[106]
Vaginal microbiota plays a role in the infectivity of HIV, with an increased risk of infection and transmission when the woman has bacterial vaginosis, a condition characterized by an abnormal balance of vaginal bacteria.[107] The enhanced infectivity is seen with the increase in pro-inflammatory cytokines and CCR5 + CD4+ cells in the vagina. However, a decrease in infectivity is seen with increased levels of vaginal Lactobacillus, which promotes an anti-inflammatory condition.[106]
Gut microbiome of centenarians[edit]
Humans who are 100 years old or older, called centenarians, have a distinct gut microbiome. This microbiome is characteristically enriched in microorganisms that are able to synthesize novel secondary bile acids.[108] These secondary bile acids include various isoforms of lithocholic acid that may contribute to healthy aging.[108]
Death[edit]
With death, the microbiome of the living body collapses and a different composition of microorganisms named necrobiome establishes itself as an important active constituent of the complex physical decomposition process. Its predictable changes over time are thought to be useful to help determine the time of death.[109][110]
Environmental health[edit]
Studies in 2009 questioned whether the decline in biota (including microfauna) as a result of human intervention might impede human health, hospital safety procedures, food product design, and treatments of disease.[111]
Changes, modulation and transmission[edit]
Microbiome-based interventions to modulate gut ecology and the immune system[112]
Hygiene, probiotics,[112] prebiotics, microbiota transplants (fecal or skin[113]), medications, antibiotics, diseases, exercise,[114][115] diet, breastfeeding, aging, and other factors[further explanation needed] can change the human microbiome across various anatomical systems or regions such as skin and gut.
Migration[edit]
Preliminary research indicates that immediate changes in the microbiota may occur when a person migrates from one country to another, such as when Thai immigrants settled in the United States[116] or when Latin Americans immigrated into the United States.[117] Losses of microbiota diversity were greater in obese individuals and children of immigrants.[116][117]
Transfer[edit]
A 2023 metagenomic analysis time provided various comprehensive data and insights into microbial sharing between individuals, finding substantial strain sharing among cohabiting individuals, with median strain-sharing rates for the gut and oral microbiomes being 12% (34% for mothers and their 0–3-years-old offspring) and 32% (38% for partners) in the used data. Time since cohabitation was the largest factor and bacterial strain sharing «recapitulated host population structures better[clarification needed] than species-level profiles did».[118][119]
See also[edit]
- Carbon monoxide-releasing molecules
- Drug resistance
- Human Microbiome Project
- Human milk microbiome
- Human virome
- Initial acquisition of microbiota
- List of bacterial vaginosis microbiota
- Microbiome
- Microbiome Immunity Project
- Microorganism
- uBiome
Bibliography[edit]
- Ed Yong. I Contain Multitudes: The Microbes Within Us and a Grander View of Life. 368 pages, Published 9 August 2016 by Ecco, ISBN 0062368591.
References[edit]
- ^ a b Marchesi JR, Ravel J (2015). «The vocabulary of microbiome research: a proposal». Microbiome. 3: 31. doi:10.1186/s40168-015-0094-5. PMC 4520061. PMID 26229597.
Microbiome
This term refers to the entire habitat, including the microorganisms (bacteria, archaea, lower and higher eurkaryotes, and viruses), their genomes (i.e., genes), and the surrounding environmental conditions. This definition is based on that of «biome,» the biotic and abiotic factors of given environments. Others in the field limit the definition of microbiome to the collection of genes and genomes of members of a microbiota. It is argued that this is the definition of metagenome, which combined with the environment constitutes the microbiome. - ^ a b c d e f g h i j k Sherwood L, Willey J, Woolverton C (2013). Prescott’s Microbiology (9th ed.). New York: McGraw Hill. pp. 713–721. ISBN 9780073402406. OCLC 886600661.
- ^ a b Sender R, Fuchs S, Milo R (January 2016). «Are We Really Vastly Outnumbered? Revisiting the Ratio of Bacterial to Host Cells in Humans». Cell. 164 (3): 337–40. doi:10.1016/j.cell.2016.01.013. PMID 26824647.
- ^ a b c d e f Quigley EM (September 2013). «Gut bacteria in health and disease». Gastroenterology & Hepatology. 9 (9): 560–9. PMC 3983973. PMID 24729765.
- ^ Falony G, Vieira-Silva S, Raes J (2015). «Microbiology Meets Big Data: The Case of Gut Microbiota-Derived Trimethylamine». Annual Review of Microbiology. 69: 305–21. doi:10.1146/annurev-micro-091014-104422. PMID 26274026.
we review literature on trimethylamine (TMA), a microbiota-generated metabolite linked to atherosclerosis development.
- ^ Gaci N, Borrel G, Tottey W, O’Toole PW, Brugère JF (November 2014). «Archaea and the human gut: new beginning of an old story». World Journal of Gastroenterology. 20 (43): 16062–78. doi:10.3748/wjg.v20.i43.16062. PMC 4239492. PMID 25473158.
Trimethylamine is exclusively a microbiota-derived product of nutrients (lecithin, choline, TMAO, L-carnitine) from normal diet, from which seems originate two diseases, trimethylaminuria (or Fish-Odor Syndrome) and cardiovascular disease through the proatherogenic property of its oxidized liver-derived form.
- ^ a b «NIH Human Microbiome Project defines normal bacterial makeup of the body». NIH News. 13 June 2012.
- ^ a b American Academy of Microbiology FAQ: Human Microbiome Archived 31 December 2016 at the Wayback Machine January 2014
- ^ Rosner, Judah L. (1 February 2014). «Ten Times More Microbial Cells than Body Cells in Humans?». Microbe Magazine. 9 (2): 47. doi:10.1128/microbe.9.47.2. ISSN 1558-7452.
- ^ Abbott, Alison (8 January 2016). «Scientists bust myth that our bodies have more bacteria than human cells». Nature. doi:10.1038/nature.2016.19136. ISSN 1476-4687. S2CID 190879263.
- ^ Gilbert JA, Blaser MJ, Caporaso JG, Jansson JK, Lynch SV, Knight R (April 2018). «Current understanding of the human microbiome». Nature Medicine. 24 (4): 392–400. doi:10.1038/nm.4517. PMC 7043356. PMID 29634682.
- ^ a b c Peterson J, Garges S, Giovanni M, McInnes P, Wang L, Schloss JA, et al. (December 2009). «The NIH Human Microbiome Project». Genome Research. 19 (12): 2317–23. doi:10.1101/gr.096651.109. PMC 2792171. PMID 19819907.
- ^ Kuczynski J, Lauber CL, Walters WA, Parfrey LW, Clemente JC, Gevers D, Knight R (December 2011). «Experimental and analytical tools for studying the human microbiome». Nature Reviews. Genetics. 13 (1): 47–58. doi:10.1038/nrg3129. PMC 5119550. PMID 22179717.
- ^ Vestheim H, Jarman SN (July 2008). «Blocking primers to enhance PCR amplification of rare sequences in mixed samples — a case study on prey DNA in Antarctic krill stomachs». Frontiers in Zoology. 5: 12. doi:10.1186/1742-9994-5-12. PMC 2517594. PMID 18638418.
- ^ Tap J, Mondot S, Levenez F, Pelletier E, Caron C, Furet JP, et al. (October 2009). «Towards the human intestinal microbiota phylogenetic core». Environmental Microbiology. 11 (10): 2574–84. doi:10.1111/j.1462-2920.2009.01982.x. PMID 19601958.
- ^ Hamady M, Knight R (July 2009). «Microbial community profiling for human microbiome projects: Tools, techniques, and challenges». Genome Research. 19 (7): 1141–52. doi:10.1101/gr.085464.108. PMC 3776646. PMID 19383763.
- ^ Methé BA, Nelson KE, Pop M, Creasy HH, Giglio MG, Huttenhower C, et al. (Human Microbiome Project Consortium) (June 2012). «A framework for human microbiome research». Nature. 486 (7402): 215–21. Bibcode:2012Natur.486..215T. doi:10.1038/nature11209. PMC 3377744. PMID 22699610.
- ^ The Human Microbiome Project Consortium (June 2012). «Structure, function and diversity of the healthy human microbiome». Nature. 486 (7402): 207–14. Bibcode:2012Natur.486..207T. doi:10.1038/nature11234. PMC 3564958. PMID 22699609.
- ^ Quince C, Walker AW, Simpson JT, Loman NJ, Segata N (September 2017). «Shotgun metagenomics, from sampling to analysis» (PDF). Nature Biotechnology. 35 (9): 833–844. doi:10.1038/nbt.3935. hdl:2164/10167. PMID 28898207. S2CID 19041044.
- ^ Claesson MJ, Clooney AG, O’Toole PW (October 2017). «A clinician’s guide to microbiome analysis». Nature Reviews. Gastroenterology & Hepatology. 14 (10): 585–595. doi:10.1038/nrgastro.2017.97. PMID 28790452. S2CID 24644894.
- ^ a b c Knight R, Vrbanac A, Taylor BC, Aksenov A, Callewaert C, Debelius J, et al. (July 2018). «Best practices for analysing microbiomes». Nature Reviews. Microbiology. 16 (7): 410–422. doi:10.1038/s41579-018-0029-9. PMID 29795328. S2CID 43936002.
- ^ a b Washburne AD, Morton JT, Sanders J, McDonald D, Zhu Q, Oliverio AM, Knight R (June 2018). «Methods for phylogenetic analysis of microbiome data». Nature Microbiology. 3 (6): 652–661. doi:10.1038/s41564-018-0156-0. PMID 29795540. S2CID 43962376.
- ^ Berg, Gabriele; et al. (2020). «Microbiome definition re-visited: Old concepts and new challenges». Microbiome. 8 (1): 103. doi:10.1186/s40168-020-00875-0. PMC 7329523. PMID 32605663.
- ^ Faust, Karoline; Raes, Jeroen (2016). «Rules of the game for microbiota». Nature. 534 (7606): 182–183. doi:10.1038/534182a. PMID 27279206. S2CID 205089271.
- ^ Bashan, Amir; Gibson, Travis E.; Friedman, Jonathan; Carey, Vincent J.; Weiss, Scott T.; Hohmann, Elizabeth L.; Liu, Yang-Yu (2016). «Universality of human microbial dynamics». Nature. 534 (7606): 259–262. Bibcode:2016Natur.534..259B. doi:10.1038/nature18301. PMC 4902290. PMID 27279224.
- ^ Liu, Yang-Yu (15 February 2023). «Controlling the human microbiome» (PDF). Cell Systems. 14 (2): 135–159. doi:10.1016/j.cels.2022.12.010. PMC 9942095. PMID 36796332.
- ^ PLoS Human Microbiome Project Collection Manuscript Summaries Archived 4 March 2014 at the Wayback Machine 13 June 2012
- ^ «Consortium of Scientists Map the Human Body’s Bacterial Ecosystem». ucsf.edu. 13 June 2012.
- ^ Sommer F, Bäckhed F (April 2013). «The gut microbiota—masters of host development and physiology». Nature Reviews. Microbiology. 11 (4): 227–38. doi:10.1038/nrmicro2974. PMID 23435359. S2CID 22798964.
- ^ Eckburg PB, Bik EM, Bernstein CN, Purdom E, Dethlefsen L, Sargent M, et al. (June 2005). «Diversity of the human intestinal microbial flora». Science. 308 (5728): 1635–8. Bibcode:2005Sci…308.1635E. doi:10.1126/science.1110591. PMC 1395357. PMID 15831718.
- ^ Duncan SH, Louis P, Flint HJ (April 2007). «Cultivable bacterial diversity from the human colon». Letters in Applied Microbiology. 44 (4): 343–50. doi:10.1111/j.1472-765X.2007.02129.x. PMID 17397470. S2CID 43706882.
- ^ Florin TH, Zhu G, Kirk KM, Martin NG (October 2000). «Shared and unique environmental factors determine the ecology of methanogens in humans and rats». The American Journal of Gastroenterology. 95 (10): 2872–9. CiteSeerX 10.1.1.606.4187. doi:10.1111/j.1572-0241.2000.02319.x. PMID 11051362. S2CID 1087298.
- ^ Eckburg PB, Lepp PW, Relman DA (February 2003). «Archaea and their potential role in human disease». Infection and Immunity. 71 (2): 591–6. doi:10.1128/IAI.71.2.591-596.2003. PMC 145348. PMID 12540534.
- ^ Cavicchioli R, Curmi PM, Saunders N, Thomas T (November 2003). «Pathogenic archaea: do they exist?». BioEssays. 25 (11): 1119–28. doi:10.1002/bies.10354. PMID 14579252.
- ^ Lepp PW, Brinig MM, Ouverney CC, Palm K, Armitage GC, Relman DA (April 2004). «Methanogenic Archaea and human periodontal disease». Proceedings of the National Academy of Sciences of the United States of America. 101 (16): 6176–81. Bibcode:2004PNAS..101.6176L. doi:10.1073/pnas.0308766101. PMC 395942. PMID 15067114.
- ^ a b c d e f g h i j k Cui L, Morris A, Ghedin E (July 2013). «The human mycobiome in health and disease». Genome Medicine. 5 (7): 63. doi:10.1186/gm467. PMC 3978422. PMID 23899327. Figure 2: Distribution of fungal genera in different body sites
- ^ a b Martins N, Ferreira IC, Barros L, Silva S, Henriques M (June 2014). «Candidiasis: predisposing factors, prevention, diagnosis and alternative treatment» (PDF). Mycopathologia. 177 (5–6): 223–40. doi:10.1007/s11046-014-9749-1. hdl:10198/10147. PMID 24789109. S2CID 795450.
Candida species and other microorganisms are involved in this complicated fungal infection, but Candida albicans continues to be the most prevalent. In the past two decades, it has been observed an abnormal overgrowth in the gastrointestinal, urinary and respiratory tracts, not only in immunocompromised patients, but also related to nosocomial infections and even in healthy individuals. There is a widely variety of causal factors that contribute to yeast infection which means that candidiasis is a good example of a multifactorial syndrome.
- ^ a b c d e Wang ZK, Yang YS, Stefka AT, Sun G, Peng LH (April 2014). «Review article: fungal microbiota and digestive diseases». Alimentary Pharmacology & Therapeutics. 39 (8): 751–66. doi:10.1111/apt.12665. PMID 24612332. S2CID 22101484.
In addition, GI fungal infection is reported even among those patients with normal immune status. Digestive system-related fungal infections may be induced by both commensal opportunistic fungi and exogenous pathogenic fungi. … Candida sp. is also the most frequently identified species among patients with gastric IFI. … It was once believed that gastric acid could kill microbes entering the stomach and that the unique ecological environment of the stomach was not suitable for microbial colonisation or infection. However, several studies using culture-independent methods confirmed that large numbers of acid-resistant bacteria belonging to eight phyla and up to 120 species exist in the stomach, such as Streptococcus sp., Neisseria sp. and Lactobacillus sp. etc.26, 27 Furthermore, Candida albicans can grow well in highly acidic environments,28 and some genotypes may increase the severity of gastric mucosal lesions.29
- ^ a b c Erdogan A, Rao SS (April 2015). «Small intestinal fungal overgrowth». Current Gastroenterology Reports. 17 (4): 16. doi:10.1007/s11894-015-0436-2. PMID 25786900. S2CID 3098136.
Small intestinal fungal overgrowth (SIFO) is characterized by the presence of excessive number of fungal organisms in the small intestine associated with gastrointestinal (GI) symptoms. Candidiasis is known to cause GI symptoms particularly in immunocompromised patients or those receiving steroids or antibiotics. However, only recently, there is emerging literature that an overgrowth of fungus in the small intestine of non-immunocompromised subjects may cause unexplained GI symptoms. Two recent studies showed that 26 % (24/94) and 25.3 % (38/150) of a series of patients with unexplained GI symptoms had SIFO. The most common symptoms observed in these patients were belching, bloating, indigestion, nausea, diarrhea, and gas. … Fungal-bacterial interaction may act in different ways and may either be synergistic or antagonistic or symbiotic [29]. Some bacteria such as Lactobacillus species can interact and inhibit both the virulence and growth of Candida species in the gut by producing hydrogen peroxide [30]. Any damage to the mucosal barrier or disruption of GI microbiota with chemotherapy or antibiotic use, inflammatory processes, activation of immune molecules and disruption of epithelial repair may all cause fungal overgrowth [27].
- ^ Marcon MJ, Powell DA (April 1992). «Human infections due to Malassezia spp». Clinical Microbiology Reviews. 5 (2): 101–19. doi:10.1128/CMR.5.2.101. PMC 358230. PMID 1576583.
- ^ Roth RR, James WD (1988). «Microbial ecology of the skin». Annual Review of Microbiology. 42 (1): 441–64. doi:10.1146/annurev.mi.42.100188.002301. PMID 3144238.
- ^ Hannigan GD, Meisel JS, Tyldsley AS, Zheng Q, Hodkinson BP, SanMiguel AJ, et al. (October 2015). «The human skin double-stranded DNA virome: topographical and temporal diversity, genetic enrichment, and dynamic associations with the host microbiome». mBio. 6 (5): e01578-15. doi:10.1128/mBio.01578-15. PMC 4620475. PMID 26489866.
- ^ Minot S, Sinha R, Chen J, Li H, Keilbaugh SA, Wu GD, et al. (October 2011). «The human gut virome: inter-individual variation and dynamic response to diet». Genome Research. 21 (10): 1616–25. doi:10.1101/gr.122705.111. PMC 3202279. PMID 21880779.
- ^ Young JC, Chehoud C, Bittinger K, Bailey A, Diamond JM, Cantu E, et al. (January 2015). «Viral metagenomics reveal blooms of anelloviruses in the respiratory tract of lung transplant recipients». American Journal of Transplantation. 15 (1): 200–9. doi:10.1111/ajt.13031. PMC 4276431. PMID 25403800.
- ^ Abeles SR, Robles-Sikisaka R, Ly M, Lum AG, Salzman J, Boehm TK, Pride DT (September 2014). «Human oral viruses are personal, persistent and gender-consistent». The ISME Journal. 8 (9): 1753–67. doi:10.1038/ismej.2014.31. PMC 4139723. PMID 24646696.
- ^ Ly M, Abeles SR, Boehm TK, Robles-Sikisaka R, Naidu M, Santiago-Rodriguez T, Pride DT (May 2014). «Altered oral viral ecology in association with periodontal disease». mBio. 5 (3): e01133-14. doi:10.1128/mBio.01133-14. PMC 4030452. PMID 24846382.
- ^ Monaco CL, Gootenberg DB, Zhao G, Handley SA, Ghebremichael MS, Lim ES, et al. (March 2016). «Altered Virome and Bacterial Microbiome in Human Immunodeficiency Virus-Associated Acquired Immunodeficiency Syndrome». Cell Host & Microbe. 19 (3): 311–22. doi:10.1016/j.chom.2016.02.011. PMC 4821831. PMID 26962942.
- ^ Norman JM, Handley SA, Baldridge MT, Droit L, Liu CY, Keller BC, et al. (January 2015). «Disease-specific alterations in the enteric virome in inflammatory bowel disease». Cell. 160 (3): 447–60. doi:10.1016/j.cell.2015.01.002. PMC 4312520. PMID 25619688.
- ^ Grice EA, Kong HH, Conlan S, Deming CB, Davis J, Young AC, et al. (NISC Comparative Sequencing Program) (May 2009). «Topographical and temporal diversity of the human skin microbiome». Science. 324 (5931): 1190–2. Bibcode:2009Sci…324.1190G. doi:10.1126/science.1171700. PMC 2805064. PMID 19478181.
- ^ a b «The Normal Bacterial Flora of Humans». textbookofbacteriology.net.
- ^ a b c d e f g h i Zhang LS, Davies SS (April 2016). «Microbial metabolism of dietary components to bioactive metabolites: opportunities for new therapeutic interventions». Genome Med. 8 (1): 46. doi:10.1186/s13073-016-0296-x. PMC 4840492. PMID 27102537.
Lactobacillus spp. convert tryptophan to indole-3-aldehyde (I3A) through unidentified enzymes [125]. Clostridium sporogenes convert tryptophan to IPA [6], likely via a tryptophan deaminase. … IPA also potently scavenges hydroxyl radicals
Table 2: Microbial metabolites: their synthesis, mechanisms of action, and effects on health and disease
Figure 1: Molecular mechanisms of action of indole and its metabolites on host physiology and disease - ^ Wikoff WR, Anfora AT, Liu J, Schultz PG, Lesley SA, Peters EC, Siuzdak G (March 2009). «Metabolomics analysis reveals large effects of gut microflora on mammalian blood metabolites». Proc. Natl. Acad. Sci. U.S.A. 106 (10): 3698–3703. Bibcode:2009PNAS..106.3698W. doi:10.1073/pnas.0812874106. PMC 2656143. PMID 19234110.
Production of IPA was shown to be completely dependent on the presence of gut microflora and could be established by colonization with the bacterium Clostridium sporogenes.
IPA metabolism diagram - ^ «3-Indolepropionic acid». Human Metabolome Database. University of Alberta. Retrieved 12 June 2018.
- ^ Chyan YJ, Poeggeler B, Omar RA, Chain DG, Frangione B, Ghiso J, Pappolla MA (July 1999). «Potent neuroprotective properties against the Alzheimer beta-amyloid by an endogenous melatonin-related indole structure, indole-3-propionic acid». J. Biol. Chem. 274 (31): 21937–21942. doi:10.1074/jbc.274.31.21937. PMID 10419516. S2CID 6630247.
[Indole-3-propionic acid (IPA)] has previously been identified in the plasma and cerebrospinal fluid of humans, but its functions are not known. … In kinetic competition experiments using free radical-trapping agents, the capacity of IPA to scavenge hydroxyl radicals exceeded that of melatonin, an indoleamine considered to be the most potent naturally occurring scavenger of free radicals. In contrast with other antioxidants, IPA was not converted to reactive intermediates with pro-oxidant activity.
- ^ Yang I, Corwin EJ, Brennan PA, Jordan S, Murphy JR, Dunlop A (2016). «The Infant Microbiome: Implications for Infant Health and Neurocognitive Development». Nursing Research. 65 (1): 76–88. doi:10.1097/NNR.0000000000000133. PMC 4681407. PMID 26657483.
- ^ Mueller NT, Bakacs E, Combellick J, Grigoryan Z, Dominguez-Bello MG (February 2015). «The infant microbiome development: mom matters». Trends in Molecular Medicine. 21 (2): 109–17. doi:10.1016/j.molmed.2014.12.002. PMC 4464665. PMID 25578246.
- ^ Wall R, Ross RP, Ryan CA, Hussey S, Murphy B, Fitzgerald GF, Stanton C (4 March 2009). «Role of gut microbiota in early infant development». Clinical Medicine. Pediatrics. 3: 45–54. doi:10.4137/cmped.s2008. PMC 3676293. PMID 23818794.
- ^ a b c Clarke G, Stilling RM, Kennedy PJ, Stanton C, Cryan JF, Dinan TG (August 2014). «Minireview: Gut microbiota: the neglected endocrine organ». Molecular Endocrinology. 28 (8): 1221–38. doi:10.1210/me.2014-1108. PMC 5414803. PMID 24892638.
- ^ a b Shen S, Wong CH (April 2016). «Bugging inflammation: role of the gut microbiota». Clinical & Translational Immunology. 5 (4): e72. doi:10.1038/cti.2016.12. PMC 4855262. PMID 27195115.
- ^ Wang H, Lee IS, Braun C, Enck P (October 2016). «Effect of Probiotics on Central Nervous System Functions in Animals and Humans: A Systematic Review». Journal of Neurogastroenterology and Motility. 22 (4): 589–605. doi:10.5056/jnm16018. PMC 5056568. PMID 27413138.
These probiotics showed efficacy in improving psychiatric disorder-related behaviors including anxiety, depression, autism spectrum disorder (ASD), obsessive-compulsive disorder, and memory abilities, including spatial and non-spatial memory. Because many of the basic science studies showed some efficacy of probiotics on central nervous system function, this background may guide and promote further preclinical and clinical studies. … According to the qualitative analyses of current studies, we can provisionally draw the conclusion that B. longum, B. breve, B. infantis, L. helveticus, L. rhamnosus, L. plantarum, and L. casei were most effective in improving CNS function, including psychiatric disease-associated functions (anxiety, depression, mood, stress response) and memory abilities
- ^ a b Drake MJ, Morris N, Apostolidis A, Rahnama’i MS, Marchesi JR (April 2017). «The urinary microbiome and its contribution to lower urinary tract symptoms; ICI-RS 2015». Neurourology and Urodynamics. 36 (4): 850–853. doi:10.1002/nau.23006. hdl:1983/3b024f95-9f86-406a-9be3-ce35984b8de1. PMID 28444712. S2CID 27636043.
- ^ a b Aragón IM, Herrera-Imbroda B, Queipo-Ortuño MI, Castillo E, Del Moral JS, Gómez-Millán J, et al. (January 2018). «The Urinary Tract Microbiome in Health and Disease». European Urology Focus. 4 (1): 128–138. doi:10.1016/j.euf.2016.11.001. PMID 28753805.
- ^ a b Schmiemann G, Kniehl E, Gebhardt K, Matejczyk MM, Hummers-Pradier E (May 2010). «The diagnosis of urinary tract infection: a systematic review». Deutsches Ärzteblatt International. 107 (21): 361–7. doi:10.3238/arztebl.2010.0361. PMC 2883276. PMID 20539810.
- ^ Al, Kait F.; Denstedt, John D.; Daisley, Brendan A.; Bjazevic, Jennifer; Welk, Blayne K.; Pautler, Stephen E.; Gloor, Gregory B.; Reid, Gregor; Razvi, Hassan; Burton, Jeremy P. (September 2020). «Ureteral Stent Microbiota Is Associated with Patient Comorbidities but Not Antibiotic Exposure». Cell Reports Medicine. 1 (6): 100094. doi:10.1016/j.xcrm.2020.100094. PMC 7659606. PMID 33205072.
- ^ Wolfe AJ, Brubaker L (February 2019). «Urobiome updates: advances in urinary microbiome research». Nature Reviews. Urology. 16 (2): 73–74. doi:10.1038/s41585-018-0127-5. PMC 6628711. PMID 30510275.
- ^ a b Petrova MI, Lievens E, Malik S, Imholz N, Lebeer S (2015). «Lactobacillus species as biomarkers and agents that can promote various aspects of vaginal health». Frontiers in Physiology. 6: 81. doi:10.3389/fphys.2015.00081. PMC 4373506. PMID 25859220.
- ^ a b c d Witkin SS, Linhares IM, Giraldo P (June 2007). «Bacterial flora of the female genital tract: function and immune regulation». Best Practice & Research. Clinical Obstetrics & Gynaecology. 21 (3): 347–54. doi:10.1016/j.bpobgyn.2006.12.004. PMID 17215167.
- ^ Todar K (2012). «The Normal Bacterial Flora of Humans». Todar’s Online Textbook of Bacteriology. Madison, WI: Kenneth Todar. Retrieved 6 April 2012.
- ^ Onderdonk AB, Zamarchi GR, Walsh JA, Mellor RD, Muñoz A, Kass EH (February 1986). «Methods for quantitative and qualitative evaluation of vaginal microflora during menstruation». Applied and Environmental Microbiology. 51 (2): 333–9. Bibcode:1986ApEnM..51..333O. doi:10.1128/AEM.51.2.333-339.1986. PMC 238869. PMID 3954346.
- ^ Antonio MA, Hawes SE, Hillier SL (December 1999). «The identification of vaginal Lactobacillus species and the demographic and microbiologic characteristics of women colonized by these species». The Journal of Infectious Diseases. 180 (6): 1950–6. doi:10.1086/315109. PMID 10558952.
- ^ Usyk M, Zolnik CP, Castle PE, Porras C, Herrero R, Gradissimo A, et al. (March 2020). «Cervicovaginal microbiome and natural history of HPV in a longitudinal study». PLOS Pathogens. 16 (3): e1008376. doi:10.1371/journal.ppat.1008376. PMC 7098574. PMID 32214382.
- ^ Fox C, Eichelberger K (December 2015). «Maternal microbiome and pregnancy outcomes». Fertility and Sterility. 104 (6): 1358–63. doi:10.1016/j.fertnstert.2015.09.037. PMID 26493119.
- ^ Wassenaar TM, Panigrahi P (December 2014). «Is a foetus developing in a sterile environment?». Letters in Applied Microbiology. 59 (6): 572–9. doi:10.1111/lam.12334. PMID 25273890. S2CID 206169539.
- ^ Schwiertz A (2016). Microbiota of the human body : implications in health and disease. Switzerland: Springer. p. 1. ISBN 978-3-319-31248-4.
- ^ Tamburini S, Shen N, Wu HC, Clemente JC (July 2016). «The microbiome in early life: implications for health outcomes». Nature Medicine. 22 (7): 713–22. doi:10.1038/nm.4142. PMID 27387886. S2CID 2462790.
- ^ de Goffau MC, Lager S, Sovio U, Gaccioli F, Cook E, Peacock SJ, et al. (August 2019). «Human placenta has no microbiome but can contain potential pathogens». Nature. 572 (7769): 329–334. Bibcode:2019Natur.572..329D. doi:10.1038/s41586-019-1451-5. PMC 6697540. PMID 31367035.
- ^ Eisenhofer R, Minich JJ, Marotz C, Cooper A, Knight R, Weyrich LS (February 2019). «Contamination in Low Microbial Biomass Microbiome Studies: Issues and Recommendations». Trends in Microbiology. 27 (2): 105–117. doi:10.1016/j.tim.2018.11.003. hdl:2440/122027. PMID 30497919. S2CID 54166123.
- ^ Franasiak JM, Scott RT (December 2015). «Reproductive tract microbiome in assisted reproductive technologies». Fertility and Sterility. 104 (6): 1364–71. doi:10.1016/j.fertnstert.2015.10.012. PMID 26597628.
- ^ Sutter VL (1984). «Anaerobes as normal oral flora». Reviews of Infectious Diseases. 6 (Suppl 1): S62-566. doi:10.1093/clinids/6.Supplement_1.S62. PMID 6372039.
- ^ a b c d e Kumar PS (December 2013). «Oral microbiota and systemic disease». Anaerobe. 24: 90–3. doi:10.1016/j.anaerobe.2013.09.010. PMID 24128801. S2CID 40735283.
- ^ Arweiler NB, Netuschil L (May 2016). «The Oral Microbiota». In Schwiertz A (ed.). Microbiota of the Human Body: Implications in Health and Disease. Advances in Experimental Medicine and Biology. Vol. 902. Springer, Cham. pp. 45–60. doi:10.1007/978-3-319-31248-4_4. ISBN 978-3-319-31248-4. PMID 27161350.
- ^ a b c d Avila M, Ojcius DM, Yilmaz O (August 2009). «The oral microbiota: living with a permanent guest». DNA and Cell Biology. 28 (8): 405–11. doi:10.1089/dna.2009.0874. PMC 2768665. PMID 19485767.
- ^ a b Rogers AH, ed. (2008). Molecular Oral Microbiology. Caister Academic Press. ISBN 978-1-904455-24-0.
- ^ a b c d e Zarco MF, Vess TJ, Ginsburg GS (March 2012). «The oral microbiome in health and disease and the potential impact on personalized dental medicine». Oral Diseases. 18 (2): 109–20. doi:10.1111/j.1601-0825.2011.01851.x. PMID 21902769. S2CID 24411104.
- ^ Rhoades NS, Pinski AN, Monsibais AN, Jankeel A, Doratt BM, Cinco IR, et al. (August 2021). «Acute SARS-CoV-2 infection is associated with an increased abundance of bacterial pathogens, including Pseudomonas aeruginosa in the nose». Cell Reports. 36 (9): 109637. doi:10.1016/j.celrep.2021.109637. ISSN 2211-1247. PMC 8361213. PMID 34433082.
- ^ Wing Ho Man; Wouter A A de Steenhuijsen Piters; Debby Bogaert (2017). «The microbiota of the respiratory tract: gatekeeper to respiratory health». Nature Reviews Microbiology (published 20 March 2017). 15 (5): 259–270. doi:10.1038/NRMICRO.2017.14. hdl:20.500.11820/f1137874-9c51-401b-bca4-e2a5da3e219b. ISSN 1740-1534. PMC 7097736. PMID 28316330.Wikidata: Q34553608
- ^ a b Beringer PM, Appleman MD (November 2000). «Unusual respiratory bacterial flora in cystic fibrosis: microbiologic and clinical features» (PDF). Current Opinion in Pulmonary Medicine. 6 (6): 545–50. doi:10.1097/00063198-200011000-00015. PMID 11100967. S2CID 845977. Archived from the original (PDF) on 16 October 2013.
- ^ Verdier J, Luedde T, Sellge G (June 2015). «Biliary Mucosal Barrier and Microbiome». Viszeralmedizin. 31 (3): 156–61. doi:10.1159/000431071. PMC 4569210. PMID 26468308.
- ^ Yu B, Yu B, Yu L (June 2020). «Commentary: Reconciling Hygiene and Cleanliness: A New Perspective from Human Microbiome». Indian Journal of Microbiology. 60 (2): 259–261. doi:10.1007/s12088-020-00863-w. PMC 7105528. PMID 32255860.
- ^ Copeland CS. The World Within Us: Health and the Human Microbiome. Healthcare Journal of New Orleans, Sept-Oct 2017.
- ^ Honda K, Littman DR (July 2016). «The microbiota in adaptive immune homeostasis and disease». Nature. 535 (7610): 75–84. Bibcode:2016Natur.535…75H. doi:10.1038/nature18848. PMID 27383982. S2CID 4461492.
- ^ «Serum metabolites reflecting gut microbiome alpha diversity predict type 2 diabetes». Metabolon. Retrieved 3 November 2022.
- ^ Liubakka A, Vaughn BP (July 2016). «Clostridium difficile Infection and Fecal Microbiota Transplant». AACN Advanced Critical Care. 27 (3): 324–337. doi:10.4037/aacnacc2016703. PMC 5666691. PMID 27959316.
- ^ Burton JH, Johnson M, Johnson J, Hsia DS, Greenway FL, Heiman ML (July 2015). «Addition of a Gastrointestinal Microbiome Modulator to Metformin Improves Metformin Tolerance and Fasting Glucose Levels». Journal of Diabetes Science and Technology. 9 (4): 808–14. doi:10.1177/1932296815577425. PMC 4525649. PMID 25802471.
- ^ a b Bakken JS, Borody T, Brandt LJ, Brill JV, Demarco DC, Franzos MA, et al. (December 2011). «Treating Clostridium difficile infection with fecal microbiota transplantation». Clinical Gastroenterology and Hepatology. 9 (12): 1044–9. doi:10.1016/j.cgh.2011.08.014. PMC 3223289. PMID 21871249.
- ^ Gough E, Shaikh H, Manges AR (November 2011). «Systematic review of intestinal microbiota transplantation (fecal bacteriotherapy) for recurrent Clostridium difficile infection». Clinical Infectious Diseases. 53 (10): 994–1002. doi:10.1093/cid/cir632. PMID 22002980.
- ^ Brown WR (August 2014). «Fecal microbiota transplantation in treating Clostridium difficile infection». Journal of Digestive Diseases. 15 (8): 405–8. doi:10.1111/1751-2980.12160. PMID 24825534. S2CID 44651256.
- ^ Burke KE, Lamont JT (August 2013). «Fecal transplantation for recurrent Clostridium difficile infection in older adults: a review». Journal of the American Geriatrics Society. 61 (8): 1394–8. doi:10.1111/jgs.12378. PMID 23869970. S2CID 34998497.
- ^ Drekonja D, Reich J, Gezahegn S, Greer N, Shaukat A, MacDonald R, et al. (May 2015). «Fecal Microbiota Transplantation for Clostridium difficile Infection: A Systematic Review». Annals of Internal Medicine. 162 (9): 630–8. doi:10.7326/m14-2693. PMID 25938992. S2CID 1307726.
- ^ Brandt LJ, Borody TJ, Campbell J (September 2011). «Endoscopic fecal microbiota transplantation: «first-line» treatment for severe clostridium difficile infection?». Journal of Clinical Gastroenterology. 45 (8): 655–7. doi:10.1097/MCG.0b013e3182257d4f. PMID 21716124. S2CID 2508836.
- ^ Kelly CR, de Leon L, Jasutkar N (February 2012). «Fecal microbiota transplantation for relapsing Clostridium difficile infection in 26 patients: methodology and results». Journal of Clinical Gastroenterology. 46 (2): 145–9. doi:10.1097/MCG.0b013e318234570b. PMID 22157239. S2CID 30849491.
- ^ a b c d e f g h Garrett WS (April 2015). «Cancer and the microbiota». Science. 348 (6230): 80–6. Bibcode:2015Sci…348…80G. doi:10.1126/science.aaa4972. PMC 5535753. PMID 25838377.
- ^ a b c d Gagnière J, Raisch J, Veziant J, Barnich N, Bonnet R, Buc E, et al. (January 2016). «Gut microbiota imbalance and colorectal cancer». World Journal of Gastroenterology. 22 (2): 501–18. doi:10.3748/wjg.v22.i2.501. PMC 4716055. PMID 26811603.
- ^ a b Sartor RB, Mazmanian SK (July 2012). «Intestinal Microbes in Inflammatory Bowel Diseases». The American Journal of Gastroenterology Supplements. 1 (1): 15–21. doi:10.1038/ajgsup.2012.4.
- ^ Hold GL, Smith M, Grange C, Watt ER, El-Omar EM, Mukhopadhya I (February 2014). «Role of the gut microbiota in inflammatory bowel disease pathogenesis: what have we learnt in the past 10 years?». World Journal of Gastroenterology. 20 (5): 1192–210. doi:10.3748/wjg.v20.i5.1192. PMC 3921503. PMID 24574795.
- ^ a b Zilberman-Schapira G, Zmora N, Itav S, Bashiardes S, Elinav H, Elinav E (June 2016). «The gut microbiome in human immunodeficiency virus infection». BMC Medicine. 14 (1): 83. doi:10.1186/s12916-016-0625-3. PMC 4891875. PMID 27256449.
- ^ Petrova MI, van den Broek M, Balzarini J, Vanderleyden J, Lebeer S (September 2013). «Vaginal microbiota and its role in HIV transmission and infection». FEMS Microbiology Reviews. 37 (5): 762–92. doi:10.1111/1574-6976.12029. PMID 23789590.
- ^ a b Sato Y, Atarashi K, Plichta DR, Arai Y, Sasajima S, Kearney SM, Suda W, Takeshita K, Sasaki T, Okamoto S, Skelly AN, Okamura Y, Vlamakis H, Li Y, Tanoue T, Takei H, Nittono H, Narushima S, Irie J, Itoh H, Moriya K, Sugiura Y, Suematsu M, Moritoki N, Shibata S, Littman DR, Fischbach MA, Uwamino Y, Inoue T, Honda A, Hattori M, Murai T, Xavier RJ, Hirose N, Honda K. Novel bile acid biosynthetic pathways are enriched in the microbiome of centenarians. Nature. 2021 Nov;599(7885):458-464. doi: 10.1038/s41586-021-03832-5. Epub 2021 Jul 29. PMID 34325466
- ^ Intagliata C (22 December 2016). ««Necrobiome» Reveals a Corpse’s Time of Death». Scientific American. Retrieved 26 March 2018.
- ^ Young E (10 December 2015). «Meet the Necrobiome: The Waves of Microbes That Will Eat Your Corpse». The Atlantic. Retrieved 26 March 2018.
- ^ Harmon K (16 December 2009). «Bugs Inside: What Happens When the Microbes That Keep Us Healthy Disappear?». Scientific American. Retrieved 27 December 2008.
- ^ a b Hitch, Thomas C. A.; Hall, Lindsay J.; Walsh, Sarah Kate; Leventhal, Gabriel E.; Slack, Emma; de Wouters, Tomas; Walter, Jens; Clavel, Thomas (June 2022). «Microbiome-based interventions to modulate gut ecology and the immune system». Mucosal Immunology. 15 (6): 1095–1113. doi:10.1038/s41385-022-00564-1. ISSN 1935-3456. PMC 9705255. PMID 36180583.
- ^ Callewaert, Chris; Knödlseder, Nastassia; Karoglan, Ante; Güell, Marc; Paetzold, Bernhard (1 January 2021). «Skin microbiome transplantation and manipulation: Current state of the art». Computational and Structural Biotechnology Journal. 19: 624–631. doi:10.1016/j.csbj.2021.01.001. ISSN 2001-0370. PMC 7806958. PMID 33510866.
- ^ Monda, Vincenzo; Villano, Ines; Messina, Antonietta; Valenzano, Anna; Esposito, Teresa; Moscatelli, Fiorenzo; Viggiano, Andrea; Cibelli, Giuseppe; Chieffi, Sergio; Monda, Marcellino; Messina, Giovanni (5 March 2017). «Exercise Modifies the Gut Microbiota with Positive Health Effects». Oxidative Medicine and Cellular Longevity. 2017: e3831972. doi:10.1155/2017/3831972. ISSN 1942-0900. PMC 5357536. PMID 28357027.
- ^ Mailing, Lucy J.; Allen, Jacob M.; Buford, Thomas W.; Fields, Christopher J.; Woods, Jeffrey A. (April 2019). «Exercise and the Gut Microbiome: A Review of the Evidence, Potential Mechanisms, and Implications for Human Health». Exercise and Sport Sciences Reviews. 47 (2): 75–85. doi:10.1249/JES.0000000000000183. ISSN 0091-6331. PMID 30883471. S2CID 83461620.
- ^ a b Vangay P, Johnson AJ, Ward TL, Al-Ghalith GA, Shields-Cutler RR, Hillmann BM, et al. (November 2018). «US Immigration Westernizes the Human Gut Microbiome». Cell. 175 (4): 962–972.e10. doi:10.1016/j.cell.2018.10.029. PMC 6498444. PMID 30388453.
- ^ a b Kaplan RC, Wang Z, Usyk M, Sotres-Alvarez D, Daviglus ML, Schneiderman N, et al. (November 2019). «Gut microbiome composition in the Hispanic Community Health Study/Study of Latinos is shaped by geographic relocation, environmental factors, and obesity». Genome Biology. 20 (1): 219. doi:10.1186/s13059-019-1831-z. PMC 6824043. PMID 31672155.
- ^ «Comprehensive profiling of human-to-human oral and intestinal microbial transmission». News-Medical.net. 20 January 2023. Archived from the original on 16 February 2023. Retrieved 16 February 2023.
- ^ Valles-Colomer, Mireia; Blanco-Míguez, Aitor; Manghi, Paolo; et al. (February 2023). «The person-to-person transmission landscape of the gut and oral microbiomes». Nature. 614 (7946): 125–135. Bibcode:2023Natur.614..125V. doi:10.1038/s41586-022-05620-1. ISSN 1476-4687. PMC 9892008. PMID 36653448.
External links[edit]
- The Secret World Inside You Exhibit 2015–2016, American Museum of Natural History
- FAQ: Human Microbiome, January 2014 American Society For Microbiology
Микробиом человека
МИКРОБИОМ ЧЕЛОВЕКА
МИКРОБИОМ ЧЕЛОВЕКА — НОВАЯ ЭРА В ПОНИМАНИИ МИКРОФЛОРЫ
СОДЕРЖАНИЕ
- I. Определение микробиома
- II. Здоровый микробиом человека
- Резюме
- Фон (введение)
- Наше эволюционирующее понимание здорового микробиома
- Базовые когорты в масштабе популяции
- Типичные компоненты и разнообразие микробиома
- Бактериальные компоненты здорового микробиома
- Рис.1. Возможные определения здорового микробиома
- Археи, вирусы, грибы и др. эукариоты
- Географическая изменчивость в здоровом микробиоме
- Установление микробиома и ранняя колонизация
- Отличительные признаки здоровья
- Функциональное ядро
- Здоровая экология микробного сообщества
- Сопротивление, устойчивость, стабильность
- Прогноз
- III. О структуре здорового микробиома человека
- Обильные таксоны в микробиоме человека
- Бактериальные функции, важные для жизни в кишечнике
- IV. Содержание и разнообразие микробного сообщества (от MetaHIT)
- 16S секвенирование
- Метагеномное секвенирование
- Общее бактериальное ядро
- V. Дополнительный материал
- Бактероидеты, фирмикуты и другие
- Питание и конфигурация кишечного микробиома
- VI. Первые результаты проекта HMP (Human Microbiome Project)
- VII. Интегративный проект микробиома человека (2-й этап HMP)
- Введение
- Рис. 1. Первая и вторая фазы проекта NIH Human Microbiome.
- Микробиом влагалища, беременность и преждевременные роды
- Рис. 2. Микробиом влагалища и его связь с факторами хозяина во время беременности и преждевременных родов.
- Микробиом кишечника и воспалительные заболевания кишечника
- Рис. 3. Динамика «хозяин-микробиом» при ВЗК.
- Мульти-омическое профилирование при преддиабете
- Рис. 4. Дифференциальный ответ хозяина и микробов на диетические нарушения и инфекционные заболевания у лиц с преддиабетом.
- Ресурсы из HMP2
- Комплементарные взаимодействия хозяина и микробиома
- Следующие шаги в мульти-омике микробиома
- VIII. Видеолекции о микробиоме + статьи об исследовании микробиом
ОПРЕДЕЛЕНИЕ МИКРОБИОМА ЧЕЛОВЕКА
Бактериальное и микробное «население» человеческого организма отличается исключительно большим разнообразием — число микроорганизмов в организме у разных людей может быть равно числу собственных клеток, а может в десять раз превосходить его, свидетельствуют первые результаты работы международного проекта «Микробиом человека» (Human Microbiome Project — HMP).
Микробиом — собирательное название микроорганизмов, находящихся в симбиозе с организмом хозяина. Также под микробиомом подразумевают совокупность геномов микробных популяций в человеке. Различают микробиом кожи, полости рта, кишечника и т.д.

Вместе с понятием «микробиом» существует и понятие «микробиота». По сути, они несут одно и тоже смысловое значение, особенно если к этим терминам добавлена локализация, например, микробиом кишечника или кишечная микробиота. Однако в отдельности (в строго научной среде) термины понимаются по-разному:
Термин «микробиота» относится к совокупности микробов (бактерий, архей, грибов, вирусов и простейших) в конкретной среде, другими словами, к таксономии и обилию членов сообщества, а «микробиом» — это совокупность геномов микробиоты, и его часто используют для описания сущности микробных признаков (функций), кодируемых микробиотой [Schlaeppi, K.; Bulgarelli, D. The plant microbiome at work. Mol. Plant Microbe Interact.2015, 28, 212–217.].
См. подробнее:
Представления о микробиоте человека
Микробиота используется для характеристики микробиоценоза отдельных органов и систем, генетического материала и взаимосвязей внутри экологической ниши в определенный временной период на определенной территории. Микробиота взаимодействует с остальными органами и системами, определяя функционирование организма в целом как у здорового человека, так и при заболеваниях.
См. отдельно:
Глобальное исследование Микробиома открывает тысячи новых видов, отображает городскую устойчивость к противомикробным препаратам и выявляет новых кандидатов на лекарственные препараты
Здоровый микробиом человека
Jason Lloyd-Price, Galeb Abu-Ali & Curtis Huttenhower
The healthy human microbiome
Genome Medicine volume8, Article number: 51 (2016)
Резюме. Люди практически идентичны по своему генетическому составу, однако небольшие различия в нашей ДНК порождают огромное фенотипическое разнообразие во всей человеческой популяции. В отличие от этого, метагеном человеческого микробиома — общее содержание ДНК микробов, населяющих наши тела, — является довольно изменчивым, и только треть составляющих его генов обнаруживается у большинства здоровых людей. Понимание этой изменчивости в «здоровом микробиоме», таким образом, было одной из главных проблем в исследованиях микробиома, начиная по крайней мере с 1960-х годов, продолжающихся в рамках проекта «Микробиом человека» и за его пределами. Каталогизация необходимых и достаточных наборов признаков микробиома, поддерживающих здоровье, и нормальных диапазонов этих признаков в здоровых популяциях является важным первым шагом к выявлению и коррекции микробных конфигураций, которые вовлечены в болезнь. Для достижения этой цели в ряде популяционных исследований были задокументированы диапазоны и разнообразие как таксономических составов, так и функциональных потенциалов, обычно наблюдаемых в микробиомах здоровых популяций, а также возможные движущие факторы, такие как география, диета и образ жизни. Здесь мы рассмотрим несколько появившихся определений «здорового микробиома», современное понимание диапазонов здорового микробного разнообразия и такие пробелы, как характеристика молекулярной функции и разработка экологических методов лечения, которые будут рассмотрены в будущем.
ФОН
Люди эволюционировали вместе с триллионами микробов, населяющих наши тела и создающих сложные, специфичные для среды обитания адаптивные экосистемы, тонко настроенные на неустанно меняющуюся физиологию хозяина. Дисбактериоз в микробиоме был связан с многочисленными заболеваниями, включая воспалительные заболевания кишечника, рассеянный склероз, сахарный диабет (типы 1 и 2), аллергию, астму, аутизм и рак [1-5]. Как и понятие патогенности отдельного микробного таксона, дисбактериоз микробного сообщества может быть трудно поддающимся определению, но может рассматриваться как возмущение, которое отклоняется от сбалансированной в остальном экологии [1], чтобы продлить, усугубить или вызвать вредное воздействие на здоровье. Таким образом, обнаружение признаков, которые в целом отличают здоровые микробиомы от нездоровых, поможет в диагностике заболеваний, связанных с микробиомом, и потенциально может предоставить новые средства для предотвращения возникновения заболевания или улучшения прогноза. Было предложено много потенциальных признаков, общих для здоровых микробиомов, включая преобладающие организмы или молекулярные пути [6], а также нормы определенных экологических свойств, таких как разнообразие или стабильность [7, 8]. Микробиомы регулярно демонстрируют большую степень межличностного разнообразия даже при отсутствии заболевания [7, 9]. Это затрудняет идентификацию простых микробных компонентов или дисбалансов, которые либо вызывают заболевание, либо отражают болезненное состояние. Таким образом, понимание свойств здорового микробиома и множества различных микробных экологий, которые встречаются в отсутствие явного заболевания, является необходимым первым шагом к выявлению и коррекции микробных конфигураций, вовлеченных в болезнь.
В этом обзоре мы используем термин «здоровый» для обозначения отсутствия какого-либо явного заболевания (как определено в [10], если иное не указано для конкретных исследований). Большинство доступных данных описывают микробиом кишечника, и поэтому многие выводы, обсуждаемые здесь, относятся к этой области, хотя большинство принципов применимы к микробным средам обитания по всему организму. Ранние исследования экологии микробиома были направлены на выявление «основного» набора микробных таксонов, повсеместно присутствующих у здоровых людей, у которых отсутствуют явные фенотипы заболеваний, в соответствии с гипотезой, что отсутствие таких микробов будет указывать на дисбактериоз [11]; но исследования экологического разнообразия среди здоровых особей выявили достаточную вариабельность в таксономическом составе микробиома, чтобы быстро сделать такую гипотезу маловероятной [11, 12]. Было обнаружено, что даже общие таксоны, от отдельных видов до целых типов, различаются по численности более чем на порядок среди здоровых особей [7, 11]. Поэтому характеристика «здорового» микробиома как идеального набора специфических микробов уже не является практическим определением [2, 6].
Альтернативной гипотезой является гипотеза о здоровом «функциональном ядре»: дополнении метаболических и других молекулярных функций, которые выполняются микробиомом в пределах определенной среды обитания, но не обязательно обеспечиваются одними и теми же организмами у разных людей [6]. Такое ядро, возможно, должно присутствовать как генетический потенциал (то есть закодированный в ДНК-метагеномах), так же как человеческий геном не должен кодировать серьезные вредные мутации, чтобы быть здоровым, или оно должно быть выражено и хорошо отрегулировано внутри индивида, чтобы он оставался здоровым (то есть оно должно быть закодировано метатранскриптомами РНК или присутствовать в виде белковых или мелкомолекулярных продуктов, или, конечно, их комбинации). Функциональное ядро должно, конечно, включать в себя, по крайней мере, хозяйственные функции, необходимые для индивидуальной микробной жизни, которые должны присутствовать геномно и быть правильно выражены; интересно, что эти свойства могут также включать функции, характерные для микробных ниш в человеческой экосистеме. Такие функции могут включать в себя процессы, которые не осуществляются человеческими клетками и, таким образом, представляют собой потенциальную основу для симбиотических отношений хозяина и микроба. Здоровый микробиом может быть дополнительно охарактеризован его поведением с течением времени [2, 8]; интуитивно, связанный со здоровьем микробиом должен обладать определенной степенью устойчивости к внешним (например, диетическим или фармацевтическим) или внутренним (например, возрастным или стохастическим) изменениям. Даже если та или иная структура сообщества обеспечивала все необходимые основные функции, без этой устойчивости она не могла бы гарантировать выполнение этих функций в течение длительного времени. Таким образом, устойчивость микробиома к стрессам и возмущениям и его способность впоследствии восстанавливаться до здорового функционального профиля относятся к числу потенциальных свойств, характеризующих здоровый микробиом [2, 13].
Здесь мы рассматриваем текущую характеристику здорового микробиома с точки зрения нормальных микробных резидентов и их основных функций, экологических свойств и временной динамики. В заключение мы определяем ключевые нерешенные вопросы и направления исследований в этой области и размышляем об их решениях и влиянии. Сочетание последних технологических достижений и деятельности в этой области вызвало всплеск интереса к человеческому микробиому в области здоровья и болезней (Таблица 1), и поэтому настоящий обзор направлен на обобщение различных современных перспектив того, что может представлять собой здоровый микробиом.
Таблица 1. Разнообразие последних исследований микробиома, которые были сосредоточены в основном на кишечнике
|
Условия |
Публикации |
|
|
Все |
2011–2016 |
|
|
Пищеварительный тракт | толстая кишка | кишечник |
17,546 |
10,707 |
|
Оральный | рот | язык | зуб | поддесневой | наддесневой |
4843 |
2089 |
|
Урогенитальный | вагинальный | половой |
1477 |
706 |
|
Кожа | кожный покров |
1372 |
754 |
|
Дыхательные пути | легкие |
764 |
524 |
|
Плацента | грудное молоко |
702 |
426 |
|
Зрительный | глаз |
152 |
82 |
Количество результатов, полученных в результате поиска по запросу (микробиом | микробиота | микрофлора) (<термины>) на PubMed (получено 31 марта 2016 г)
Наше эволюционирующее понимание здорового микробиома

Ранние исследования были направлены на выявление нормального набора микробов, которые колонизируют здоровых людей, прежде всего в кишечнике, по культуре и характеристике физиологических свойств. Такие исследования лучше всего выделяют организмы, которые хорошо растут в лабораторных условиях, такие как Escherichia coli. Это смещение привело к восприятию того, что кишечная палочка является обильным и распространенным членом кишечного микробиома человека [14]. Внедрение строго анаэробных методов в 1970-х годах позволило извлечь из кишечника более 300 видов бактерий [15]; кроме того, подсчет жизнеспособных клеток в стандартизированных серийных разведениях в селективных средах позволил количественно оценить эти виды. Краткое изложение четырех крупных исследований этой эпохи [12], изучив образцы стула 141 американца на разных диетах, обнаружило, что бактерии рода Bacteroides и анаэробные кокки были как преобладающими, так и обильными, в то время как род Clostridium был повсеместным в более низком обилии, хотя и не существовало единого вида (как потом определили) наблюдаемого у всех испытуемых. Другие распространенные, но менее обильные бактерии включали представителей родов Bifidobacterium, Eubacterium, Lactobacillus и Streptococcus, а также факультативные анаэробы, такие как Escherichia.
В то время уже предполагалось, что большое количество видов микроорганизмов, связанных с человеком, так и не было обнаружено, причем одно исследование оценило одновременное присутствие около 400 видов микроорганизмов в здоровой толстой кишке [16, 17]. Тем не менее, привередливые требования некоторых микробов и трудоемкая работа, необходимая для их культивирования, представляли собой значительный барьер для их открытия [12]. Кроме того, не все микробы могут быть хорошо различимы как виды или штаммы путем культивирования только на селективных средах; например, особенно трудно распутать различные высокообильные виды Bacteroides [12, 17]. Кроме того, такие исследования состава сообществ были еще более трудны для распространения на небактериальные микробы, такие как вирусы и грибы, и были еще более непрактичны для изучения местообитаний тела, которые менее богаты микробами, чем кишечник. Для изучения этих аспектов здорового микробиома требовались новые методы.
В настоящее время широкое распространение получили культурально-независимые методы, такие как секвенирование ДНК [18] и флуоресцентная гибридизация in situ (FISH) [19], а их демократизация позволила непосредственно исследовать содержание ДНК в микробных образцах [20]. Ранние исследования с использованием FISH, нацеленных на ген 16S рибосомальной РНК, показали, что по меньшей мере две трети кишечных бактерий в западноевропейской когорте могут быть отнесены к набору из шести групп примерно на уровне вида/рода: два Bacteroides, два Clostridium, Streptococcus/Lactococcus и Eubacterium rectale [19]. С тех пор это оказалось оптимистичным, и даже в то время наблюдалась большая вариабельность в содержании этих групп между выборками (стандартные отклонения ~60-80% от их средних значений) [19].
Некоторые из ранних попыток секвенировать гены 16S рРНК непосредственно из образцов показали, что 85–95% бактериальной численности, соответствующей известным видам, можно отнести к трем бактериальным группам, связанным с Bacteroides, Clostridium кластера XIVa и Clostridium кластера IV [21, 22]. Исследования 16S также показали большое разнообразие в таксономическом составе как между здоровыми людьми, так и среди тесно связанных биогеографических участков в пределах одного человека (таких как образцы слизистой оболочки и кала [23]). Однако во всех этих исследованиях большинство (75–80%) кластеров последовательностей не соответствовало ни одному из документированных видов в то время [21–23], что во многом объясняет недооценку разнообразия в предыдущих работах.
Появление массового параллельного секвенирования методом дробовика (технологии высокопроизводительного секвенирования) существенно изменило таксономический состав этой микробной «темной материи» [24], хотя еще предстоит охарактеризовать поразительный процент функционального разнообразия (до 50 % [25]), как и состав некондиционных популяций (обсуждаемый ниже). Первоначальные находки перекликались с большими межличностными различиями даже между близнецами [26], но также подразумевали существование набора микробных генов, общих для всех индивидов [26, 27]. Это помогло создать модель, согласно которой, подобно сохраненным хозяйственным генам в отдельных организмах, «основной микробиом» может быть определен на функциональном, а не на таксономическом уровне [26, 27].
Базовые когорты в масштабе популяции
С тех пор были начаты крупномасштабные проекты по характеристике разнообразия микробного состава и его функционального потенциала, опираясь на все еще увеличивающуюся пропускную способность и экономическую эффективность секвенирования и других молекулярных анализов. В 2010 году исследование метагеномов кишечного тракта человека (MetaHIT) сообщило о метагеномах кишечника из образцов кала из когорты 124 взрослых европейцев (преимущественно «здоровых»), которые в то время превышали объем секвенирования всех предыдущих исследований микробиома почти в 200 раз [9]. В 2012 году проект Human Microbiome Project (HMP) сообщил о результатах профилирования 16S на 242 здоровых взрослых людях из США и метагеномного секвенирования на подмножестве из 139 индивидов, причем образцы представляли собой 18 местообитаний тела, распределенных между пятью основными областями тела [7]. Крупное китайское исследование диабета 2-го типа вскоре дало дополнительные 145 кишечных метагеномов, примерно половина из которых были получены из недиабетического контроля [28]. Кроме того, консорциум MetaHIT с тех пор продолжает публиковать новые данные кишечных метагеномов от взрослых европейцев [29-31]. В целом, число популяционных здоровых микробиомов, обследованных в кишечнике и других участках тела, в настоящее время превышает 2000 индивидов, охватывающих несколько континентов.
Типичные компоненты и разнообразие микробиома
Бактериальные компоненты здорового микробиома
Экосистема толстой кишки была наиболее интенсивно изученной средой обитания организма (табл. 1), поскольку она может похвастаться замечательным разнообразием между людьми и микробной биомассой (количеством клеток), которая затмевает другие участки тела более чем на порядок [32]. В сочетании с ранним появлением секвенирования генов 16S рРНК и методов анаэробного культивирования эти свойства кишечника привели к тому, что в литературе особое внимание уделяется бактериальным резидентам кишечного микробиома. В настоящее время было охарактеризовано более 1000 видов кишечных бактерий [15], что обеспечивает значительный «список частей» бактериальных компонентов. Интересно, что молекулярная филогенетика привела к реклассификации многих из этих видов за последние 20 лет. Особый интерес представляют виды внутри Bacteroides, ранее считавшимся наиболее распространенным и обильным бактериальным родом в кишечнике, который был реклассифицирован в пять родов: Alistipes, Prevotella, Paraprevotella, Parabacteroides и Odoribacter, при этом дополнительная культуральная и культурально-независимая молекулярная работа продолжается [15]. По оценкам, в кишечных микробиомах когорты MetaHIT преобладали 1000-1150 видов бактерий, из которых каждый человек нес в среднем около 160 видов [9]. В здоровых микробиомах кишечника, оцениваемых с помощью секвенирования, последовательно доминируют бактерии двух типов — Bacteroidetes и Firmicutes [7, 9], хотя даже при рассмотрении этого широкого уровня классификации отдельные особи различаются более чем на порядок в своих соотношениях Firmicutes / Bacteroidetes [7]. Распространенные бактерии в фекалиях, которые были идентифицированы с помощью молекулярных методов, расширили вышеприведенные списки, включив в них бактерии по меньшей мере из восьми семейств (рис. 1А).
Рис.1. Возможные определения здорового микробиома: состав, функция, динамика и экология. А) Ранние определения «здорового» микробиома обычно фокусировались на наборах таксонов, которые, как можно было бы ожидать, будут преобладать у здоровых людей. В то время как чисто таксономические ядра любого типа остаются неуловимыми даже в относительно узко определенных популяциях, каждая среда обитания в теле обладает сильным филогенетическим обогащением. Здесь показаны типичные роды (или семейства в кишечнике) в здоровых популяциях на разных участках [7, 9, 15, 33-35]. В) Метагеномные измерения позволили оценить функциональный потенциал микробиома на различных участках. Эти исследования позволили получить более последовательно разделяемые функциональные ядра общечеловеческих и нишевых путей, которые поддерживаются в здоровом состоянии [6, 7, 9, 98]. LPS — Липополисахарид, PAMP — патоген-ассоциированный молекулярный паттерн. С) Экологические модели сборки обеспечивают еще одно возможное определение здорового микробиома, поскольку каждый хозяин может черпать из «типичной» метапопуляции потенциальные микробы через смесь частично стохастических процессов. Эти процессы могут включать порядок, в котором микробы колонизируют свою соответствующую человеческую среду обитания (например, под влиянием географии и раннего воздействия), длительное наличие каждого микроба в местной среде обитания хозяина и отбор хозяина (с помощью диеты или генетики — адаптировано из рис. 1 [101]). D) Здоровый микробиом также может быть охарактеризован с точки зрения его динамики, изображенной здесь в упрощенной модели как концептуальный энергетический ландшафт. Младенческий микробиом (желтая точка) начинается в нестабильном состоянии и постепенно спускается к одному из нескольких потенциально здоровых состояний аттрактора взрослого человека. Возмущениям микробиома (пунктирные красные стрелки) можно либо противостоять (зеленая точка), либо вывести микробиом из здорового состояния, после чего устойчивый микробиом вернется в здоровое состояние (не обязательно исходное здоровое состояние) или впадет в нездоровое состояние (красная точка).
Хотя они менее изучены, чем кишечник, многие другие среды обитания в здоровых людях заняты микробными сообществами [7]. Состав сообществ более сходен внутри местообитаний, чем между ними (например, оральные сообщества имеют большее сходство с оральными сообществами у других людей, чем с другими местообитаниями у одного и того же человека), хотя, в свою очередь, межиндивидуальные различия внутри местообитаний гораздо больше, чем внутри-индивидуальная изменчивость во времени [7]. Оральные участки содержат особенно разнообразные микробиомы [33], сходные по сложности с микробиомом кишечника [7], и, как правило, там доминируют Streptococcus spp. [7]. Участки кожи различаются в основном местными свойствами кожи (сухая, влажная и сальная [34]) и колонизируются преимущественно Коринебактериями (Corynebacterium), кожными Пропионибактериями (Propionibacterium) и Стафилококками (Staphylococcus) [34]. Здоровое влагалище содержит одну из наиболее удивительно структурированных микробных экосистем, по крайней мере, с пятью воспроизводимыми типами сообществ или «типами состояний сообществ», в каждом из которых преобладает один вид Lactobacillus (L. crispatus, L. iners, L. jensenii или L. gasseri) или смесью других микробов, включая Gardnerella [35, 36]. Значимые детерминанты типа состояния сообщества женщины включают расу / этническую принадлежность [35, 37] и беременность [37], хотя даже в этой структурированной экосистеме внутри субъекта лонгитюдные вариации существенны и на сегодняшний день не имеют полностью объясненных причин.
Несколько значительных местообитаний тела, как правило, имеют особенно низкую микробную биомассу у здоровых людей и поэтому их труднее охарактеризовать. Легкое, например, почти стерильно при отсутствии инфекции или хронического заболевания, что приводит к большому интересу к выявлению его нормальных обитателей, но также и к существенным техническим проблемам при отборе проб и секвенировании участка [38-40]. Точно так же грудное молоко [41] и плацента [42] представляют интерес для раннего установления как здорового микробиома, так и потенциальных циркулирующих микробиомов крови [43] или тканей [44, 45] для нормального иммунного контроля оппортунистов. Существуют значительные трудности в получении метагеномов из таких сред, и поэтому большинство исследований опираются на обследования чувствительных к загрязнению ампликонов [46] и относительно низкопроизводительные одноэлементные методы, такие как FISH или микрофлюидика. Таким образом, необходимы более масштабные, тщательно контролируемые исследования, чтобы установить функциональность этих сложных микробных мест обитания с низкой плотностью.
Археи, вирусы, грибы и другие эукариоты
Изучение здорового микробиома было значительно обогащено для бактерий [7, 9], при этом меньшее внимание уделялось другим микробным доменам. Однако человеческий микробиом широко охватывает древо жизни и, таким образом, включает археи, вирусы и эукариоты (например, дрожжи). В микробиоме здорового человека, прежде всего в кишечнике, было обнаружено небольшое количество архейских родов. Виды рода Methanobrevibacter являются наиболее распространенными [47] в кишечнике, при этом их статус как «здоровых» членов сообществ других участков тела остается несколько неясным [48]. В частности, было обнаружено, что Methanobrevibacter smithii хорошо адаптируется к кишечнику человека, оптимизируя переваривание пищевых полисахаридов другими микробами [49] и адаптируя экспрессию своих генов в присутствии обычных кишечных бактерий, таких как Bacteroides thetaiotaomicron [49]. Человеческий виром особенно обширен и, хотя и недостаточно охарактеризован, признан неотъемлемой частью здоровой экосистемы человека [50]. При гипервариабельной природе вирусов каждый человек, как ожидается, будет иметь уникальный Виром [51, 52], состоящий в основном из бактериофагов [50] (по оценкам, 5% генного комплемента кишечных бактерий кодирует профаговые белки [9]). Фаги также обеспечивают дополнительное средство горизонтального переноса генов среди других отдаленно родственных бактерий [53]. Поскольку методы молекулярного профилирования для архей, вирусов и эукариот все еще менее развиты, чем для бактерий (даже те, которые используют культурально-независимые подходы [47, 54]), информация о молекулярной функциональности этих организмов в сообществах in situ остается ограниченной.
Хотя наиболее известные эукариотические микроорганизмы, обнаруженные в организме человека или на его поверхности (главным образом грибы и протисты), как правило, являются патогенами, важно помнить, что многие такие эукариоты, в частности Кандида (Candida), Малассезия (Malassezia) и Сахаромицеты (Saccharomyces), широко распространены даже в здоровых популяциях [55-58]. Взаимодействия между царствами несут ответственность по крайней мере за часть экологического и иммунного баланса здорового микробиома; например, существует очевидная конкуренция между бактериями и грибами в биохимических средах кожи [59] или в контроле лактобацилл над грибами в кишечнике [55] и влагалище [60]. Хотя существует мало примеров, были обнаружены прямые мутуалистические отношения между людьми и грибами, из которых наиболее характерными являются дрожжи Saccharomyces boulardii, первоначально выделенные для борьбы с холерой [61]. Некоторые простейшие являются даже обычными обитателями здоровых микробиомов [58, 62], хотя (как и вирусы) с еще большей межличностной изменчивостью, чем бактерии [58]. Кроме того, наличие некоторых простейших, таких как Blastocystis, было связано со снижением риска развития желудочно-кишечных заболеваний [63]. Наконец, хотя многоклеточные эукариоты, такие как гельминты, как правило, были исключены из микробиома кишечника в западных культурах, они были компонентом микробиома кишечника на протяжении значительной части нашей недавней эволюционной истории [64]. Учитывая их мощные иммуномодулирующие возможности и взаимодействие с другими обитателями нормального микробиома кишечника (такими как лактобациллы [65]), их элиминация, возможно, удалила важного воспитателя нашей иммунной системы [64].
Географическая изменчивость в здоровом микробиоме
Исследования, сопоставляющие микробиомы кишечника из разных стран, выявили систематические различия в микробном составе, хотя по-прежнему трудно отделить межпопуляционные технические эффекты от межпопуляционной биологии. Сравнение между крупнейшими когортами из трех континентов — MetaHIT (европейская), HMP (американская) и китайская диабетическая когорта (Chinese diabetes cohorts) — показало, что межстрановые (географические) различия в таксономическом составе значительно превышали межличностные различия, что объясняется не только техническими различиями в экспериментальных методологиях [29]. Тем не менее, небольшие международные исследования также определили географию как один из основных источников крупномасштабных изменений в микробиоме, в том числе между Северной и Южной Америкой [66], Европой и Африкой [67], Кореей и Японией [68], а также между сельским и городским населением России [69] и Китая [70]. Среди возможных движущих сил этой вариации в качестве важного фактора была предложена диета [67], наряду с другими факторами, включая географию, воздействие на ранних этапах жизни и генетику [29, 71]. Ни одно исследование до сих пор не показало, что какой-либо из этих факторов является причинным в больших наблюдаемых межпопуляционных различиях в здоровых микробиомах [72].
Географические различия на уровне штаммов также представляют интерес, особенно в связи с тем, что сигнатуры штаммов проявляют большую временную стабильность, чем профили микробного изобилия [8, 73, 74]. Исследования в этой области носят предварительный характер, но показывают, что различия между штаммами не особенно выражены между странами или континентами. Такие виды, как Bacteroides coprocola и Prevotella copri, демонстрируют наибольшие различия [73] и варианты на уровне штаммов в генах устойчивости к антибиотикам, охватывающих популяции [75]. Микробиологическая экспертиза на уровне штаммов высоко наследуемых видов, таких как Helicobacter pylori, была удивительно проницательной в отслеживании исторических эффектов на микробиом [76, 77], и в будущем для тщательных крупномасштабных популяционных обследований следует использовать культурально-независимые методы.
Установление микробиома и ранняя колонизация
Факторы, влияющие на динамику микробиома в раннем возрасте, являются важными преципитаторами здорового микробиома. Интродукция и персистенция микроорганизмов — это полустохастический процесс, на который влияют многие элементы (Рис.1 С), давая здоровую взрослую конфигурацию только после первых нескольких лет жизни [66, 78-80]. Обогащение микробиома кишечника ребенка такими симбионтами, как Bacteroides, Parabacteroides, Clostridium, Lactobacillus, Bifidobacterium и Faecalibacterium prausnitzii, обеспечивает несколько детерминант здорового микробиома. Как только они установлены, они являются основными продуцентами короткоцепочечных жирных кислот (SCFAs), важного источника энергии из неперевариваемых углеводов [81]. SCFAs являются иммуномодулирующими [82], подавляют распространенные патогены, и, предположительно, обладают опухоль-супрессорными свойствами [83, 84]. Микробиом кишечника является неотъемлемым требованием для образования иммунной системы, и обеспечение образования полезных микробных родов в раннем возрасте способствует иммунной толерантности и, следовательно, может ослабить или отменить аутоиммунные заболевания [1, 85-89].
Способ родоразрешения может влиять на формирование микробиоты в раннем возрасте, так что кесарево сечение связано с обогащением у оппортунистов, включая Haemophilus spp., Enterobacter cancerogenus / E. hormaechei, Veillonella dispar / V. parvula [78] и Staphylococcus [80]. Эти микробы продолжают сохраняться, по крайней мере, в течение первого года жизни [78] и, возможно, способствуют бремени инфекций у детей. Диета также оказывает сильное селективное давление на микробиом [71, 90], и грудное вскармливание (как первая диета) благоприятствует определенным микробным группам из числа исходной микробиоты, которые могут собираться случайным образом. Например, олигосахариды человеческого молока (HMO) могут быть использованы в качестве единственного источника углерода лишь несколькими видами Bifidobacterium и Bacteroides [91], и, более того, недавно было показано, что олигосахариды коровьего молока (BMO) способствуют росту и метаболизму в микробиото-зависимом порядке в моделях младенцев на животных [92]. Хотя эти модели не могут быть непосредственно применены к человеческим младенцам из-за уникального структурного разнообразия, сложности и высокой концентрации HMO [93, 94], они дают дополнительную поддержку выводу о том, что долгосрочные преимущества грудного вскармливания [95] частично опосредованы микробиомом.
Отличительные признаки здоровья
Функциональное ядро
В то время как большие межличностные различия наблюдаются в таксономическом составе микробиома на всех участках, обилие метаболических путей значительно более устойчиво у людей для данных участков [7, 9, 26, 27]. Кроме того, хотя состав микробиома резко меняется в течение первых лет жизни, этот функциональный профиль устанавливается на ранней стадии и после этого остается стабильным, по крайней мере, в кишечнике [72]. Это говорит о том, что одно определение «основного» здорового микробиома может включать комбинации специфических семейств микробных генов, метаболические модули и регуляторные пути, которые вместе способствуют устойчивой экологии, связанной с хозяином [96, 97]. В это ядро входят функции как минимум из трех групп: во-первых и, самое простое, вспомогательные функции, необходимые для всей микробной жизни, такие как транскрипция и трансляция, производство энергии и структурные компоненты [6, 7, 9]. Во-вторых, это ядро включает процессы, специфичные для связанных с человеком микробиомов в местах обитания на участках тела, такие как адгезия к поверхностям клеток-хозяев и производство соединений, участвующих во взаимодействии хозяина-микроба (включая необходимые витамины, такие как витамин К, и иммуностимуляторные соединения) [6, 7]. Наконец, разные среды обитания тела имеют свои специализированные основные функции [98]. Например, в кишечнике основные функции включают биодеградацию гликозаминогликана, выработку короткоцепочечных жирных кислот, обогащение специфическими липополисахаридами и выработку витаминов и незаменимых аминокислот [6, 9, 98, 99] (рис. 1 b). Какая из этих функций, как правило, обогащается в данной популяции, может зависеть от долгосрочного избирательного давления, такого как диета [67]. Поэтому необходимым условием для здорового микробиома является наличие множества видов микроорганизмов, которые могут выполнять определенные наборы биомолекулярных функций в каждой из специфических для ниши биохимических сред в организме.
Здоровая экология микробного сообщества
Если микробные сообщества собираются на основе их охвата основным набором функций при отборе из большой метапопуляции потенциальных колонизаторов, то они, вероятно, будут экологически разнообразны [100-102], как с точки зрения богатства (количество присутствующих таксонов), так и с точки зрения равномерности (обилие многих микробных составляющих). Высокое разнообразие обычно ассоциируется со здоровьем [11] и временной стабильностью [103]. Последнее может, например, быть результатом повышенной функциональной избыточности, которая приходит с более разнообразным набором микробов, даже если функциональный потенциал сборки минимально достижим с меньшим количеством таксонов. И наоборот, относительное отсутствие разнообразия проявляется в микробиоме кишечника при заболеваниях, варьирующих от ожирения [26] до воспалительных заболеваний кишечника [104] и диабета 1-го типа [72] и 2-го типа [28]; а также в микробиоме кожи при атопическом дерматите [105] и псориазе [106]. Антибиотики также вызывают резкое снижение разнообразия микробиома с сильно изменяющейся динамикой восстановления [107], потенциально ослабляя способность сообщества исключать патогенные микроорганизмы. Это может расчистить путь для заражения патобионтами — нормальными членами микробного сообщества, которые становятся вредными при возмущении, такими как Candida albicans [57]. Принцип, согласно которому большое разнообразие является «здоровым», не распространяется на все участки тела, так как разнообразие во влагалищном микробиоме может быть связано с бактериальным вагинозом [108], интраэпителиальной неоплазией шейки матки [109] (аномальный рост шейки матки), преждевременными родами [36] и воспалением [110].
Учитывая типичное наблюдение увеличения разнообразия микробиомов в состоянии здоровья, было выдвинуто предположение [111], что последовательное снижение микробного разнообразия кишечника в развитых странах может объяснять более высокие показатели хронических заболеваний по сравнению с теми, которые наблюдаются в развивающихся странах и примитивных обществах [66, 112, 113], что получило название «гипотеза исчезающего микробиома» [111]. Эта потеря разнообразия может быть связана с диетой с высоким содержанием жиров, рафинированного сахара и низким содержанием клетчатки [114]. Гуманизированные мыши на такой диете демонстрируют истощение микробного разнообразия [114], и хотя это можно восстановить, вернувшись к диете с высоким содержанием клетчатки в течение одного поколения, оно (истощение) становится фиксированным после четырех поколений [114]. Если этот результат обобщается на человеческие популяции, то это повышает актуальность разработки рационально направленных методов поддержания и лечения микробиома, с тем чтобы направить менее здоровые микробиомы в сторону более естественных сообществ. Гипотеза об исчезающем микробиоме в некотором смысле представляет собой эволюцию гипотез «гигиены» или «старых друзей» [115], все из которых предполагают, что в то время как современные североамериканские или европейские когорты могут представлять «здоровые» микробиомы, их отношение к тому, что эволюционно «нормально», может быть более сложным.
Сопротивление, устойчивость и стабильность
Другими отличительными признаками здоровья с точки зрения микробной экологии являются способность противостоять возмущению (которое может возникнуть в результате проникновения патогена, изменения рациона питания или приема лекарств) и впоследствии вернуться к здоровому состоянию. Эти свойства были названы сопротивлением и устойчивостью соответственно [2]. Например, после лечения антибиотиками здоровые кишечные сообщества обычно восстанавливаются до своего прежнего состояния через несколько недель-месяцев [116]. Таким образом, недавнее определение микробного здоровья явно включает в себя не одно статическое состояние, а скорее динамическое равновесие [2]. С этой точки зрения здоровый микробиом соответствует аттрактору лежащей в его основе динамической системы (рис. 1 d), по аналогии с судьбой клеток в многоклеточных животных [117]. Аттракторы захватывают как сопротивление, так и устойчивость, в том смысле, что система будет сопротивляться отходу от аттрактора, и если флуктуация (которая может быть вызвана внешним возмущением или внутренней стохастичностью) достаточно велика, она будет стремиться вернуться в область устойчивого состояния [117]. Наиболее заметными примерами в микробиоме человека могут быть переходы между типами состояния сообщества в здоровом влагалище; хотя их конкретные последствия для здоровья еще не перечислены, не все типы состояния сообщества имеют одинаковую степень стабильности [36]. Кишечный микробиом также находится в постоянном движении, приобретая и теряя виды с течением времени, причем различные таксоны имеют разную стабильность и некоторые из них последовательно остаются в кишечнике в течение многих лет [8]. Механизмы, с помощью которых сохраняются конкретные таксоны, еще не вполне четко определены, но интересно предположить, могут ли такие механизмы иметь отношение к движущим принципам сборки микробиома. Если конкретные сообщества собираются в основном для заполнения набора подходящих для среды обитания функциональных ниш [6], то виды, которые обеспечивают ключевые метаболические, сигнальные, иммуномодулирующие или другие роли в конкретном собрании, могут быть более устойчивыми во времени, чем те, которые находятся на функциональной периферии. Таким образом, связь динамики с таксономическим разнообразием и огромным молекулярно-функциональным потенциалом микробиома является напоминанием о сложности микробиома человека и, как следствие, о трудностях определения даже кажущейся простой концепции микробного здоровья.
Прогноз
Эпоха эпидемиологии целых микробиомов в масштабах всего населения началась совсем недавно, когда HMP [7, 118] и MetaHIT [9, 29] были в числе первых крупных когорт, включивших широкие справочные данные в области здравоохранения, и вскоре появятся еще несколько когорт. На сегодняшний день преобладают данные кросс-секционных исследований западных популяций на основе ампликона, все из которых являются эффективными и доступными, но которые еще не дают последовательной, всеобъемлющей картины глобального, динамичного, здорового микробиома. Крупномасштабная эпидемиология в других областях человеческого здоровья, таких как питание и образ жизни, создала прочную основу для перспективных, длительных когорт, кропотливых анализов и тщательно проверенных инструментов измерения [119-121], все из которых представляют собой особенно перспективные направления исследования микробиома. Вложенные исследования продольных данных микробиомов в существующих когортах имеют то преимущество, что они используют долгосрочную собранную информацию о образе жизни, рационе питания, медицинскую информацию и фенотип, а также интеграцию с накопленными биопробами. Примером нетрадиционного крупномасштабного исследования, отличающегося своей инфраструктурой и охватом, является американский проект American Gut project: краудфандинговый источник справочных данных по микробиому в сочетании с тематическими экологическими метаданными. Перспективные исследования с детальными молекулярными данными, хотя и более дорогостоящие и сложные с точки зрения логистики, также будут необходимы для облегчения прогностических моделей и установления причинно-следственной связи дисбиозов. Текущий проект «HMP2» или Интегративный проект микробиома человека (iHMP) [122] включает в себя три таких продольных исследования, которые предоставляют мульти-омические данные для здоровья и хронических заболеваний, а также протоколы и вычислительные инструменты в качестве основы для будущей работы.
В то время как многие современные исследования микробиома сосредоточены на болезнях, более глубокое понимание здорового микробиома само по себе поможет разработать новые методы диагностики и терапии микробного сообщества [123]. В той мере, в какой можно определить универсальные признаки здорового микробиома, их отсутствие может быть предиктором возникновения заболевания в целом, так же как и наличие признаков, характерных для дисбактериоза какого-либо одного состояния (особенно полезно, если это происходит до начала заболевания). С другой стороны, персонализированная медицина и лонгитюдный мониторинг могут служить той же цели в отношении отклонения от собственного «здорового» состояния индивида [1, 104]. С терапевтической точки зрения, поскольку целенаправленные вмешательства разрабатываются для манипулирования микробиомом, лечение дисбактериоза не направлено для возвращения в исходное состояние, из которого человек вышел (из-за возмущения, такого как лечение антибиотиками или вторжение патогена), а, по возможности, только в здоровое состояние (рис. 1d). Точно так же, даже если микробный дисбактериоз окажется скорее ответным, чем причинным в любом конкретном болезненном состоянии, возвращение к «здоровому» состоянию все равно может принести терапевтическую пользу [73, 101, 124].
Один из самых больших выдающихся пробелов в понимании основной биологии «здорового» микробиома, возможно, находится на уровне аннотирования его молекулярной функции: до 50% семейств микробных генов, встречающихся в человеческом микробиоме, остаются функционально нехарактерными даже в хорошо изученных средах, таких как кишечник [9, 25, 29]. Это в определенной степени справедливо и для отдельных геномов микробных изолятов, где даже хорошо изученная E. coli K12 содержит около 18% генных продуктов без какой-либо заявленной функции [125], причем значительно больше на уровне пангенома видов E. coli [126]. Вполне вероятно, например, что некоторые из этих генов ответственны за взаимодействие микроба с микробом или хозяина с микробом и, таким образом, будут экспрессироваться или характеризоваться только в условиях сообщества. Популяционные исследования микробиома сами по себе могут быть использованы для частичного смягчения этой ситуации, в том смысле, что микробные семейства генов, которые широко распространены и многочисленны, но еще недостаточно хорошо изучены, могут быть приоритетными для характеристики. Кроме того, микробные сообщества обеспечивают новый источник информации о виновности по ассоциации, который может быть использован вычислительно для получения прогнозов функции генов [127, 128]. Тем не менее, возвращение к микробиологическим корням в конечном итоге может оказаться наиболее важным в этой области: лучшие биохимические характеристики все еще основаны на физиологии культуры, микробном метаболизме, совместном культивировании и взаимодействиях, а также на контролируемой лабораторной среде в сочетании с высокопроизводительными молекулярными анализами [15, 129, 130].
Исследования микробиома, как в области здоровья, так и в области болезней, должны и впредь обеспечивать интеграцию эпидемиологии популяционного масштаба с узкими, но глубокими клиническими исследованиями в контексте персонализированной медицины. В обоих случаях исследования микробиома всего организма можно рассматривать как расширение микробных методов, уже используемых для наблюдения за инфекционными заболеваниями [131]: вместо того чтобы ждать, чтобы контролировать вспышку патогена в популяции или его персистентность в пределах отдельного человека, наше полное микробное сообщество можно было бы контролировать для поддержания здоровья или ухода в болезни. Это в равной степени относится и к интеграции активности микробиома с иммунным, транскрипционным, эпигенетическим и клиническим состоянием хозяина: точная медицина микробного сообщества должна опираться на взаимодействие хозяина и микробиома в качестве ключевого компонента. Это поможет быстро выявить потенциальные патогенные микроорганизмы [132] и позволит определить «правильные» меры вмешательства для восстановления здоровья после дисбактериоза, начиная от изменения рациона питания или образа жизни через пробиотики и заканчивая микробно-целевыми фармацевтическими препаратами [133]. Таким образом, более глубокое понимание здорового микробиома должно подходить к нему как к одному из аспектов глубоко контролируемого персонализированного здоровья (например, [121]) и должно интегрировать популяционную оценку микробного сообщества с хорошо охарактеризованным молекулярным пониманием и анализом того, как благоприятные состояния микробного сообщества поддерживаются в масштабах всего организма и на протяжении всей жизни.
- Литература к разделу «Здоровый микробиом человека»
О структуре микробиома здорового человека
По материалам Консорциума HMP (The Human Microbiome Project Consortium). Structure, function and diversity of the healthy human microbiome. Nature. 2012; 486: 207–14)
Исследования микробиома человека показали, что даже здоровые люди заметно отличаются по микробам, которые занимают такие места обитания, как кишечник, кожа и влагалище. Большая часть этого разнообразия остается необъяснимой, хотя причастны диета, окружающая среда, генетика хозяина и раннее воздействие микробов. Соответственно, чтобы охарактеризовать экологию микробных сообществ, связанных с человеком, проект «Микробиом человека» или HMP (Human Microbiome Project) проанализировал самую большую когорту и набор отдельных клинически значимых сред обитания на сегодняшний день. Исследователи обнаружили, что разнообразие и обилие сигнатурных микроорганизмов в каждой среде обитания широко варьируются даже среди здоровых людей, с сильной специализацией ниши как внутри, так и среди людей. Проект столкнулся с примерно 81-99% родов, семей ферментов и конфигураций сообществ, занятых здоровым западным микробиомом. Метагеномный перенос метаболических путей был стабильным среди индивидуумов, несмотря на различия в структуре сообщества, а этнический/расовый фон оказался одной из самых сильных ассоциаций как путей, так и микробов с клиническими метаданными. Таким образом, эти результаты очерчивают диапазон структурных и функциональных конфигураций, нормальных в микробных сообществах здоровой популяции, позволяя в будущем характеризовать эпидемиологию, экологию и трансляционные приложения микробиома человека.
Обильные таксоны в микробиоме человека, которые были метагеномно и таксономически четко определены в популяции HMP
Рис. 2. Бактериальные таксоны в здоровом микробиоме. А-С) Распространенность (интенсивность, цвет, обозначающий тип / класс) и изобилие (наличие и размер ветвей) таксонов в здоровом микробиоме. Наиболее распространенные: A) метагеномно-идентифицированные, B) PATRIC «патогены» (метагеномные) и C) 16S-идентифицированные роды. Размер популяции и глубина секвенирования HMP хорошо определили микробиом на всех анализируемых участках тела, что оценивалось по насыщенности добавленного сообщества.
Бактериальные функции важные для жизни в кишечнике
Обширный неизрасходованный каталог бактериальных генов из кишечного тракта человека дает возможность выявить бактериальные функции, важные для жизни в этой среде. Существуют функции, необходимые бактерии для процветания в кишечном контексте (т.е. «минимальный геном кишечника») и те, которые участвуют в гомеостазе всей экосистемы, закодированные во многих видах («минимальный метагеном кишечника»). Первый набор функций, как ожидается, будет присутствовать в большинстве или всех видах бактерий кишечника, второй набор — в большинстве или всех образцах кишечника человека.
Для определения функций, кодируемых минимальным геномом кишечника, ученые использовали тот факт, что эти функции должны присутствовать в большинстве или во всех видах бактерий кишечника и поэтому появляются в генном каталоге с частотой, превышающей частоту функций, присутствующих только в некоторых видах бактерий кишечника. Относительная частота различных функций может быть выведена из числа генов, набранных в различные кластеры Базы данных биологической информации eggNOG, после нормализации для длины гена и числа копий. Исследователи ранжировали все кластеры по частотам генов и определили диапазон, который включал кластеры, определяющие хорошо известные существенные бактериальные функции, такие как те, которые определены экспериментально для хорошо изученного представителя Фирмикутов, Bacillus subtilis, предполагая, что дополнительные кластеры в этом диапазоне одинаково важны. Как и ожидалось, диапазон, который включал большинство основных кластеров B. subtilis (86%), находился в самом верху порядка ранжирования. Около 76% кластеров с эссенциальными генами Escherichia coli находились в пределах этого диапазона, подтверждая обоснованность подхода авторов. Это говорит о том, что 1244 метагеномных кластера, обнаруженных в пределах диапазона, определяют функции, важные для жизни в кишечнике.
Ученые обнаружили два типа функций среди кластеров диапазона: те, которые требуются для всех бактерий («ведение домашнего хозяйства»), и те, которые потенциально специфичны для кишечника. Среди многих примеров первой категории являются функции, которые являются частью основных метаболических путей (например, центральный метаболизм углерода, синтез аминокислот), а также важных белковых комплексов (РНК-полимеразы, ДНК-полимеразы, АТФ-синтазы, общего секреторного аппарата).
Рис. 3. Функциональный состав минимального кишечного генома и метагенома (функции минимального генома и метагенома: редк. — редкие, част. — частые).
Предполагаемые кишечно-специфические функции включают те, которые участвуют в адгезии к белкам хозяина (коллаген, фибриноген, фибронектин) или в сборке сахаров глобо-серий гликолипидов, которые «декорируют» клетки крови и эпителиальные клетки. Кроме того, 15% кластеров диапазона кодируют функции, которые присутствуют в <10% геномов Базы данных eggNOG и в значительной степени (74,3%) не определены (рис. 3). Детальное их изучение должно привести к более глубокому пониманию бактериальной жизни в кишечнике.
Содержание и разнообразие микробного сообщества кишечника
(по результатам 16S секвенирования и метагномного секвенирования от авторов проекта MetaHIT)
Прим. ред.: Проект метагенома кишечного тракта человека MetaHIT является 7-м Рамочным проектом Европейской комиссии, осуществляемым консорциумом из 13 учреждений из Европы и Китая. Он фокусируется на микроорганизмах кишечника, которые особенно многочисленны и сложны и играют важную роль для здоровья и благополучия человека. MetaHIT реализует и объединяет следующие виды деятельности:
- Создание эталонного набора генов и геномов кишечных микробов с использованием высокоточного метагеномного секвенирования и полного секвенирования бактериального генома;
- Создание универсальных инструментов, основанных на массивах ДНК высокой плотности и высокопроизводительных методах секвенирования ДНК, для изучения вариабельности микробиоты кишечника человека;
- Использование инструментов для поиска ассоциаций между генами, присутствующими в микробиоте кишечника, и болезнями, уделяя особое внимание воспалительным заболеваниям кишечника и ожирению;
- Изучение генов, связанных с заболеванием, с точки зрения их функции в микробах и их влияния на хозяина, с акцентом на взаимодействие хозяин-микроб;
- Разработка информационного ресурса для хранения, систематизации и анализа разнородной информации, относящейся к микробиоте кишечника человека и ее влиянию на здоровье;
- Создание интерфейса с заинтересованными сторонами и широкой общественностью для содействия сотрудничеству и координации в области метагенома человека.
Было подсчитано, что микробы в нашем организме в совокупности составляют до 100 триллионов клеток, что в десять раз превышает число человеческих клеток, и предположено, что они кодируют в 100 раз больше уникальных генов, чем наш собственный геном. Большинство микробов обитает в кишечнике, оказывают глубокое влияние на физиологию человека и питание и имеют решающее значение для жизни человека. Кроме того, кишечные микробы способствуют получению энергии из пищи, а изменения микробиома кишечника могут быть связаны с заболеваниями кишечника или ожирением.
А) 16S секвенирование
По материалам David A. Relman, et. al. Diversity of the Human Intestinal Microbial Flora. Science. 2005 Jun 10; 308(5728): 1635–1638.
Чтобы понять и использовать влияние кишечных микробов на здоровье и благополучие человека, необходимо расшифровать содержание, разнообразие и функционирование микробного сообщества кишечника. Методы, основанные на секвенировании генов 16S рибосомных РНК (16S рРНК), показали, что два бактериальных отдела, Бактероидеты и Фирмикуты (Bacteroidetes & Firmicutes), составляют более 90% известных филогенетических категорий и доминируют в микробиоте дистального отдела кишечника. В соответствующем исследовании ученые сфокусировались на 16S рДНК, учитывая ее универсальное распределение среди всех прокариот и надежность для определения филогенетических связей. Образцы слизистой оболочки и фекалии были получены от трех здоровых взрослых субъектов (А, B, С), которые были частью более крупного популяционного исследования «случай-контроль». Образцы слизистой оболочки были получены во время колоноскопии из здоровых участков в шести основных отделах толстой кишки человека: слепая кишка, восходящая толстая кишка, поперечная толстая кишка, нисходящая толстая кишка, сигмовидная кишка и прямая кишка. Образцы фекалий отбирали у каждого субъекта также через 1 месяц после колоноскопии (итого было взято по 7 образцов от каждого субъекта). Исследователи, как было сказано выше, сфокусировались на 16S рДНК. 16S рДНК амплифицировали из образцов с полимеразной цепной реакцией (ПЦР) и бактериальными и архейными праймерами широкого спектра действия. Семь образцов от субъекта B и образец фекалий от субъекта C дали археальные продукты; Все 21 образец дал бактериальные продукты. Продукты ПЦР были клонированы и секвенированы в двух направлениях, и были применены численные экологические подходы.
Из 395 бактериальных филотипов 244 (62%) были новыми, а 80% представляли последовательности из видов, которые не культивировались. Большинство предполагаемых организмов были членами Firmicutes и Bacteroidetes, что согласуется с другими молекулярными анализами кишечной флоры (1, 2, 4). Тип Firmicutes состоял из 301 филотипа, 191 из которых были новыми; большинство (95%) последовательностей Firmicutes были членами класса Clostridia. Исследователи обнаружили значительное количество Firmicutes, связанных с известными бутират-продуцирующими бактериями (2454 последовательности, 42 филотипа), все из которых являются членами кластеров клостридий IV, XIVa и XVI. Мы ожидали заметного представления этой функциональной группы среди наших здоровых контрольных субъектов, учитывая ее роль в поддержании и защите нормального эпителия толстой кишки. Большие различия между 65 филотипами Bacteroidetes были отмечены между субъектами, как описано ранее (1, 2). B. thetaiotaomicron был обнаружен у каждого субъекта и данный вид, как известно, участвует в полезных функциях, включая всасывание питательных веществ и созревание и поддержание эпителиальных клеток. Относительно небольшое количество последовательностей было связано с типами Proteobacteria, Actinobacteria, Fusobacteria и Verrucomicrobia. Низкое содержание последовательностей протеобактерий (включая Escherichia coli) не было неожиданным, учитывая, что факультативные виды могут представлять около 0,1% бактерий в строгой анаэробной среде толстой кишки; это согласуется с предыдущими результатами (1, 3, 4).
Б) Метагеномное секвенирование
По материалам Qin J, Li R, Raes J, Arumugam M, Burgdorf KS, Manichanh C, et al. A human gut microbial gene catalogue established by metagenomic sequencing. Nature. 2010;464:59–65.

Общее бактериальное ядро

Глубокое метагеномное секвенирование дает возможность исследовать существование общего набора видов микробов (общего ядра) в когорте. Для этой цели авторы использовали нередуцированный набор из 650 секвенированных бактериальных и археальных геномов. Они выровняли показания Illumina GA каждого микробного образца кишечника человека по набору геномов, используя 90%-ный порог идентичности, и определили долю геномов, охваченных чтениями, которые выровнены только по одной позиции в наборе. При охвате 1%, который для типичного кишечного бактериального генома соответствует средней длине около 40 кб, что примерно в 25 раз больше, чем у гена 16S, обычно используемого для идентификации видов, исследователи обнаружили 18 видов у всех идивидуумов, 57 в ≥90% и 75% ≥50% людей. При 10% охвате, требующем ~ 10-кратного увеличения численности в выборке, ученые все же обнаружили 13 из вышеуказанных видов у ≥90% индивидуумов и 35 у ≥50%.
Когда накопленная длина последовательности увеличилась с 3,96 Гб до 8,74 Гб и с 4,41 Гб до 11,6 Гб для используемых образцов MH0006 и MH0012 (156,9 Мб и 154,7 Мб), соответственно, число штаммов, общих для двух образцов при пороге охвата 1%, увеличилось на 25%, с 135 до 169. Это говорит о существовании значительно большего общего ядра, чем то, которое мы могли бы наблюдать на глубине последовательности, обычно используемой для каждого человека.
Изменчивость численности микробных видов у индивидуумов может сильно повлиять на идентификацию общего ядра. Чтобы визуализировать эту изменчивость, исследователи сравнили количество считываний секвенирования, выровненных по разным геномам у людей из их когорты MetaHIT. Даже для наиболее распространенных 57 видов, присутствующих у ≥90% индивидуумов с охватом генома >1%, межвидовая изменчивость варьировала от 12- до 2187 кратной (рис. 4.). Как и ожидалось, Bacteroidetes и Firmicutes имели наибольшую численность.
Дополнительный материал
БАКТЕРОИДЕТЫ, ФИРМИКУТЫ и ДРУГИЕ




Пища для кишечной микробиоты
Питание и конфигурация кишечного микробиома
Со времени возрождения микробиомных исследований в последнее десятилетие накопилось много знаний о силах, формирующих архитектуру и функциональность резидентных микроорганизмов в кишечнике человека. Из множества вовлеченных эндогенных и экзогенных факторов, связанных с хозяином, основным детерминантом структуры и функционирования сообщества микробиоты кишечника является питание. Вводя пищевые сигналы в связь между хозяином и его микробиотой, питание поддерживает гомеостаз или способствует восприимчивости к болезням. Микробиота модулирует патогенез, прогрессирование и лечение заболеваний, начиная от метаболических нарушений до неврологических патологий. Изменение формы взаимодействия хозяина и микробиоты посредством персонифицированного питания является новым терапевтическим направлением как для борьбы с болезнями, так и для их профилактики. Состав и функция микробиоты кишечника формируется с младенчества, и в дальнейшем на нее оказывают сильное влияние многие факторы окружающей среды, из которых диета — является ключевым детерминантом конфигурации микробиоты, посредством модуляции обилия конкретных видов и их индивидуальных или коллективных функций. Кроме того, воздействие конкретного рациона питания на отдельных людей в популяции отличается от человека к человеку и может зависеть от сочетания особенностей хозяина и микробиома, причем последнее влияние в основном определяется окружающей средой, а не генетическим фоном, и поэтому потенциально более поддается вмешательству. Понимание того, как диетические питательные вещества модулируют микробиом кишечника, представляет большой интерес для разработки пищевых продуктов и моделей питания для борьбы с глобальным бременем неинфекционных заболеваний. Современные исследования показывают, что пищевые волокна, включая арабиноксиланы, галактоолигосахариды, инулин и олигофруктозу, способствуют развитию ряда полезных бактерий и подавляют потенциально вредные виды. Доклинические данные свидетельствуют о том, что количество и тип жира модулируют как полезные, так и потенциально вредные микробы, а также соотношение Firmicutes / Bacteroidetes в кишечнике. Клинические и доклинические исследования показывают, что тип и количество белков в рационе также оказывает существенное и дифференцированное влияние на микробиоту кишечника. То же относится к витаминам, микроэлементам, минералам, полифенолам, пищевым добавкам и т.д.
О важности оси «диета-микробиом-хозяин» см. по ссылкам:
- Прецизионное питание и микробиом: Часть 1 и Часть 2
- Диета-взаимодействие микробиоты и персонализированное питание
- Питание, здоровье и микробиота кишечника
- Влияние диеты на эволюцию микробиоты и здоровье человека
- Роль диетических питательных веществ в модуляции микробиоты
- Кето диета и микробиом
- Взаимодействие микробиома и диеты: концепция гологенома
Из истории изучения микробиома
Первые результаты проекта HMP
Human Microbiome Project
Прим. ред.: рамках Проекта человеческого микробиома (HMP) были разработаны исследовательские ресурсы, позволяющие изучать микробные сообщества, которые живут в нашем теле и на нем, и роль, которую они играют в здоровье и болезнях человека.
Начальная цель проекта — расшифровка 900 полных геномов микробов, представленных простейшими одноклеточными животными, бактериями и археями, однако даже это количество, в конечном итоге, будет лишь малой толикой информации о всех микробах, живущих в человеческом теле, которые и составляют так называемый «МИКРОБИОМ».
Проект «Микробиом человека» является логическим развитием проекта «Геном человека», целью которого является полная расшифровка человеческого генома. Несмотря на то, что проект стартовал в 1990 году, а первый полный геном человека был опубликован в 2003-м, этот проект до сих пор не завершен, так как нерасшифрованными остаются некоторые участки человеческой ДНК.

Пищеварительный тракт – пристанище большинства наших микроскопических сожителей, именно здесь находится микробиом человека. То есть кишечный микробиом – это то, что прежде называлось микрофлорой кишечника.
Сейчас, с началом масштабных геномных исследований самых разных бактериальных сообществ (например, некоторых участков дна океанов, сточных вод и пр.), название микробиом стало более популярным. Оно подразумевает совокупность не столько самих микробов, сколько всех микробных генов, оказывающих влияние на среду, в которой они существуют. Человеческий организм – это тоже среда обитания микробов…
НОВЫЙ ВЗГЛЯД НА БИОЛОГИЮ ЧЕЛОВЕКА
Итоги проекта «Микробиом человека»

Целью проекта было охарактеризовать все микробы, присутствующие в организме человека, для чего ученые взяли образцы тканей из 15 мест на теле 129 мужчин и из 18 мест у 113 женщин. Все добровольцы — здоровые люди в возрасте от 18 до 40 лет — предоставили по три образца слизистой с внутренней стороны щек, носа, кожи за ухом и локтевого сгиба, а также фекальные пробы.
По результатам генетического анализа биоматериала было установлено, что в человеческом организме обитает свыше 10 тысяч видов различных микробов. Как утверждают авторы исследования, такое разнообразие микробиома обеспечивает человека гораздо большим количеством генов, чем можно было представить. Так, если в геноме человека содержится 22 тысячи генов, кодирующих белки для регуляции метаболизма, микробиом добавляет еще около восьми миллионов уникальных бактериальных генов.
«Вопрос о том, как индивидуальные вариации типов бактерий среди здоровых людей влияют на возможное развитие болезней, пока остается открытым», — комментирует один из членов консорциума Энтони Фодор (Anthony Fodor).

Следующий момент, который хотят выяснить врачи — почему патогенные микроорганизмы вредят некоторым людям и одновременно не влияют на других? Какие изменения происходят в микросреде человека, которые приводят к риску различных заболеваний от инфекций синдрома раздраженного кишечника до псориаза?
Все эти и многие другие вопросы ставились перед учеными, участвующими в одном из самых масштабных проектов Национального института здоровья в США. Полученные результаты уже меняют взгляды ученных на то, каким образом люди остаются здоровыми и наоборот — болеют.
«Это совершенно новый взгляд на биологию человека и человеческие болезни, и это впечатляет», — прокомментировал доктор Филипп Тарр из Вашингтонского университета в Сент-Луисе, один из ведущих ученых в $ 173-милионном проекте, который финансируется Национальным институтом здоровья США.
«Эти бактерии не являются «пассажирами» в нашем теле, — подчеркнул Тарр, — они метаболически активные. Они, как сообщество, и мы должны считаться с ними так же, как мы считаемся с экосистемой леса или воды. И так же, как и в экосистемах окружающей среды, состав микробов на каждой части вашего тела разный. Ваша кожа, например, сравнима с тропическими лесами, а кишечник изобилует различными видами микроорганизмов, как океан.

Сначала ученые должны были собрать образцы тканей с разных участков тела человека – рта, носа, некоторых участков кожи, влагалища у женщин и с кала. Затем они отделили ДНК бактерий от ДНК человека и начали анализ сложных бактерий: лактобациллус, стрептококк, коринебактерии.
«Каждая клетка человеческого тела является «домом» для около 10 бактериальных клеток, но они насколько микроскопические, что общая масса всех микроорганизмов составляет от 1 до 3-х процентов массы тела человека», — объяснил доктор Эрик Грин из Национального Центра Исследований Человеческого Генома в Национальной Организации Здравоохранения США (NIH). Это значит, что человек весом 90 кг содержит в себе до 3-х кг бактерий.
Человеческий геном содержит около 22 000 генов. Но, по оценкам нового проекта, микробы наделяют наш организм еще большей силой, равной 8-ми миллионов генов. Гены бактерий выполняют определенную работу по отношению к нашему организму. Некоторые из них играют важную роль для здоровья и развития человеческой клетки, в которой они содержатся, как рассказал доктор Брюс Биррен, другой исследователь проекта. Гены кишечной бактерии, например, расщепляют некоторые белки и жиры. А также они вырабатывают полезные соединения, которые борются с разного рода воспалениями. И еще, ученые обнаружили, что не существует какого-то основного состава микроорганизмов, которые выполняют определенные функции, это могут делать разные сочетания бактерий.
ОТЛИЧИЕ МИКРОБИОМА СРЕДИ ЭТНИЧЕСКИХ ГРУПП
Небольшое отступление. В 2018 г. ученые обнаружили 12 конкретных типов бактерий, которые регулярно варьируются в изобилии в зависимости от этнической принадлежности. Поскольку этническая принадлежность захватывает многие факторы, начиная от диеты до генетики, трудно сказать, почему это так, сказал Эндрю Брукс (Andrew Brooks), докторант университета Вандербильта (Vanderbilt University) в Институте генетики Вандербильта, который проанализировал данные, предоставленные американским проектом Gut (American Gut Project) и проектом микробиома человека (Human Microbiome Project). Но это основа для понимания здоровых различий в микробиомах между людьми…
Содержание микробов в организме адаптируется в зависимости от местности проживания человека, его диеты и ряда других факторов. Среда микробов в вашем организме может также изменяться после приема антибиотиков, которые убивают некоторых из них. Кишечные палочки одного вида могут заменяться другими, при этом не менее эффективными.
«Каждый человек имеет разный состав бактерий в организме, при этом, они организованы так, чтобы выполнять определенные функции», — сказал Биррен. Вполне возможно, что наш образ жизни и окружающая среда стимулировала появление такого рода механизма, который работает на нас.
На данный момент проводятся исследования, которые бы показали, как отличается состав микроорганизмов у человека с определенной болезнью от здорового человека с дальнейшей целью профилактики и лечения заболеваний.
Рассмотрим, например, желудочно-кишечный сверхинфект (бактерии, устойчивые к воздействию антибиотиков, к ним относится также золотистый стафилококк), которым часто заражаются люди, пребывающие в больнице и от которого иногда умирают. Филипп Тарр (Вашингтонский университет) хочет узнать, какой состав из кишечных бактерий может предотвратить поражение ЖКТ дизбактериозом, или же уменьшить его пагубное действие и выяснить, кто из людей более подвержен заражению.
Исследователи Медицинского колледжа Бейлора сообщили, что бактерии, которые находятся во влагалище беременной женщины, имеют свойства меняться во время протекания беременности, возможно для того, чтобы создавалась наиболее благоприятная здоровая среда при рождении ребенка. Предыдущие исследования также показали, что состав бактерий у детей, которые принимались традиционными родами, отличается от бактерий детей, которые рождались с помощью кесарева сечения. Это объясняет, почему кесарево сечение повышает риск заражения ребенка некоторыми инфекциями.
«Каждая новая информация становится шокирующей для нас, потому что она показывает как много еще нужно работать над тем, чтобы понять тот мир, который существует внутри нас», — отметил специалист по инфекционным заболеваниям доктор Дэвид Рэльман (Стэнфордский университет), который написал обзор по результатам этого проекта в журнале Nature.
Например, в проекте принимали участие в основном белокожие добровольцы, живущие в округах Хьюстона и Сэнт Льюиса. Д-р Рэльман отметил, что нужно будет еще проделать большую работу над определением микробиома людей с разной расовой принадлежностью, этнического и географического происхождения. Есть также много открытых вопросов касательно того как эти микробы взаимодействуют с генетикой человека.
«Мы, по сути, совсем не осведомлены о том какие функции обеспечивает микробная экосистема, которая находится внутри нас и как она влияет на наше здоровье», — отметил Рэльман.
Второй этап проекта микробиом человека
Интегративный Проект Микробиома Человека
The Integrative HMP (iHMP) Research Network Consortium.
The Integrative Human Microbiome Project.
Nature volume569, pages641–648(2019)
Проект NIH Human Microbiome Project (HMP) осуществлялся в течение десяти лет и состоял из двух этапов, чтобы предоставить ресурсы, методы и открытия, которые связывают взаимодействие между людьми и их микробиомами с результатами, связанными со здоровьем. Недавно завершенная вторая фаза, «Интегративный Проект Микробиома человека», включала исследования динамических изменений в микробиоме и хозяине при трех условиях: беременность и преждевременные роды; воспалительные заболевания кишечника; и стрессоры, которые влияют на людей с преддиабетом. Соответствующие исследования начинают выяснять механизмы взаимодействия хозяина с микробиомом в этих условиях, предоставляют уникальные ресурсы данных (в DCC — Координационном центре данных HMP) и представляют собой парадигму для будущих мульти-омических исследований микробиома человека.
ВВЕДЕНИЕ
Хотя эпоха ОМИКИ ускорила все аспекты биологических исследований, ее влияние особенно заметно проявилось при изучении микробных сообществ и микробиома человека. За 18 лет, прошедших с момента публикации первого генома человека,исследования микробиома выросли от культуральных исследований полости рта и кишечника до молекулярных профилей микробной биохимии во всех экологических нишах человеческого организма1,2,3. Эпидемиология и модельные системы были использованы для выявления ассоциаций между изменениями в микробиоме и состояниями, варьирующими от аутизма4 до рака5,6,7, а также были выявлены микробные и иммунологические механизмы, влияющие, например, на эффективность лекарств, используемых для лечения сердечных состояний8 или выживаемость при болезни трансплантат-хозяин9.
Современные исследования микробиома человека также были источником основных биологических и трансляционных сюрпризов, обнажая убедительный диапазон новых открытий и открытых вопросов. Кажется, что каждый человек несет свой собственный, в значительной степени индивидуальный набор микробных штаммов10,11, которые приобретаются в раннем возрасте12,13,14, различаются между средами и популяциями15,16 и могут сохраняться в течение многих лет17 или подвергаться относительно быстрым переходам18. Микробное разнообразие проявляется по-разному в разных экологических нишах организма; например, в кишечнике, как правило, ожидается большее разнообразие, но это может быть связано с дисбиотическими состояниями и риском неблагоприятных событий в женском репродуктивном тракте. Микробиом может быть нарушен такими состояниями, как воспалительные заболевания кишечника и диабет, но различные состояния здоровья, связанные с микробиомами, и основы этих связей остаются неисследованными. Насколько динамичен микробиом во время таких процессов, как беременность или вирусная инфекция? Какие изменения в микробиоме представляют собой причины, а не последствия изменений в здоровье? Какие молекулярные элементы персонализированного микробиома могут отвечать за результаты в отношении здоровья, и как они интегрируются и поддерживают физиологические процессы, такие как иммунная система и обмен веществ? И какие экологические элементы определяют успех трансплантации микробиоты, и почему они успешны в лечении некоторых людей и состояний, но не других?
Проект Национального института здоровья «Микробиом человека» был одной из первых крупномасштабных инициатив, направленных на решение подмножества этих вопросов19 (рис. 1). Начатая в 2007 году первая фаза программы была направлена на определение наличия общих элементов в «здоровых» микробиомах при отсутствии явных заболеваний. Исследования как базовой взрослой популяции21,22,23, так и «демонстрационных» популяций со специфическими болезнетворными состояниями установили типичные диапазоны (для некоторых популяций) микробной принадлежности и ферментативных репертуаров по всему организму, комбинации метаболических функций, которые были либо преобладающими, либо специфичными для штамма, а также некоторые факторы хозяина (такие как раса или этническая принадлежность), определяющие эту вариацию. Исследования целевых популяций выявили экологические состояния таких ниш, как вагина24,25, кожа26,27,28, кишечник29,30,31,32,33 и многие другие (https://www.hmpdacc.org/health/projectdemos.php). Таким образом, эта первая фаза HMP (HMP1) дала множество ресурсов сообщества: нуклеотидные последовательности микроорганизмов и сообществ из большого числа изолятов, отдельных лиц и популяций (http://hmpdacc.org)34,35,36,37; протоколы для поддержки воспроизводимой выборки микробиома по всему телу и генерации данных38,39,40; и вычислительные методы для анализа микробиома и эпидемиологии41,42,43,44,45,46,47.
Рис. 1. Первая и вторая фазы проекта NIH Human Microbiome.
В рамках десятилетней программы NIH Human Microbiome Project (HMP), организованной в два этапа (HMP1 и HMP2), были разработаны эталонные последовательности, мульти-омические наборы данных, вычислительные и статистические инструменты, а также аналитические и клинические протоколы в качестве ресурсов для более широкого исследовательского сообщества. HMP1 фокусировался на характеристике микробных сообществ из многочисленных участков тела (ротовой полости, носа, влагалища, кишечника и кожи) в базовом исследовании здоровых взрослых испытуемых и включал ряд демонстрационных проектов, которые были сосредоточены на конкретных заболеваниях или расстройствах. HMP2 расширил репертуар биологических свойств, проанализированных как для хозяина, так и для микробиома в трех продольных когортных исследованиях репрезентативных микробиом-ассоциированных состояний: беременность и преждевременные роды (вагинальные микробиомы беременных женщин), воспалительные заболевания кишечника (кишечный микробиом) и преддиабет (кишечный и назальный микробиомы). Эти исследования следили за динамикой этих состояний с помощью мульти-омических анализов различных типов измерений с течением времени, включая изменения состава микробного сообщества, виромики, метаболомных профилей, экспрессии генов и белковых профилей как хозяина, так и микробиома, а также специфичных для хозяина свойств, таких как генетический, эпигеномный, антителовый и цитокиновый профили, наряду с другими специфичными для исследования особенностями. Все последовательности и мульти-омические данные, клиническая информация и инструменты из HMP1 и HMP2 размещаются в Координационном центре данных HMP (DCC) или ссылаются на общедоступные или контролируемые хранилища доступа, чтобы служить центральным ресурсом для исследовательского сообщества.
Одним из главных выводов HMP1 было то, что таксономический состав микробиома сам по себе часто не был хорошо коррелирован с фенотипом хозяина — это, как правило, лучше предсказывалось преобладающей микробной молекулярной функцией или персонализированным штамм-специфическим составом21. Это открытие послужило основой для разработки второй фазы HMP, интегративной HMP (iHMP или HMP2)48, которая была разработана для изучения взаимодействия хозяина и микробиома, включая иммунитет, метаболизм и динамическую молекулярную активность, чтобы получить более целостное представление о взаимодействии хозяина и микроба с течением времени. Эта мульти-омическая программа была направлена на расширение ресурсной базы, доступной для сообщества исследователей микробиома, чтобы начать рассматривать отношения между хозяином и микробиомом механистически и решать вопросы, представленные выше. Поэтому в проектах HMP2, направленных на борьбу с заболеваниями, было рекомендовано использовать несколько взаимодополняющих подходов, чтобы продольно оценить механизмы человеческой и микробной активности и предоставить протоколы, данные и биологические образцы для будущей работы. Эти проекты включали три исследования, которые отслеживали динамику состояния здоровья и заболеваемости людей в условиях с известным взаимодействием микробиомов, таким образом, непосредственно затрагивая важные последствия для здоровья, а также служили в качестве моделей «типичных» состояний, связанных с микробиомами, представляющих широкий интерес для исследовательского сообщества. Они включали беременность и преждевременные роды (PTB – или preterm birth); воспалительные заболевания кишечника (ВЗК); и стрессоры, которые влияют на людей с преддиабетом. Эти исследования, которые в настоящее время достигли первой стадии завершения49,50,51, в совокупности предоставляют обширную информацию и понимание не только микробной динамики, но также связанных с ними реакций организма-хозяина и микробных взаимосвязей. На сегодняшний день коллекция из более чем 20 рукописей описывает некоторые из этих результатов по адресу https://www.nature.com/collections/fiabfcjbfj, и вместе они обеспечивают богатый мульти-омический ресурс данных, который будет добываться в ходе будущей работы (http://www.ihmpdcc.org).
Микробиом влагалища, беременность и преждевременные роды
Преждевременные роды могут иметь разрушительные последствия для новорожденных, включая смерть и длительную инвалидизацию. В Соединенных Штатах примерно 10% рождений приходится на недоношенных детей52, а в странах с более низкими ресурсами этот показатель еще выше. Факторы окружающей среды, включая микробиом женского репродуктивного тракта, играют важную роль в развитии недоношенности. Примечательно, что эти факторы оказывают большее влияние на женщин африканского происхождения, которые также несут самое большое бремя преждевременных родов (PTB)53. В последние десятилетия младенческая смертность снизилась, но заболеваемость PTB не уменьшилась, и прогресс в прогнозировании индивидуального риска развития PTB застопорился. Во время беременности материнская иммунная система поддерживает тонкий баланс провоспалительных и противовоспалительных факторов55, и факторы, способствующие развитию PTB, включают нарушение толерантности матери и плода, сосудистые нарушения, стресс, недостаточность шейки матки, преждевременный разрыв плодных оболочек и амниотическую инфекцию56. Считается, что микробное проникновение в матку вызывает PTB, нарушая иммунный баланс матери, приводя к спонтанным преждевременным родам и / или высвобождению микробных продуктов (например, коллагеназ, протеаз или токсинов), которые нарушают целостность плодных оболочек и приводят к преждевременному разрыву мембран57.
Исследовательская группа Multi-Omic Microbiome Study: Pregnancy Initiative (MOMS-PI), в составе HMP2, охарактеризовала микробиомы беременных женщин, чтобы оценить их влияние на риск возникновения PTB (Рис. 2). В рамках этого проекта было прослежено 1527 женщин в течение всей беременности, и в него был вовлечен сбор 206 437 образцов, включая мазки из влагалища, ротовой полости, прямой кишки, кожи и ротовой полости матери, кровь, мочу и продукты родов, а также пуповинную кровь, меконий и первый сту, щечные, кожные и ректальные мазки. Подмножества этих образцов подвергались таксономическому анализу генов 16S рРНК, метагеномному и метатранскриптомному секвенированию, цитокиновому профилированию, липидомическому анализу и анализу бактериального генома. Команда MOMS-PI проанализировала 12 039 образцов из 597 беременностей, чтобы исследовать динамику микробиома и его взаимодействия с хозяином во время беременности, приводящую к PTB50.
Рис. 2. Микробиом влагалища и его связь с факторами хозяина во время беременности и преждевременных родов.
Проект MOMS-PI отслеживал 1527 беременностей в продольном направлении и включал сбор 206 437 биологических образцов для анализа факторов хозяина и микроорганизмов (ампликон 16S, метагеномное и метатранскриптомное секвенирование; профилирование цитокинов; метаболомика; протеомика; геномика и культура микробного изолята). Около 600 беременностей были тщательно проанализированы, чтобы оценить особенности, которые приводят к преждевременным родам; этот анализ выявил как факторы хозяина (например, цитокины), так и микробные (например, экологический и специфический штаммы) факторы. По мере того как беременность прогрессирует, с предсказуемыми изменениями в системных уровнях эстрадиола, матка и влагалищная среда претерпевают различные изменения. Матка переходит от раннего провоспалительного состояния к противовоспалительному состоянию во втором триместре, а затем обратно к провоспалительному состоянию до начала родов. Между тем, специфические изменения в микробиоме просвета влагалища могут быть связаны с преждевременными родами, возможно, через механизмы, включающие микроорганизмы, перемещающиеся из влагалища в матку. На рисунке представлен обзор продольных изменений в экосистеме слизистой оболочки влагалища и матки во время беременности.
Эти мульти-омические исследования выявили временные изменения в микробиоме влагалища, связанные с доношенной беременностью. Женщины, которые часто начинали беременность с вагинальным микробиомом большей экологической сложности, как правило, сходились к более однородному микробиому с преобладанием лактобацилл ко второму триместру58. Интересно, что эта тенденция была наиболее выражена у женщин африканского происхождения с более низким социально-экономическим профилем. Хотя общая когорта MOMS-PI была демографически разнообразна, большинство женщин, которые испытали спонтанный PTB менее чем на 37 неделе беременности, имели африканское происхождение. Команда MOMS-PI (http://vmc.vcu.edu/momspi) также выявила признаки более высокого риска развития PTB у женщин, у которых спонтанные преждевременные роды были менее чем на 37 неделе беременности50. Женщины, у которых в дальнейшем наблюдался спонтанный PTB, с меньшей вероятностью демонстрировали вагинальную микробиоту с преобладанием Lactobacillus crispatus, как ранее сообщалось в других популяциях59,60,61,62, и с большей вероятностью демонстрировали повышенную пропорциональную численность нескольких таксонов, включая Sneathia amnii, Prevotella-родственные клады, таксон Lachnospiraceae, известный как BVAB1 и бактерию Saccharibacteria, известную как TM7-H1. Примечательно, что эти таксоны также были связаны с низким уровнем витамина D63, предполагая, что вагинальный микробиом может опосредовать связь между риском PTB и дефицитом витамина D64. Сигнатуры PTB также были отражены в метагеномных и метатранскриптомных измерениях, а вагинальные провоспалительные цитокины (включая IL-1β, IL-6, MIP-1β и эотаксин-1) были положительно коррелированы с PTB-ассоциированными таксонами. Для будущих возможных вмешательств удобно то, что влагалищные микробиомы матерей, которые испытали PTB, были наиболее отличными от таковых у контрольных матерей на ранних сроках беременности, и предварительная модель для предсказания риска PTB была наиболее чувствительной и специфичной, используя влагалищные профили микробиома из образцов, собранных до 24 недель беременности
Исследовательская группа MOMS-PI выявила интригующие связи между микробиотой влагалища, реакцией хозяина и исходами беременности, которые согласуются с вовлечением микроорганизмов, восходящих из влагалища, по крайней мере, в некоторых случаях спонтанного PTB. В качестве важного следующего шага вклад расового и демографического фона во влагалищный микробиом во время беременности в связи с исходами беременности должен быть полностью изучен в рамках согласованных широкомасштабных исследований50. Понятно, что PTB имеет сложную этиологию56. Следует изучить относительный вклад генетики и эпигенетики плода и матери, особенно в связи с генетическим изменением врожденной иммунной системы. Гармонизированные крупномасштабные исследования позволят разработать алгоритмы оценки риска для конкретных популяций с использованием профилей вагинального микробиома, особенностей генетического и пренатального (внутриутробного) генетического скрининга, биомаркеров, таких как цитокины и метаболиты, и ключевых клинических признаков из классических маркеров риска, включая материнский. возраст, индекс массы тела, история беременности (включая историю PTB), длина шейки матки и показатели стресса и других воздействий окружающей среды. С добавлением новых данных из микробиома, других факторов окружающей среды и мульти-омических входных данных новые алгоритмы обещают улучшить нашу способность предсказывать риск PTB на ранних сроках беременности, облегчить клинические испытания путем выявления пациентов с высоким риском и, в конечном счете, стратифицировать популяции пациентов на группы лечения.
Микробиом кишечника и воспалительные заболевания кишечника
Исследования микробиома кишечника при желудочно-кишечных заболеваниях имеют особенно долгую и детальную историю, особенно при сложных хронических состояниях, таких как воспалительные заболевания кишечника (ВЗК). ВЗК, включая болезнь Крона и язвенный колит, поражает миллионы людей во всем мире, причем рост заболеваемости за последние 50 лет или более совпадает с множеством факторов, таких как вестернизация, урбанизация, изменения в рационе питания, воздействие противомикробных препаратов и многие другие, которые могут влиять на гомеостаз микробиома хозяина65. Микробиом уже давно вовлечен в ВЗК, потенциально в качестве причинного фактора или фактора риска66,67, как объяснение гетерогенности реакции на лечение (то есть некоторые люди хорошо реагируют на относительно доброкачественные аминосалицилаты или кортикостероиды, в то время как другие все еще испытывают сильное воспаление даже после хирургического вмешательства)68, или как новый пункт терапевтического вмешательства (например, путем трансплантации фекальной микробиоты69,70). Хотя мета-омические методы были использованы для выявления функционально последовательных микробных реакций, которые помогают объяснить роль микробиома кишечника как части провоспалительной обратной связи в кишечнике во время болезни71, и было показано, что некоторые штаммы микроорганизмов являются специфичными для ВЗК72, до сих пор не существует всеобъемлющей модели специфических микробных, молекулярных и иммунных взаимодействий, чтобы объяснить начало заболевания и его динамическое прогрессирование.
Поэтому, чтобы лучше охарактеризовать механизмы дисрегуляции хозяина и микробиома во время заболевания, проект базы данных по воспалительным заболеваниям кишечника (IBDMDB) в течение одного года включал 132 человека из пяти клинических центров в составе HMP2 (рис. 3). Интегрированные продольные молекулярные профили активности микроорганизмов и хозяев были получены путем анализа 1785 образцов стула (самостоятельно собранных и отправленных по почте каждые две недели), 651 кишечной биопсии (собранных колоноскопически в начале исследования) и 529 ежеквартальных образцов крови. Насколько это было возможно, из одних и тех же наборов образцов были получены множественные молекулярные профили, в том числе метагеномы стула, метатранскриптомы73, метапротеомы, виромы, метаболомы74,75, экзомы хозяина, эпигеномы, транскриптомы и серологические профили, среди прочего, позволяющие одновременно наблюдать изменения в различных типах молекулярной и клинической активности хозяина и микробов с течением времени. Протоколы и результаты исследования, дополнительная информация о его инфраструктуре, а также необработанные и обработанные продукты данных76,77 доступны через информационный портал IBDMDB (http://ibdmdb.org), из Центра координации данных HMP2 (DCC; http://ihmpdcc.org), и в сопроводительной рукописи49.
Рис. 3. Динамика «хозяин-микробиом» при ВЗК.
IBDMDB наблюдала за более чем 100 участниками с ВЗК (болезнью Крона или язвенным колитом), а также контрольными лицами без ВЗК в течение одного года для оценки молекулярной активности хозяина и микробов во время изменений активности заболевания и воспаления желудочно-кишечного тракта. Почти все измеренные свойства микробиома хозяина показали изменения в активности или стабильности во время болезни, включая показанные здесь — не только микробные таксоны и микробную транскрипцию, но также небольшие молекулы в кишечнике, происходящие из хозяина и микробиоты, эпителиальные транскрипционные ответы в нескольких точках вдоль толстой кишки и уровень циркулирующих антител в серологии периферической крови.
Этот уникальный дизайн исследования позволил IBDMDB выявить множество различий в микробиоме и иммунном ответе хозяина с течением времени во время течения заболевания. Действительно, эти динамические изменения имели гораздо большую величину, чем кросс-секционные различия между клиническими фенотипами,которые были подчеркнуты предыдущими исследованиями67,71,78,79. Отчасти это было связано с проспективным характером когорты, которая набирала пациентов с болезнью Крона или язвенным колитом как в активный, так и в спокойный периоды заболевания, показывая, что микробные композиции у пациентов с ВЗК часто возвращаются к более контрольным, «базовым» конфигурациям, когда болезнь не активна. Выявив микробные конфигурации кишечника, которые наиболее сильно отличались от исходного уровня — независимо от конкретного болезненного состояния — исследование определило показатель дисбактериоза, в котором выявлялись сильно расходящиеся микробные композиции, которые имеют много общих черт, общих для общего воспалительного ответа (например, толерантность к окислительному стрессу). Однако этот дисбактериоз не был уникальным для микробной реакции на воспаление и был связан с другими биохимическими изменениями хозяина, указывая на новые потенциальные направления для управления системной дисрегуляцией при ВЗК. Они включали большие сдвиги в пулах ацилкарнитина и желчных кислот, повышение уровня антител в сыворотке крови и изменения в транскрипции для нескольких видов микробов. Параллельная транскриптомика и профилирование сообщества слизистой оболочки 16S-ампликонов из биопсий также выявили потенциальные факторы хозяина, которые могли бы формировать микробное сообщество, в частности, несколько хемокинов, выделяя их как вовлеченные в потенциально дисрегулированное взаимодействие в периоды активности болезни49.
Лонгитюдные мульти-омические профили исследования также позволили исследователям охарактеризовать стабильность и динамику взаимодействий хозяина и микробиома во время болезни, в частности выделив способы, при которых состояние сообщества и иммунные реакции явно менее стабильны у участников с ВЗК, чем у контрольных здоровых лиц. Во многих случаях микробиом участника с ВЗК полностью менялся в течение всего лишь нескольких недель (измеренных как максимальное несходство Брей-Кертиса с более ранними образцами от того же субъекта), в то время как такие сдвиги были редки у лиц без ВЗК. Основной вклад микробов в эти крупномасштабные сдвиги от одной временной точки к другой в значительной степени отражал различия, наблюдаемые при дисбактериозе, и эти сдвиги часто отмечали вход в периоды дисбиоза или выход из них. Наконец, долгосрочные комплементарные молекулярные измерения исследования позволили построить сеть из более чем 2900 значимых клеточных и молекулярных взаимодействий хозяина и микробов во время ВЗК, начиная от специфических микробных таксонов и заканчивая человеческими транскриптами и мелкомолекулярными метаболитами. Эта сеть механистических ассоциаций выявила несколько ключевых компонентов, которые являются центральными для изменений, наблюдаемых в ВЗК, выделив октаноилкарнитин, несколько липидов и короткоцепочечных жирных кислот, таксоны Faecalibacterium, Subdoligranulum, Roseburia, Alistipes и Escherichia, некоторые из которых находятся как на метагеномном, так и на метатранскриптомном уровнях, а также регуляторы интерлейкинов49. Сети механистических ассоциаций, подобные этой, могут дать ключ к распутыванию сложной системы взаимодействий, которая приводит к хроническому воспалению при ВЗК и других системных иммунных заболеваниях, связанных с микробиомом.
Мульти-омическое профилирование при преддиабете
Сахарный диабет 2-го типа (СД2) поражает более 10% взрослого населения США, и еще 30% проявляют ранние признаки заболевания (называемые преддиабетом)80; 70% последних разовьют диабет в течение своей жизни. СД2 характеризуется сложными взаимодействиями хозяина с микробиомом81,82, но мало что известно о системных изменениях во время преддиабета, их влиянии на биологические процессы или критическом переходе к полномасштабному СД2. Преддиабет и СД2 часто ассоциируются с инсулинорезистентностью, и поэтому исследования лиц с преддиабетом или инсулинорезистентностью открывают уникальные возможности для изучения самых ранних стадий диабета. Крайне важно создать глобальный и одновременный профиль как молекул хозяина, так и микробных молекул у людей с преддиабетом с течением времени, чтобы полностью понять молекулярные пути, которые затрагиваются у людей с преддиабетом и/или инсулинорезистентностью, и как эти условия влияют как на биологические реакции на экологические вызовы (например, вирусные инфекции83,84), так и на начало СД2.
Чтобы лучше понять СД2 на самых ранних стадиях, в рамках iHMP, интегрированного персонального проекта «Омикс» (IPOP)85 следили за 106 здоровыми и преддиабетическими лицами в течение ежеквартальных периодов здоровья, респираторной вирусной инфекции (ОРВИ) и других пертурбаций в течение примерно четырех лет51 (рис. 4). В одном таком возмущении подгруппа из 23 человек подверглась целенаправленному увеличению веса с последующей потерей веса86. В общей сложности было проведено профилирование 1092 коллекций по всем участникам. При каждом посещении кровь анализировалась на предмет молекулярного «омического» профилирования хозяина, а для микробиологического профилирования отбирались два типа образцов — мазки из носа и фекалии. Экзом каждого участника секвенировался один раз; в противном случае для каждого визита было отобрано 13 379 транскриптов из мононуклеарных клеток периферической крови, 722 метаболита и 302 белка из плазмы, а также 62 цитокина и фактора роста из сыворотки крови. Кроме того, тысячи кишечных и назальных микробных таксонов и вычислительно предсказанных генов были профилированы с использованием ампликонов 16S рРНК. Все посещения также интенсивно характеризовались 51 клиническим лабораторным исследованием. Кроме того, из-за акцента на СД2 был проведен ряд тестов на нарушение регуляции глюкозы, включая измерение уровня глюкозы натощак и гемоглобина А1с, пероральные тесты на толерантность к глюкозе и тесты на инсулинорезистентность.
Рис. 4. Дифференциальный ответ хозяина и микробов на диетические нарушения и инфекционные заболевания у лиц с преддиабетом.
Для комплексного персонального профилирования микробиома во время преддиабета за 106 пациентами проводилось наблюдение в течение четырех лет, причем пробы (в основном кровь и стул) собирались ежеквартально, а дополнительные пробы — в периоды ОРВИ и других стрессов. Геномы участников были секвенированы и измерения транскриптомов, протеомов, метаболомов и микробиомов проводились при каждом посещении, в дополнение к клиническим деталям. Инсулинорезистентные индивидуумы показали отличия от инсулино-чувствительных участников в различных измерениях, как на исходном уровне, так и в ответ на стрессы, такие как потеря веса и ОРВИ.
Исходные измерения, как правило, были стабильны у отдельных лиц, даже в течение длительных периодов времени, и лишь некоторые анализируемые вещества существенно изменялись с течением времени51. Однако многие анализы, такие как клинические лабораторные измерения, цитокиновые профили и таксоны кишечных микробов (в основном те, что имеют низкое обилие), сильно варьировали между отдельными индивидуумами. Участники, которые в конечном счете были инсулинорезистентны, имели различимые молекулярные и микробные паттерны в исходном состоянии от тех, кто был в конечном счете чувствителен к инсулину, и в рамках исследования был разработан аналит-тест, чтобы дифференцировать их. Примечательно, что люди, перенесшие ОРВИ или изменения веса, демонстрировали тысячи специфических молекулярных и микробных изменений во время этих возмущений, а инсулинорезистентные и чувствительные к инсулину люди реагировали на эти возмущения совершенно по-разному. Например, во время ОРВИ резистентные к инсулину участники продемонстрировали существенно сниженные и отсроченные воспалительные реакции (например, ответ острой фазы и передачу сигналов IL-1) и измененные микробные изменения кишечника по сравнению с чувствительными к инсулину участниками (например, у Lachnospiraceae и Rikenellaceae, но не бациллы). Соответственно, было меньше изменений в носовой микробиоте у участников, резистентных к инсулину, и как богатство, так и разнообразие носовых микроорганизмов снизились во время ОРВИ у участников, чувствительных к инсулину, но не резистентных к инсулину. Кроме того , глобальный анализ коассоциации среди тысяч профилированных молекул выявил специфические ассоциации у инсулинорезистентных индивидуумов, которые отличаются от тех, что наблюдаются у инсулиночувствительных участников, и наоборот, указывая на различные паттерны взаимодействия хозяина и микробиома в этих двух группах51.
Еще одна важная цель исследования состояла в том, чтобы оценить, как мульти–Омика микробиома хозяина и связанные с ней новые технологии могут быть использованы для лучшего управления здоровьем пациентов. Мы обнаружили, что проведение миллионов измерений на человека с течением времени позволило раннее выявление потенциальных болезненных состояний51,87. Они включали раннее обнаружение СД2, который развивался по-разному среди участников и был лучше обнаружен при различных анализах; например, некоторые люди впервые показали измерения в диабетическом диапазоне на тестах глюкозы натощак, в то время как другие делали это на тестах гемоглобина A1c, пероральных тестах толерантности к глюкозе или даже непрерывном контроле глюкозы. Эти результаты вместе с детальной характеристикой дисрегуляции глюкозы с течением времени иллюстрируют гетерогенность развития СД2. В целом эти данные привели к микробиологически связанным, клинически действенным открытиям в области здравоохранения в ряде заболеваний в дополнение к СД2, включая метаболические заболевания, сердечно-сосудистые заболевания, гематологические или онкологические заболевания и другие области; эти признаки часто присутствовали до появления симптомов, демонстрируя способность использования больших данных, включая микробиом, для лучшего управления здоровьем человека.
Ресурсы из HMP2
В совокупности на этапах HMP1 и HMP2 было получено в общей сложности 42 терабайта мульти-омических данных, которые архивируются и хранятся в DCC (Data Coordination Center — центр координации данных) по адресу http://ihmpdcc.org, а также в общедоступных и/или контролируемых хранилищах доступа, таких как архив чтения последовательности (SRA; https://www.ncbi.nlm.nih.gov/sra), база данных генотипов и фенотипов (dbGaP; https://www.ncbi.nlm.nih.gov/gap/), верстак Метаболомики (https://www.metabolomicsworkbench.org/) и другие. Все данные в DCC доступны для неограниченного использования, при этом подмножество метаданных проекта также передается, когда это разрешено институциональными наблюдательными советами (IRB), а также другие ограниченные данные (например, последовательности генома человека и защищенные метаданные), доступные через контролируемый доступ на dbGaP (проекты PRJNA398089, PRJNA430481, PRJNA430482, PRJNA326441, phs001719, phs000256, phs001626, phs001523 и другие). Формальные модели данных и связанные схемы отношений сущностей, созданные на всех этапах HMP, свободно доступны по адресу https://github.com/ihmpdcc/osdf-schemas. Веб-сайт DCC позволяет пользователям находить, запрашивать, искать, визуализировать и загружать данные из тысяч образцов с соответствующими метаданными. Как только пользователь определил набор файлов, условий, предметов или фенотипов, представляющих интерес, он или она может добавить этот набор в корзину для дальнейших операций. Затем файлы можно напрямую загрузить для использования на локальном сайте пользователя или в облаке. Таким образом, усилия HMP DCC проектируются в соответствии с заявленными целями NIH, чтобы сделать все данные, полученные из финансирования NIH, доступными, совместимыми и многоразовыми88. Успех этих усилий подтверждается стабильно высоким уровнем доступа пользователей к веб-ресурсам.
Комплементарные взаимодействия хозяина и микробиома
Хотя каждое из трех исследований HMP2 выявило новую биологию в своих соответствующих областях здоровья и болезней, среди них было удивительно много иммунных и экологических особенностей микробиома хозяина. Сочетание метагеномики дробовика, нецелевой метаболомики и иммунопрофилирования было особенно эффективным, поскольку во всех проектах это подмножество молекулярных измерений имело тенденцию эффективно улавливать интерпретируемые свойства хозяина и микробов, которые связаны с болезнью. И наоборот, генетические варианты, как правило, было трудно связать с микробиомом в таких небольших популяциях, которые были необходимы для того, чтобы глубоко профилировать мульти-омики с течением времени, и мы ожидаем, что секвенирование хозяев будет более полезным при интеграции в более крупные перекрестные обследования. Другим примечательным свойством было то, что, как и в большинстве исследований микробиома, изменения, происходящие внутри отдельных особей, популяций или фенотипов, часто были намного меньше, чем исходные различия между отдельными индивидуумами. Это особенно верно в отношении временных масштабов, связанных с микробиомом, для которых требовались повторные измерения с быстротой от нескольких дней до нескольких недель, чтобы зафиксировать наиболее специфические взаимодействия хозяина и микробиома. Таким образом, взаимодействие микробиома, связанного со здоровьем, может проявляться чрезвычайно разнообразно среди отдельных людей, что делает сочетание крупномасштабных популяционных обследований с внутриобъектными продольными профилями необходимым для понимания механизмов заболеваний, связанных с микробиомами.
В результате, другие аспекты взаимодействия хозяина с микробиомом были сильно локализованы и предметно-специфичны в каждом из трех исследований. Во всех трех состояниях микробные изменения и связанные с ними реакции хозяина были наиболее сильными, когда они регистрировались в то время, когда происходили изменения, и часто в ткани происхождения. Таким образом, из этих и других исследований становится ясно, что взаимодействия между хозяином и микробиомом оказывают как локальное, так и системное воздействие. Сильные локальные возмущения, инициируемые со стороны хозяина или микроба, могут индуцировать последующие пространственно-временные реакции, которые могут продолжаться во времени и/или в других тканях, предположительно с сигналами, переносимыми пространственно циркулирующими мелкими молекулами и/или временно генной регуляцией или ростом микроорганизмов, и вовлекать регуляторные цепи как с хозяином, так и с межмикробными компонентами. Таким образом, непрерывные скоординированные усилия по измерению различных свойств хозяина и микробов, участвующих в каждом состоянии, будут иметь важное значение для разработки целенаправленных и, при необходимости, персонализированных методов лечения связанных с микробиомом состояний, а также для выявления общих принципов, регулирующих взаимодействие хозяина и микробиома. Другие динамические взаимодействия, которые не были измерены во всех исследованиях, такие как первое микробное воздействие человека при рождении и последующее развитие иммунитета, также могут представлять собой ключевые факторы, влияющие на базовую персонализацию микробиома и помогающие объяснить связанную с болезнью динамику, основанную на событиях, имевших место годами или даже десятилетиями ранее.
Следующие шаги в мульти-омике микробиома
Коллективные результаты проектов NIH HMP, наряду со многими другими исследованиями, показывают, что микробиом является неотъемлемым компонентом биологии человека, играющим важную роль в здоровье и благополучии. Межиндивидуальная изменчивость и очень разнообразные ответы хозяин-микробиом с течением времени привели к разработке новых методов исследования популяционного микробиома с использованием множественных, дополнительных продольных измерений, а также подчеркивали необходимость дополнения таких исследований механистическими моделями для подтверждения причинных ассоциаций. Успешное закрытие самой программы HMP оставило прочное наследие нескольких научных поколений обученных исследователей микробиома человека; обеспечило сообщество богатыми данными, аналитическими и биоспецифическими ресурсами, а также предоставило возможность NIH и другим финансирующим учреждениям продолжать работу в широком диапазоне связанных с микробиомом состояний89. Финансирование науки о микробиомах, человека и других, в настоящее время координируется между центрами и институтами NIH (https://www.niaid.nih.gov/research/trans-nih-microbiome-working-group), а также многими др. государственными учреждениями и частными организациями. По мере того, как запускаются комплексные глобальные проекты по изучению аспектов персонализированной медицины, становится очевидным, что включение компонентов, ориентированных на воздействие микробиома человека, является информативным.
Как и в любом крупном исследовании, HMP2 вызвал больше новых вопросов, чем дал ответов. Этиология исходных межиндивидуальных различий в микробиоме и его динамических изменений с течением времени не были очевидны даже из широкого диапазона типов измерений, включенных в эти три исследования и популяции. Многие иммунные и биохимические реакции, по-видимому, связаны со специфическими штаммами, которые уникальны для одного или нескольких индивидуальных хозяев, но неясно, являются ли такие штаммы достаточными или необходимыми для их ассоциированных фенотипов заболеваний. Было выявлено несколько механизмов, с помощью которых сигналы в кишечнике могут передаваться системным состояниям, таким как диабет, но не специфическим малым молекулам или подмножествам иммунных клеток, с помощью которых они, вероятно, будут передаваться — особенно в других состояниях здоровья, которые еще не были изучены столь подробно. Наконец, каждое исследование HMP2 обязательно проводилось в пределах географически и генетически ограниченной популяции, и глобальные различия в ранних жизненных событиях, воздействии инфекционных заболеваний или диете могут изменить то, как динамика микробиома вносит свой вклад в развитие болезни человека. Микробиология, связанная с человеком, теперь явно простирается за пределы инфекционных и желудочно-кишечных заболеваний в области, едва мыслимые несколько десятилетий назад, включая метаболизм, неоплазию, здоровье матери и ребенка и функции центральной нервной системы. По мере того как NIH HMP подходит к концу, становится ясно, что его результаты выявили множество новых направлений исследований и технологий для будущих исследований, и мы с нетерпением ожидаем новых открытий, основанных на ресурсах программы и захватывающих находках, которые еще предстоит сделать.
Литература к разделу «Интегративный Проект Микробиом Человека»
Дополнительная информация
ВИДЕОЛЕКЦИЯ О МИКРОБИОМЕ
Рассказывает Константин Северинов — доктор биологических наук, заведующий лабораторией регуляции экспрессии генов элементов прокариот Института молекулярной генетики РАН, заведующий лабораторией молекулярной генетики микроорганизмов Института биологии гена РАН, профессор Университета Ратгерса (США), профессор Сколковского института науки и технологий (SkolTech)
Константин Северинов:
«Самый большой орган в нашем организме не печень и не мозг, а микробы, которые образуют т.н. микробиом…» (видео→)
к.б.н. Дмитрий Алексеев
«Микробиота кишечника и заболевания» (видео→)
Другие видеолекции к.б.н. Дмитрия Алексеева см. по ссылкам:
Микробиота кишечника (к.б.н. Дмитрий Алексеев)
Пробиотики и пребиотики (к.б.н. Дмитрий Алексеев)
Школа Ферментации с Анной Дроздовой и Дмитрием Алексеевым
Примечание:
Следует отметить, что большинство предлагаемого здесь материала рассказывает об исследовании микробиома (в т.ч. в свете его связи с патологиями) без учета последних достижений некоторых омических направлений в науке по изучению микроорганизмов. Для изучения микроорганизмов сегодня в основном используют четыре подхода: метагеномное — исследование ДНК (методом секвенирования), метатранскриптомное — изучение РНК, метапротеомика — изучение белков, метаболомика — изучение метаболитов. Если метагеномика уже широко представлена в данных исследованиях, то статьи по микробиомному изучению с широким использованием метаболомики и др. омических инструментов, будут размещаться в отдельных соответствующих подразделах, обозначенных на странице по метагеномному анализу (будут даваться ссылки — как это уже сделано по метаболомике). Также некоторый материала по микробиому представлен отдельно в разделе об иммунитете, а также в разделе о микробиомно-мозговом взаимодействии, хотя такое разделение условно (ссылки на значимые работы будут дублироваться).
Обзорные статьи об исследовании микробиома и его взаимосвязей с различными патологиями:
Обновляемая информация: Новости и обзор литературы
Основные постоянные ссылки:
МИКРОБИОМ и ВЗК
МИКРОБИОМ И РАК
МИКРОБИОМ, СЕРДЦЕ И СОСУДЫ
МИКРОБИОМ И ПЕЧЕНЬ
МИКРОБИОМ И ПОЧКИ
МИКРОБИОМ И ЛЕГКИЕ
МИКРОБИОМ И ПОДЖЕЛУДОЧНАЯ ЖЕЛЕЗА
МИКРОБИОМ И ЩИТОВИДНАЯ ЖЕЛЕЗА
МИКРОБИОМ И КОЖНЫЕ БОЛЕЗНИ
МИКРОБИОМ И КОСТИ
МИКРОБИОМ И ОЖИРЕНИЕ
МИКРОБИОМ И САХАРНЫЙ ДИАБЕТ
МИКРОБИОМ И ФУНКЦИИ МОЗГА
Кое-что архивное:
- Нацеливание на микробиоту в здравоохранительной практике
- Микробиота кишечника и хроническое системное воспаление низкой степени
- Вирусные инфекции, микробиом и пробиотики
- Микробиом и нарушение функций щитовидной железы
- Взаимодействие между иммунной системой и микробиотой при воспалительных заболеваниях кишечника
- Кишечный микробиом, иммунитет и инфекционные заболевания
- Синдром раздраженного кишечника и кишечный микробиом
- Изменения микробиома кожи и кишечника при псориазе и псориатическом артрите
- Микробиота кишечника: ключевой регулятор метаболических заболеваний
- Роль микробиоты кишечника в иммунитете и воспалении
- Болезни, лекарства и дисбактериоз: понимание микробных сигнатур при метаболических заболеваниях и медицинских вмешательствах
- Влияние пробиотических штаммов на активность заболевания и проницаемость кишечника при псориатическом артрите
- Причинно-следственные эффекты микробиоты на иммуноопосредованные заболевания
- Дисбиоз микробиома кишечника и иммунометаболизм: новые рубежи в лечении метаболических заболеваний
- Последние достижения в исследовании псориаза, ключ к загадочному отношению к микробиому кишечника
- Идентификация кишечных бактерий. Бактероидеты и Фирмикуты
- Понимание о микробиоме кишечника с точки зрения токсикологии
- Формирование кишечной микробиоты ребенка
- Роль кишечного микробиома при астме.
- Дисбиоз кишечника во время гриппа способствует легочной пневмококковой суперинфекции через измененную продукцию КЦЖК, в частности, уксусной кислоты.
- Роль микробиоты в иммунитете и воспалении (+ видео)
- ВЗК, микробиота и иммунитет слизистой оболочки
- Роль кишечной микробиоты в гомеостазе кишечника и заболеваниях человека
- Микробиом кишечника как клинический инструмент лечения заболеваний желудочно-кишечного тракта
- Взаимосвязь между различными патологиями и изменениями кишечной микробиоты
- Микробиом, метаболом и воспалительные заболевания кишечника (ВЗК)
- Роль кишечной микробиоты при этиопатогенезе ВЗК
- Неалкогольная жировая болезнь печени (НАЖБП), микробиом и пробиотики
- Стеатоз печени, ось кишечник-печень и микробиом
- Триметиламин N-оксид (ТМАО), кишечная микробиота и атеросклероз
- Остеоартрит кисти и кишечный дисбактериоз
- Микробиом и сахарный диабет 2-го типа
- Энтеротип Bacteroides ассоциируется с высоким риском у пациентов с сахарным диабетом
- Влияние кишечной микробиоты на здоровье поджелудочной железы (панкреатит, диабет, рак)
- Микробиом, проницаемость кишечника и тканевые бактерии (о синдроме «дырявого кишечника» и транслокации бактерий)
- Дисбиотическое нарушение кишечной проницаемости
- Дисбиоз кишечника и хроническая болезнь почек
- Микробиота и иммуноопосредованные кожные заболевания
- Функциональная роль про- и пребиотиков в здоровье и заболевании кожи
- Микробиом кишечника при рассеянном склерозе
- Микробиом полости рта, кишечника и артрит
- Микробиомная ассоциация с воспалением и болью коленного сустава (микробиом может стать терапевтической мишенью при боли в суставах, связанной с остеоартрозом)
- Бифидобактерии могут быть эффективными в предотвращении колоректального рака
- Пропионовокислые бактерии в профилактике и терапии рака желудка и рака кишечника
- Рак молочной железы и его связь с микробиотой
- Микробы для здоровья (обзорный материал о кишечном микробиоме)
- Кишечный микробиом и нервная анорексия
- Диктатура микробиома (глава из книги Хью Леннарда)
- Миниобзор о взаимодействии кишечного микробиома и болезни Паркинсона
- Здоровье щитовидной железы и кишечный микробиом
- Измерение эффективности микробиома: роль доступных для восприятия датчиков
- Среда доминирует над генетикой хозяина в формировании микробиома
- Замедление старения: роль питательных веществ и микробиоты в модуляции эпигенома
- Фаготерапия при желудочно-кишечных заболеваниях
- Как бактерии властвуют над вашим телом. Микробиом (занимательное анимационное видео от немецкого YouTube-канала Kurzgesagt)
- Кишечная (энтеральная) нервная система (ЭНС) и микробиом
- Взаимосвязи между микробиомами полости рта и кишечника
- Новое в изучении микробиома: «Кишечник на чипе«
- Кишечная микрофлора превращает человек в сверхорганизм (доля человеческих генов в совокупном геноме с симбиотическими микроорганизмами составляет не более 1%)
- Как микробы, живущие в нашем теле, определяют наше здоровье и нашу личность (книга проф. Роба Найта и его видеолекция)
- «Бактерии» с Андреем Шестаковым (научпопфильм от ТВ-канала «Наука»)
- Взаимосвязи между микробным дисбактериозом и кожными заболеваниями (ось кишечник-кожа)
*Получить больше информации о понятии «микробиом» можно также из др. разделов и опубликованных статей на нашем сайте, например, в новостях сайта (news), или сделав запрос в поиске по сайту
Будьте здоровы!
ССЫЛКИ К РАЗДЕЛУ О ПРЕПАРАТАХ ПРОБИОТИКАХ
- ПРОБИОТИКИ
- ПРОБИОТИКИ И ПРЕБИОТИКИ
- СИНБИОТИКИ
- ДОМАШНИЕ ЗАКВАСКИ
- КОНЦЕНТРАТ БИФИДОБАКТЕРИЙ ЖИДКИЙ
- ПРОПИОНИКС
- ЙОДПРОПИОНИКС
- СЕЛЕНПРОПИОНИКС
- ГЕМОПРОПИОВИТ
- БИФИКАРДИО
- ПРОБИОТИКИ С ПНЖК
- МИКРОЭЛЕМЕНТНЫЙ СОСТАВ
- БИФИДОБАКТЕРИИ
- ПРОПИОНОВОКИСЛЫЕ БАКТЕРИИ
- МИКРОБИОМ ЧЕЛОВЕКА
- МИКРОФЛОРА ЖКТ
- ДИСБИОЗ КИШЕЧНИКА
- МИКРОБИОМ и ВЗК
- МИКРОБИОМ И РАК
- МИКРОБИОМ, СЕРДЦЕ И СОСУДЫ
- МИКРОБИОМ И ПЕЧЕНЬ
- МИКРОБИОМ И ПОЧКИ
- МИКРОБИОМ И ЛЕГКИЕ
- МИКРОБИОМ И ПОДЖЕЛУДОЧНАЯ ЖЕЛЕЗА
- МИКРОБИОМ И ЩИТОВИДНАЯ ЖЕЛЕЗА
- МИКРОБИОМ И КОЖНЫЕ БОЛЕЗНИ
- МИКРОБИОМ И КОСТИ
- МИКРОБИОМ И ОЖИРЕНИЕ
- МИКРОБИОМ И САХАРНЫЙ ДИАБЕТ
- МИКРОБИОМ И ФУНКЦИИ МОЗГА
- АНТИОКСИДАНТНЫЕ СВОЙСТВА
- АНТИОКСИДАНТНЫЕ ФЕРМЕНТЫ
- АНТИМУТАГЕННАЯ АКТИВНОСТЬ
- МИКРОБИОМ и ИММУНИТЕТ
- МИКРОБИОМ И АУТОИММУННЫЕ БОЛЕЗНИ
- ПРОБИОТИКИ и ГРУДНЫЕ ДЕТИ
- ПРОБИОТИКИ, БЕРЕМЕННОСТЬ, РОДЫ
- ВИТАМИННЫЙ СИНТЕЗ
- АМИНОКИСЛОТНЫЙ СИНТЕЗ
- АНТИМИКРОБНЫЕ СВОЙСТВА
- КОРОТКОЦЕПОЧЕЧНЫЕ ЖИРНЫЕ КИСЛОТЫ
- СИНТЕЗ БАКТЕРИОЦИНОВ
- АЛИМЕНТАРНЫЕ ЗАБОЛЕВАНИЯ
- МИКРОБИОМ И ПРЕЦИЗИОННОЕ ПИТАНИЕ
- ФУНКЦИОНАЛЬНОЕ ПИТАНИЕ
- ПРОБИОТИКИ ДЛЯ СПОРТСМЕНОВ
- ПРОИЗВОДСТВО ПРОБИОТИКОВ
- ЗАКВАСКИ ДЛЯ ПИЩЕВОЙ ПРОМЫШЛЕННОСТИ
- НОВОСТИ
Что такое микробиом человека? Какие микроорганизмы обитают в кишечнике? Какие факторы помимо диеты и места жительства влияют на состав микробиома? Сколько функций выполняет кишечный микробиом и как можно его поддержать? Если вам интересно узнать ответы на эти вопросы, скорее приступайте к чтению.
Содержание:
- Что такое микробиом и от чего зависит его состав.
- Основной состав микробиоты кишечника.
- Функции микробиоты кишечника.
- Как поддержать микробиом кишечника.
- Рецепты ферментированных продуктов.
В последние десятилетия мощно развивается наука о генах не только человека, но и микробов, которые живут в нем. Интересный факт: при исследовании генов человека выяснилось, что только около половины принадлежит ему, а остальные — микробные. Поэтому можно сделать вывод, что человек — среда обитания для большого количества микроорганизмов. Почти в каждом органе можно обнаружить микробов-«сожителей». Свой микробиом есть у дыхательной, мочеполовой, пищеварительной систем, кожи и так далее.
Хотите знать, какие функции выполняют бактерии в нашем организме? Сколько их должно быть в норме и какие анализы можно сдать для диагностики состояния микробиома? Прослушайте бесплатный двухдневный интенсив «Биохимия микробиома» и найдите ответы на свои вопросы!
Что такое микробиом и от чего зависит его состав
Микробиом (micro — «маленький», bios — «жизнь») — это собирательное понятие, включающее все микроорганизмы, которые живут на теле и в организме.
Микробиота — это более узкое понятие, включающее в себя совокупность микроорганизмов какой-то определенной области или органа. Например, кишечника, влагалища, кожи.
В организме человека насчитываются десятки триллионов разных микробов, среди них можно выделить примерно 1800 родов и 40 000 штаммов. Сюда относятся не только бактерии, но и грибы, археи, вирусы. Около трети всех микроорганизмов примерно одинаковы у всех людей, а оставшиеся — индивидуальное разнообразие. Поэтому микробиом — это индивидуальная характеристика, такая же, как отпечаток пальца. При изучении микробиоты у разных членов одной семьи находят схожие компоненты.
Кишечный микробиом склонен к изменениям и зависит от ряда факторов:
1. пищевые привычки (какие продукты преобладают в рационе — белки, жиры или углеводы, есть ли разнообразие в клетчатке, или в питании преобладают простые сахара и так далее);
2. образ жизни (напряженная работа или хронический стресс, физическая активность или сидячий образ жизни, режим дня, время отхода ко сну, смена часовых поясов и климата);
3. окружающая среда (место проживания — большой город, например Москва, или небольшая деревня, использование агрессивных моющих средств, постоянная обработка поверхностей антисептиками, экологическая обстановка);
4. хронические заболевания и различные нарушения в работе желудочно-кишечного тракта (снижение кислотности, нарушение желчеоттока, воспалительные заболевания кишечника, нарушение пищеварения);
Ощущаете чувство тяжести после еды? Беспокоят такие симптомы, как отрыжка, изжога или горечь во рту? Проверьте себя, пройдите опрос «Здоровье ЖКТ» и обратитесь к нутрициологу.
5. прием лекарств (антибиотики, пробиотики, пребиотики, цитостатики, ионизирующее излучение, обезболивающие или гормональные препараты и так далее);
6. различные инфекционные заболевания (ротавирусы, энтеровирусы, коронавирус, сальмонеллез, дизентерия и так далее);
7. возраст. Раньше считалось, что плод в животе мамы находится в стерильной среде, а заселение микробами происходит во время и после рождения. Последние исследования говорят о том, что плацента, околоплодная жидкость и меконий содержат микроорганизмы. Но основное заселение начинается после рождения. При естественных родах процесс идет более физиологично, организм ребенока заселяется флорой родовых путей матери, при операции кесарева сечения — бактериями кожи мамы или папы, рук медицинского персонала. Большое значение имеет естественное или искусственное вскармливание. При грудном вскармливании заселение идет физиологично, лакто- и бифидобактериями, при кормлении смесями преобладают бактероиды. У пожилых людей постепенно снижается разнообразие микробиома из-за уменьшения числа тех же лакто- и бифидобактерий.
Что входит в топ анализов для здоровой беременности и легких родов? Узнайте, как родить здорового ребенка и правильно подготовиться к родам, на бесплатном онлайн-вебинаре МИИН.
Основной состав микробиоты кишечника
В кишечнике находится самая густонаселенная и изученная часть микробиоты. Если на коже численность организмов составляет около 104–105, то в тонком кишечнике обнаруживается уже 105–109, а в толстом еще больше — 1011–1012. Разные виды микроорганизмов располагаются в разных местах: какие-то преимущественно в просвете, а какие-то, наоборот, предпочитают находиться около слизистой оболочки. К просветной микрофлоре относятся бактероиды, бифидобактерии, стрептококки, энтерококки, клостридии, лактобациллы и руминококки. А микробиота кишечника, связанная со слизистой, — это клостридии, лактобациллы, энтерококки. Эти бактерии тесно связаны с организмом человека, могут влиять на многие процессы, в том числе запускать и поддерживать процессы хронического воспаления.
Среди обитателей кишечного тракта можно выделить облигатную микрофлору (микроорганизмы, которые составляют 90% микробиома и находятся там постоянно). К ним относятся:
- бифидобактерии — анаэробы (для их жизнедеятельности не нужен кислород). По форме это грамположительные палочки. Есть в человеческом организме от рождения до старости, у детей служат основными обитателями (занимают 90–98%), постепенно с возрастом их количество снижается;
- лактобактерии — грамположительные палочки, которые могут располагаться в цепочке друг за другом или по одной. Заселяют организм человека в раннем послеродовом периоде, так как содержатся в большом количестве в грудном молоке;
- пропионобактерии — анаэробы, которые закисляют среду вокруг себя и препятствуют заселению кишечника патогенными и условно-патогенными бактериями;
- кишечные палочки — грамотрицательные палочковидные бактерии. Именно они участвуют в образовании витаминов группы В и К, вырабатывают антибиотикоподобные вещества против патогенных штаммов палочек;
- бактероиды — анаэробные грамотрицательные бактерии, которые заселяют кишечник постепенно после второго полугодия жизни.
Вторая группа — это факультативные микроорганизмы, состав которых может меняться время от времени (составляют около 10%). В эту группу входят:
- пептококки — анаэробные бактерии, участвуют в образовании короткоцепочечных жирных кислот;
- стафилококки — негемолитической группы, попадают в организм из окружающей среды;
- стрептококки — непатогенные штаммы, представлены молочнокислым стрептококком, препятствуют заселению кишечника патогенной флорой;
- бациллы — представлены аэробными бациллами и анаэробными клостридиями. Участвуют в переваривании углеводов. Важно, чтобы их количество не было слишком большим;
- дрожжи и дрожжеподобные грибы — также являются представителями условно-патогенной флоры. Их наличие не говорит о патологии, важно следить за количеством.
В заселении кишечника микробами можно выделить четыре основные фазы:
- первая фаза — с рождения до двухнедельного возраста малыша. В это время преобладают стрептококки и кишечные палочки;
- вторая фаза напрямую связана с видом вскармливания ребенка и длится до начала прикорма. Активно происходит заселение кишечника бифидо- и лактобактериями;
- третья фаза — от начала прикорма до завершения грудного вскармливания. В этот период постепенно растет количество микроорганизмов из рода бактероидов и грамположительных кокков;
- четвертая фаза наступает после полного прекращения кормления грудью. Эта фаза характеризуется относительным постоянством микробного состава.
Научитесь управлять своим здоровьем с помощью знаний о работе самого большого сообщества микроорганизмов в вашем организме. Регистрируйтесь по ссылке и получите гайд самодиагностики состояния микробиома в подарок!
Функции микробиоты кишечника
Кишечные микроорганизмы играют большую роль в организме человека. Некоторые источники называют микробиоту отдельным органом. Микробиота кишечника не просто участвует в пищеварении, но и выполняет многие другие функции: влияет на иммунитет, настроение, синтезирует витамины.
Рассмотрим подробнее все функции:
1. Участие в пищеварении.
В организме человека есть довольно большой спектр ферментов для расщепления белков и жиров, а вот для переваривания сложных углеводов набор скуден. Эволюционно так сложилось, что эту функцию на себя взяла кишечная микробиота, которая умеет расщеплять разные виды пищевых волокон. Бактероиды активно расщепляют крахмал и гемицеллюлозу, руминококки — крахмал и целлюлозу, бифидобактерии — олигосахариды. В результате расщепления разных пищевых волокон образуются короткоцепочечные жирные кислоты: ацетат (содержит два атома углерода), пропионат (три атома), бутират (четыре атома) и так далее.
Кишечная микробиота косвенно участвует в эмульгировании жиров, то есть разбивает крупные молекулы жира на мелкие через трансформацию желчных кислот. Некоторые из них могут расщеплять аминокислоты до азота и газов.
2. Участие в синтезе многих веществ.
Микробиом кишечника способен образовывать витамины:
- группы В (В1 — тиамин, В2 — рибофлавин, В6 — пиридоксин, В12 — цианокобаламин);
- С;
- К;
- фолиевую кислоту (В9);
- никотиновую кислоту (В3);
- пантотеновую кислоту (В5).
Эти вещества используются самими микроорганизмами и человеком.
Микробы принимают активное участие в производстве нейроактивных веществ:
- норадреналина;
- серотонина — гормона радости;
- гамма-аминомасляной кислоты, которая тормозит передачу нервного импульса и подавляет процессы возбуждения в нервной системе;
- оксида азота;
- сероводорода.
В связи с активным влиянием микрофлоры на головной мозг многие ученые называют кишечник вторым мозгом. Взаимодействие микрофлоры происходит через эти нейромедиаторы при участии блуждающего нерва, который передает информацию в центральную нервную систему.
Болезни нервной системы — одни из самых распространенных! Узнайте, в каком состоянии находится ваша нервная система и как о ней заботиться, на бесплатном интенсиве «Биохимия нервной системы».
Благодаря полезным бактериям при расщеплении пищевых волокон образуются короткоцепочечные жирные кислоты, которые выполняют в организме человека много функций:
- обеспечивают энергией колоноциты (клетки толстого кишечника);
- стимулируют рост и обновление клеток слизистой кишечника, что препятствует развитию различных новообразований (в том числе злокачественных);
- улучшают кровоток в слизистой кишечника;
- стимулируют образование в толстом кишечнике слизи, которая защищает его от проникновения вредных микроорганизмов и веществ;
- улучшают всасывание воды, солей, натрия, калия, магния;
- снижают кислотность в просвете толстой кишки;
- поддерживают целостность слизистой;
- стимулируют перистальтику кишечника, что улучшает продвижение пищевого комка и профилактирует развитие запоров;
- ацетат после всасывания в кровь служит субстратом для клеток мышц, сердца, почек, головного мозга;
- пропионат участвует в липидном обмене и образовании глюкозы из аминокислот (глюконеогенезе) в печени;
- бутират оказывает противовоспалительный эффект и снижает риск развития аутоиммунных заболеваний.
3. Создание колонизационной резистентности.
Кишечная микробиота не дает разрастаться условно-патогенным микроорганизмам и заселяться патогенным. Эта функция осуществляется за счет нескольких механизмов:
- некоторые полезные бактерии прикрепляются к слизистой кишечника и создают защитный слой, к которому не могут прикрепиться патогенные и условно-патогенные микробы;
- другие выделяют вещества, которые подавляют рост «плохих» микроорганизмов, в первую очередь это короткоцепочечные жирные кислоты, другие органические кислоты, перекись водорода (приводит к снижению рН), микробный лизоцим, бактериоцины (эндогенные антимикробные вещества бактериального происхождения);
- все они конкурируют с патогенными и условно-патогенными организмами за ресурсы (питание и энергию).
В организме человека тоже функционирует несколько механизмов, которые препятствуют заселению слизистой кишечника патогенными микроорганизмами:
- слущивание эпителия слизистых оболочек;
- продукция слизи;
- воздействие соляной кислоты в желудке и желчных кислот в кишечнике;
- выработка защитных иммуноглобулинов класса А;
- синтез бактерицидных веществ на протяжении желудочно-кишечного тракта (например, лизоцим).
4. Утилизация токсинов.
5. Профилактика нарушения чувствительности клеток к глюкозе и снижение риска развития сахарного диабета 2-го типа.

6. Участие в иммунном ответе.
Благодаря кишечному микробиому иммунитет обучается, находится в состоянии постоянного напряжения, профилактирует развитие аутоиммунных заболеваний. В кишечнике расположено около 70% иммунной системы, у нее даже есть название — GALT (лимфоидная ткань, ассоциированная со слизистой кишечника).
Часто болеете простудными заболеваниями? Ребенок пошел в детский сад и каждые две недели приходится брать больничный? Хотите узнать, как укрепить здоровье, но не знаете как? Регистрируйтесь на бесплатный интенсив «Нутрициолог с нуля» и узнайте, как профилактировать болезни.
7. Улучшение функции нервной системы благодаря синтезу нейропептидов (серотонин — гормон счастья — уменьшает проявления депрессии) и уменьшению отложения амилоида. Эти процессы профилактируют развитие болезней нервной системы, в первую очередь болезни Альцгеймера.
8. Помощь при похудении.
Ученые доказали, что кишечная микробиота худых людей отличается от микробиоты людей с лишним весом. У последних наблюдается снижение разнообразия, увеличение количества актинобактерий, золотистого стафилококка и энтеробактерий, а также снижение бактероидов.
Уровень короткоцепочечной жирной кислоты бутирата в крови влияет на чувство насыщения путем стимуляции синтеза и продукции аппетит-регулирующих пептидов, улучшает расщепление триглицеридов, уменьшает проявления инсулинорезистентности и улучшает обмен холестерина.
Надоело сидеть на диетах? Получаете кратковременный результат, вес в итоге возвращается? Хотите разобраться с темой лишнего веса раз и навсегда? Разложить информацию по полочкам поможет интенсив «Биохимия стройности».
9. Тяга к определенному виду продуктов.
Чем больше количество бактерий определенного вида и ниже разнообразие, тем выше риск злоупотребления продуктами, которые эти бактерии перерабатывают. Например, бактероиды предпочитают белковую пищу, бифидобактерии — пищевые волокна, особенно бобовые, лактобациллы — резистентный крахмал, зеленые бананы, а фирмикуты любят жир и сахар.
Как поддержать микробиом кишечника
У кишечного микробиома есть два основных источника получения питательных веществ. Первый — поступающие с рационом пищевые волокна. И чем разнообразнее эти источники, тем разнообразнее состав микробиома человека. Второй источник — кишечная слизь. Чтобы не допустить ухудшения функции кишечного барьера, необходимо поддерживать микробиом и включать в меню разнообразные источники клетчатки.
Пищевые волокна могут быть растворимыми (инулин, камедь, слизь, пектин, альгинат, резистентный крахмал) и нерастворимыми (целлюлоза, гемицеллюлоза, лигнин).
Ниже приведена таблица, где указаны основные источники клетчатки.
| Продукт | Количество клетчатки в 100 г продукта |
|---|---|
| грибы белые сушеные | 26,2 г |
| инжир сушеный | 18,2 г |
| курага | 18 г |
| яблоки сушеные | 14,9 г |
| гречка | 14 г |
| соя | 13,5 г |
| фасоль | 12,4 г |
| чечевица | 11,5 г |
| горох | 10,7 г |
| нут | 9,9 г |
| рис | 9,7 г |
| миндаль | 7 г |
Суточная норма клетчатки составляет около 30 г для взрослых и 10 г плюс 1 г за каждый год жизни для ребенка.
При включении в рацион клетчатки в виде продуктов или добавок необходимо соблюдать несколько важных условий:
- пить достаточное количество воды — не менее 30 мл на 1 кг веса;
- увеличивать количество клетчатки постепенно, при повышенном вздутии можно начинать с термически обработанного варианта;
- не принимать совместно с лекарствами (необходимо соблюдать промежуток в 2 часа);
- сухую клетчатку можно разводить в растительном молоке, овощном смузи или воде. Не рекомендуется разводить в молоке и молочных продуктах.
Ферментированные продукты — своеобразный суперфуд, который содержит полезные бактерии, пищевые волокна и продукты их жизнедеятельности. Все эти продукты можно включать в рацион:
- ферментированная капуста;
- чайный гриб (комбуча);
- соленые огурцы;
- йогурт;
- квас;
- мисо-суп;
- ферментированные соевые бобы (натто);
- выдержанный сыр;
- кефир;
- кимчи.
Хотите ознакомиться с самыми полезными продуктами для кишечника? Переходите по ссылке и читайте полезный гайд «Топ продуктов для кишечника».

Рецепты ферментированных продуктов
Ферментированные продукты, приготовленные самостоятельно, содержат индивидуальный набор бактерий, которые попадают в продукт с кожи рук, одежды, посуды. Получается, что каждый человек создает индивидуальный набор полезных бактерий. При этом они содержат пробиотики (сами полезные бактерии), пребиотики (вещества, необходимые для роста этих бактерий) и метабиотики (вещества, которые образуются в результате жизнедеятельности бактерий).
1. Ферментированная капуста.
Необходимо: кочан капусты, соль не йодированная (около 30 г).
Приготовление: нашинковать капусту, посыпать солью и отжимать ее в течение 7–10 минут, чтобы она дала сок. Переложить капусту в банку, чтобы сок покрывал продукт полностью (если не хватает, можно добавить воды). Сверху поставить груз и прикрыть крышкой, чтобы был доступ для кислорода. Оставить в темном месте (без прямых солнечных лучей) при комнатной температуре на 3–7 дней. Все зависит от предпочтений: чем дольше стоит, тем насыщеннее получается вкус. Затем можно убрать в холодильник и хранить в прохладном месте.
2. Соленые огурцы.
Необходимо: огурцы одинакового размера (1,5 кг), соль (50 г), чеснок (3–4 зубчика), лавровый лист (3–4 шт.), зонтики укропа, листья хрена, листья вишни или смородины, перец горошком (5 шт.), кипяченая вода (1 л).
Приготовление: вымыть тщательно огурцы и зелень, чеснок нарезать на дольки. Приготовить рассол: в литре холодной воды развести соль, перемешать. Огурцы сложить в банки, добавить зелень и залить рассолом, оставить при комнатной температуре на 4–5 дней. От времени выдержки зависит вкус огурцов. Затем поставить в прохладное место.
3. Кокосовый йогурт.
Необходимо: кокосовое молоко, сухая закваска (1 г), свежая или замороженная малина, семена чиа (1 ч. л.).
Приготовление: взбить кокосовое молоко миксером. Добавить закваску для йогурта, хорошо перемешать и разлить в маленькие баночки, накрыть их марлей. Оставить при комнатной температуре на 24–48 часов или при небольшой температуре в мультиварке на 8 часов. Затем переставить в холодильник. Перед подачей можно добавить свежие ягоды и семена чиа.
Добавляйте в каждый прием пищи разнообразные варианты клетчатки, чередуйте разные источники. Это гарантирует вам отличное здоровье, настроение и внешний вид.
Список литературы:
- https://ecobios.ru/novyij-resurs/
- http://gastro-rsmu.ru/wp-content/uploads/2017/06/mikrobiota.pdf
- https://omnidoctor.ru/upload/iblock/730/730db09c5d37b5835f04dbec81eaffad.pdf
- https://propionix.ru/mikroflora-zhkt
- https://cyberleninka.ru/article/n/mikrobiota-kishechnika-rol-v-razvitii-razlichnyh-patologiy/viewer
- https://cyberleninka.ru/article/n/izmeneniya-kishechnoy-mikroflory-assotsiirovannye-s-vozrastom-i-obrazom-zhizni/viewer
Дата последнего обновления: 18.02.2023
Среднее время прочтения: 10 минут
Мир бактерий, вирусов, грибов и простейших, обитающих внутри и/ или на коже человека, называют микробиотой, а совокупность их генов — микробиомом1,2. Оба названия пришли на смену привычному многим термину «микрофлора». Он считается устаревшим, поскольку «флора» относится к миру растений, а не микробов1.
Основатель учения о микробиоте и лауреат Нобелевской премии И.И. Мечников писал: «Многочисленные ассоциации микробов, населяющих кишечник человека, в значительной мере определяют его духовное и физическое здоровье»5. Спустя десятилетия его мысль была доказана. О том, что такое здоровый микробиом кишечника и как улучшить его состав — расскажем в статье.
Функции микробиома (микробиоты) кишечника
В организме здорового человека все микроорганизмы мирно сосуществуют, а микробиом кишечника даже называют «виртуальным» органом — так много ключевых ролей он играет в бесперебойной повседневной работе нашего тела3,4.
Многие микроорганизмы передаются детям через материнское молоко и продолжают заселять тело до зрелого возраста (20-25 лет)7. В кишечнике взрослого живут от 100 до 400 триллионов микроорганизмов. Все они образуют настоящий физический барьер длиной более 7 метров между слизистой кишечника и внешней средой. Каждый участок желудочно-кишечного тракта (ЖКТ) населяет особая микробиота. Количество бактерий постепенно увеличивается от желудка до тонкого кишечника, а больше всего их обитает в толстой кишке6.
Микробиом кишечника выполняет множество функций8:
- защищает от кишечных инфекций, в том числе за счет участия в иммунном ответе;
- вырабатывает витамины;
- участвует во всасывании минералов;
- активирует ферменты в тонкой и толстой кишке;
- регулирует перистальтику (волнообразные сокращения) кишечника и регулярность стула.
Кишечные бактерии — это настоящая «фабрика» по производству многих важных для организма веществ, таких как жирные кислоты и нейрогормоны, или посредники передачи нервного импульса. Между кишечником и мозгом есть прямая и двусторонняя связь, называемая осью «мозг-кишечник». Микробиота участвует в этой оси, она вырабатывает те же нейрогормоны, которые образуются в головном мозге6,11. Среди них: гамма-аминомасляная кислота (ГАМК), защищающая мозг от перевозбуждения, норадреналин — регулятор дыхания и артериального давления, дофамин, который управляет нашими движениями и памятью, мелатонин — гормон сна, а главное — серотонин6,11.
А вы знали, что…
90% серотонина — «гормона радости», который регулирует аппетит, все наши эмоции и реакцию на стресс, вырабатывается при участии микробиоты в кишечнике, а не в мозге6,11.
Вернуться к началу
Плохие и хорошие бактерии микробиома кишечника
У здорового взрослого человека микробиом кишечника — это стабильное сообщество микроорганизмов, которые хорошо приспособились к взаимному соседству7. Вся экосистема состоит как из полезных, так и из потенциально вредных обитателей. Некоторые находятся в симбиотических отношениях с «хозяином» — то есть приносят пользу организму человека и сами получают возможность питаться4. Главные участники симбиоза — бифидобактерии и лактобактерии5. Но большая часть представителей микробного сообщества относится к организму «умеренно дружелюбно», поэтому их называют условно-патогенными4. Яркие представители этой «касты» — бактероидеты и фирмикуты, которые составляют более 90% кишечных микробов6. Эти бактерии при удобном для них случае могут вызвать болезнь4.
В кишечнике здорового человека есть три группы микроогранизмов5:
- Постоянные обитатели (90%)5 — эта группа включает бактерии, предпочитающие безвоздушные условия, такие как бифидобактерии, бактероиды, непатогенные клостридии13. Состав постоянных обитателей может отличаться у людей, которые живут в разных странах, экологических условиях или питаются по-разному7.
- Непостоянные (10%) — дополнительная группа бактерий, в которую входят лактобактерии, кишечная палочка, энтерококки и другие.
- Остаточные, или «случайные» (менее 1%) — эти микроорганизмы настроены агрессивно по отношению к человеку. Разные стафилококки, клостридии и другие патогены недолго задерживаются в кишечнике при условии, что состав микробиоты в норме.
Если постоянных полезных бактерий становится меньше, а условно-патогенных — больше, развивается состояние, известное как дисбактериоз5.
Вернуться к началу
Нарушение микробиома кишечника
Поскольку микробиота влияет на весь организм, то дисбактериоз может способствовать развитию разных заболеваний. С другой стороны, хронические заболевания могут привести к дисбактериозу2.
На заметку!
Более правильным считается термин «дисбиоз», потому что микробиота включает не только бактерии, но и другие микроорганизмы. Тем не менее, дисбактериоз остается более привычным понятием для многих.
Баланс микробиома кишечника часто нарушается из-за1,5:
- приема лекарств, особенно антибиотиков, гормонов, препаратов для химиотерапии;
- операций на органах пищеварения;
- перенесенных кишечных инфекций;
- неправильного питания, например, дефицита пищевых волокон;
- стрессов;
- пищевой аллергии.
Как проявляется дисбаланс микробиома кишечника?
Характерных только для дисбактериоза (дисбиоза) проявлений нет, но раньше всех о проблемах с микробиотой сигнализирует кишечник — вздутием, распиранием, урчанием, болью в животе, запором или диареей8. Поэтому если вы заметили у себя эти симптомы, обратитесь к врачу-терапевту или гастроэнтерологу — он поможет разобраться в симптомах и назначит лечение.
У людей с нарушенной микробиотой врачи нередко обнаруживают синдром раздраженного кишечника (СРК), проще говоря, «медвежий стул». Оказывается, при СРК бактерии из толстой кишки «мигрируют» в тонкую, где в норме очень мало микроорганизмов2,20. На этом фоне нарушается двигательная активность тонкой кишки и ее взаимосвязь с нервной системой. При нервном напряжении из-за чрезмерно активного сокращения тонкой кишки пища удаляется из нее быстрее, чем обычно. А плохо переваренные пищевые массы вызывают диарею при СРК13.
А вы знали, что…
Из-за нарушения микробиоты мы можем чувствовать постоянную усталость, набирать лишний вес и даже испытывать проблемы с памятью8.
Вернуться к началу
Проверка микробиома
Диагностировать дисбактериоз только по симптомам — неверно. Еще более ошибочно — начинать восстанавливать микробиом, не зная, каких бактерий в нем стало больше, а каких меньше5.
Дисбактериоз — это диагноз, который нужно подтвердить лабораторными анализами. Для оценки микробиома кишечника врачи используют5:
- Исследование кала (бактериологическое) — чтобы определить количество разных микроорганизмов.
- Дыхательные тесты — определяют обнаружить продукты жизнедеятельности бактерий в выдыхаемом воздухе.
- Эндоскопию — для сбора материала из тонкой кишки с помощью тонкой трубки с камерой (эндоскопа).
Разобраться в такой разной и объемной информации — непростая задача6, поэтому, какой метод диагностики необходим, определит врач.
Восстановление микробиома кишечника
При дисбактериозе нужен комплексный подход. Выбор схемы лечения зависит от степени нарушения, состава бактерий и беспокоящих симптомов5. Как правило, начинают с диеты.
Какое питание может улучшить микробиом кишечника ?
От диеты зависит, какие виды микроорганизмов живут в толстой кишке. Правильное питание создает уникальный микробиом каждого человека3 и поддерживает его здоровый состав.
Чтобы несильно «подкармливать» вредные бактерии, желательно есть меньше мяса и жирной пищи. Этот «западный» тип питания нарушает защитный барьер толстой кишки, а бактерии «переключаются» на питание белками. Но при этом они выделяют в ответ не полезные организму компоненты, а токсины7.
Среди нежелательных диет также самые «модные»3,10,12:
- веганство;
- сыроедение;
- безглютеновые;
- диеты с низким содержанием FODMAPs (для профилактики обострения синдрома раздраженного кишечника).
Обсудите диету с врачом. Если из-за каких-то продуктов у вас начинается понос, не спешите полностью исключать их из своего рациона. В вашем меню обязательно должны быть фрукты, овощи, бобовые, грибы, фисташки, цельное молоко и йогурты, выпечку из пшеничной и ржаной муки10. Они содержат много клетчатки, или пищевых волокон, которые улучшают состав микробиома кишечника3.
Как улучшить состав микробиома пре- и пробиотиками?
Если изменений рациона оказалось недостаточным, врачи рекомендуют принимать, пробиотики и/или пребиотики8:
- Пробиотики — это живые бактерии, которые заселяют кишечник и улучшают здоровье организма-хозяина8. Пробиотики включают в продукты питания, пищевые добавки или принимают в виде лекарств. Большинство пробиотических добавок готовят на основе лактобактерий и бифидобактерий3.
- Пребиотики — это вещества, которые плохо перевариваются пищеварительными ферментами, но зато «кормят» разные группы полезных бактерий. В отличие от пробиотиков, большинство пребиотиков — это пищевые добавки в крупах, шоколаде, макаронах, вермишели и молочных продуктах8.
Лечение дисбаланса микробиома кишечника
Баланс микробиома восстанавливается не сразу, нужно время, чтобы полезные бактерии размножились и перестали быть в меньшинстве. В течение этого времени человека могут продолжать беспокоить неприятные симптомы дисбактериоза. Тогда врач рекомендует симптоматическое лечение разными препаратами5.
Для облегчения симптомов диареи используется лоперамид — действующее вещество Имодиум® Экспресс. Связываясь с особыми рецепторами в стенке кишечника, препарат снижает перистальтику9. В результате работа кишечника замедляется, и диарея уменьшается. Еще одно действие лоперамида связано с повышением тонуса анального сфинктера — благодаря этому предупреждается недержание каловых масс. Имодиум® Экспресс — таблетки-лиофилизат, которые быстро растворяются на языке и не требуют запивания водой9. Если вы заметили в кале кровь или у вас понос на фоне высокой температуры — не принимайте лоперамид до консультации с врачом9.
Целый мир уникальных микроорганизмов создает микробиом взрослого человека, который стоит на страже здоровья организма-хозяина от рождения до старости7. Правильное питание и образ жизни помогают поддерживать здоровье микробиома кишечника3. Но разные расстройства, например диарея, могут сигнализировать о нарушении «идиллии» между микробиотой и вашим организмом. Подробнее об этих сигналах читайте в статье «Дисбактериоз кишечника».
Информация в данной статье носит справочный характер и не заменяет профессиональной консультации врача. Для постановки диагноза и назначения лечения обратитесь к квалифицированному специалисту.
Вернуться к началу