: 20 авг 2019 , История «певчей» хромосомы , том 83,
№3
Молекулярное скорочтение: как белки ищут мишени в ДНК
Иногда привычные сравнения и аналогии, которые призваны простым языком объяснять научные концепции, упускают очень важные вещи. Кто не слышал сравнения ДНК – главного носителя генетической информации – с текстом, записанным с использованием четырех букв – отдельных нуклеотидов? Но, если вдуматься, человек читает рассказ, написанный в книге, совершенно не так, как клетка читает ДНК. Молекулярные машины – белки, которые ищут и узнают отдельные слова в длинной нуклеотидной цепи, воспринимают ДНК не как текст, а как длинную молекулу со всеми ее химическими особенностями. Да и найти нужное место в этом тексте непросто: геном человека примерно в 2000 раз длиннее «Войны и мира» и запакован в объем, сравнимый с пересечением двух паутинок. Но любая ошибка в поиске слов в ДНК может привести к гибели клетки или к превращению нормальной клетки в раковую. Как же решается проблема чтения в микромире?
Представьте себе, что глаза у вас завязаны, а в руках – четки. Одна бусина на четках своей формой не похожа на остальные, и задача – найти ее на ощупь. Вроде бы несложно, да? Ладно, пусть есть четыре возможных формы бусин, а непохожая отличается от всех них. Сложнее, но выполнимо.
Приблизим задачу к реальности. Бусин – шесть миллиардов, непохожие попадаются один раз на миллион. А кому сейчас легко?
ДНК: текст или нет?
Сегодня трудно найти человека, который бы не слышал о том, что ДНК – это двойная спираль, которая состоит из «букв»-нуклеотидов, соответствующих друг другу по принципу комплементарности: напротив аденина (А) всегда находится тимин (Т), напротив гуанина (Г) – цитозин (Ц). Почти каждый, кто пишет о ДНК, сравнивает ее с текстом, в котором буквы образуют слова-кодоны и предложения-гены, и говорит о чтении этого текста клетками. Такое сравнение очень логично, оно породило целую область наук о живом – биоинформатику, где основные объекты изучения представляют собой последовательности символов в длинных цепочках биологических молекул, будь то ДНК, РНК или белки. И в то же время уподобление биологической последовательности тексту – очень сильное упрощение, крайне вредное, если мы пытаемся понять, как же этот текст читается.
Вот как вы читаете эту статью? Первый порыв сказать «читаем по буквам, потом складываем слова» следует сразу подавить: так учатся грамоте дошколята, но взрослые при беглом чтении охватывают сразу слова, а некоторые – даже словосочетания или большие куски текста. Более того, в нашем мозгу нет специальных механизмов для узнавания букв или слов – мы читаем, опираясь на общую систему распознавания изображений, состоящих из контрастных вертикальных или горизонтальных полос.

За начало границы муниципального района Волжский Самарской области принята точка, расположенная в юго-западном углу лесного квартала 21 Советского лесничества Кинельского лесхоза на развилке лесных дорог (точка 1 – пересечение границ муниципальных районов Волжский, Красноярский и Кинельский Самарской области), далее граница проходит смежно границе муниципального района Кинельский Самарской области в восточном направлении по южным границам лесных кварталов 21, 22 Советского лесничества Кинельского лесхоза до юго-восточного угла квартала 22 Советского лесничества Кинельского лесхоза (точка 12), в южном направлении на расстоянии 360 м, пересекая автодорогу, идущую от поселка городского типа Новосемейкино муниципального района Красноярский Самарской области до кольца автодороги, идущей от города Самары до города Отрадного, далее в западном направлении по северной границе лесного квартала 99 Советского лесничества Кинельского лесхоза, в южном направлении по западным границам лесных кварталов 62, 32, 33 и 34 Советского лесничества Кинельского лесхоза, в восточном направлении по южной границе лесного квартала 34 Советского лесничества Кинельского лесхоза до юго-восточной границы этого квартала (точка 51), в юго-восточном направлении по тальвегу оврага Ближний до реки Падовка (точка 406), меняя направление с юго-восточного на юго-западное по середине реки Падовка на расстоянии 3,2 км (точка 599), в южном направлении по территории дачного массива восточнее границы карьера по добыче щебня и поселка Спутник до пересечения с автодорогой, идущей от города Самары до города Отрадного (точка 605), в западном направлении по северной границе полосы отвода (25 м) этой автодороги на расстоянии 63 м (точка 606), в южном направлении по территории дачных участков, примыкающих к поселку городского типа Смышляевка, пересекая Куйбышевскую железную дорогу на участке от станции Безымянка до станции Кинель, до южной границы полосы отвода этой железной дороги (точка 628), в северо-западном направлении по южной границе этой железной доqоги на расстоянии 590 м (точка 629), в юго-западном направлении по земляной дамбе до поворота ее на юго-восток (точка 683), в юго-восточном направлении по восточной стороне этой дамбы на расстоянии 680 м и в этом же направлении по озеру, заболоченному лугу до старицы реки Самара (точка 780), в северо-восточном направлении по этой старице на расстоянии 1,2 км (точка 804), в юго-восточном направлении на расстоянии 320 м до середины реки Самара (точка 805), в северо-восточном направлении…
Также и клетка не может «видеть» буквы А, Г, Т и Ц. С точки зрения молекул, ДНК – это не последовательность букв, а длинный тонкий цилиндр, по которому размазаны электроны: тут погуще, там пореже, а внутри этих отрицательно заряженных электронных облаков спрятаны гораздо меньшие по размеру, но намного более тяжелые положительные заряды – атомные ядра. Более того, буквы влияют друг на друга: в последовательности …ГГГГГ… центральный гуанин по своим химическим свойствам будет заметно отличаться от крайних, хотя, конечно, от оснований другой природы он будет отличаться еще больше. Добавьте сюда еще то, что в микромире нет покоя, и наш цилиндр постоянно бомбардируется другими частицами – в основном, конечно, молекулами воды (человек по весу на 60 % состоит из воды), и что в клетке ДНК вся окружена положительно заряженными ионами и тесно запакована специальными белками.
Так что вы перебираете четки с шестью миллиардами бусин, этих бусин четыре вида, но каждая хоть чуть-чуть да не похожа на остальные; эти четки запутаны, на них местами висят куски пластилина и бутылки, и во всем этом резвится стая кошек. Да, и все это плотно запихано в закрытую комнату, и вы сами внутри этой кучи. Цель прежняя – найти бусину, одну на миллион, которая ну совсем не похожа на другие. Добро пожаловать в настоящий мир молекулярных машин!
Что и зачем искать
Такая задача – найти редкую мишень на фоне огромного избытка другой, нецелевой ДНК – стоит перед очень многими белками в клетке. Все эти белки можно поделить на две большие группы по типу узнаваемых мишеней, потому что для поиска они используют общую стратегию, а вот узнавание идет немного по-разному.

– Уподобление биологической последовательности тексту – очень сильное упрощение, крайне вредное, если мы пытаемся понять, как же этот екст читается. Она скорее похожа на запутанные четки из миллиардов бусин, 
Во-первых, есть мишени, которые представляют собой какую-то конкретную последовательность нуклеотидов, «слово», обычно длиной от 4 до 20—30 букв. Белки, узнающие последовательности, чаще всего принимают участие в регуляции активности генов: связываясь со своими мишенями, они либо помогают другим белкам садиться поблизости от них на ДНК и запускать транскрипцию (копирование ДНК в РНК), либо, наоборот, ее разными способами подавляют. Особняком в этой группе стоят сайт-специфичные эндонуклеазы – белки, расщепляющие ДНК в узнаваемых ими последовательностях. Многие такие белки – рестриктазы – участвуют в защите бактерий от заражения вирусами и уже почти полвека применяются во всех молекулярно-биологических лабораториях для разных манипуляций с ДНК; другие нашли применение в современных технологиях геномного редактирования.
Во вторую группу белков входят те, которые должны узнавать в составе нормальной ДНК то, что на нее не похоже. Прежде всего это повреждения ДНК. Они постоянно возникают под действием вредных факторов внешней среды (радиация, ультрафиолет, химические вещества), но гораздо более опасны обыкновенная вода и кислород, которые на самом деле представляют собой довольно-таки реакционноспособные соединения. Ежедневно в каждой нашей клетке появляются сотни тысяч повреждений, и если их не исправлять, клетка либо погибнет, либо, что еще хуже, будет накапливать мутации, которые рано или поздно приведут к ее превращению в раковую.
Поэтому у всех живых организмов, от бактерий до человека, есть несколько систем репарации ДНК, которые заняты обнаружением и устранением повреждений. Занятые в них белки можно сравнить с путевыми обходчиками на железной дороге, которые постоянно патрулируют пути, проверяя, не случилась ли где поломка. Кроме белков репарации, есть и другие, задача которых отличить «стандартную» ДНК от «нестандартной». Какие-то могут, например, узнавать концы хромосом, другие – квадруплексы (структуры из четырех цепей), третьи – крестообразные структуры, в которые иногда укладываются отдельные участки ДНК.
Как правильно читать законы: 1D против 3D
Давайте теперь опять подумаем о ДНК, как о тексте. И посмотрим на начало самого длинного предложения в русском языке, которое можно найти в законе Самарской области «Об установлении границ муниципального района Волжский Самарской области». Полностью оно состоит из 61 061 знака (не считая пробелов) и в 4,4 раза длиннее всей этой статьи. Что в нем не так, где опечатка? Быстрее!
Смысл этого упражнения в том, чтобы искать, а не в том, чтобы найти. Те, кто искал, но не нашел, могут посмотреть ответ в конце статьи. А теперь подумайте, как вы искали. Кто-то, возможно, начал чтение с самого начала и внимательно, последовательно пробегал глазами каждую строчку слева направо. Кто-то (таких, скорее, большинство), наоборот, побегал глазами туда-сюда по одному фрагменту текста, ничего подозрительного не заметил, перескочил на другой, на третий…
Точно так же перемещаются белки по ДНК. Некоторые из них движутся только в одном направлении, просматривая все на своем пути. Так вы гарантированно обнаружите то, что ищете, но вот беда – в микромире такой путь довольно энергозатратный. Чтобы необратимо двигаться всегда в одном направлении, нужен расход энергии, второй закон термодинамики неумолим. Поэтому такие белки имеют специальные «моторчики», гидролизующие молекулы аденозинтрифосфата (АТФ), служащие топливом для очень многих клеточных процессов. На один шаг по ДНК (то есть перемещению на один нуклеотид) обычно требуется 2–3 молекулы АТФ.
Но гораздо большее число белков используют другую стратегию. Они движутся по ДНК исключительно за счет теплового движения. Такой процесс иногда называют «одномерной диффузией», чтобы отличить его от обычного броуновского движения – ненаправленного теплового движения частиц или крупных молекул в трех измерениях. Такое движение само по себе быстрее и не требует расхода энергии, но есть и проблема: так можно вообще ничего не найти.
Впервые на проблему ненаправленного поиска обратили внимание в 1981 г. американские биофизики П. фон Гиппель, О. Берг и Р. Уинтер: три их статьи, идущие подряд в одном из выпусков журнала Biochemistry, до сих пор цитируются всеми учеными, работающими в этой области. Собственно, затруднение состоит в том, что если белок гуляет по ДНК случайным образом, каждый раз делая с равной вероятностью шаг налево или направо (физики называют такие процессы «походкой пьяницы»), то в среднем через N шагов он окажется в √N нуклеотидах от начальной позиции. С такой скоростью одной молекуле белка, в зависимости от времени на один шаг, потребуется от 10 дней до 3 лет, чтобы обозреть весь геном кишечной палочки. Учитывая, что бактерия в оптимальных условиях делится раз в 20–30 минут, этого явно недостаточно для поддержания ее жизни. Конечно, в клетке не одна молекула каждого белка, но интересующие нас белки не относятся к числу самых многочисленных. Так, молекул Lac-репрессора – регулятора активности бактериальных генов, связанных с использованием лактозы, – в клетке кишечной палочки всего около десяти. А если искать мишень исключительно в трех измерениях – связывать ДНК в случайном месте, выпускать ее, если это место оказалось не мишенью, и связывать в другом случайном месте – времени на это уйдет еще больше.
Фон Гиппель и его коллеги разработали математическую модель, где показали, что существует оптимальное сочетание одномерного и трехмерного ненаправленного поиска, позволяющее находить мишень за наименьшее время, и подтвердили ее, исследуя тот самый Lac-репрессор. Белок связывает ДНК в случайном месте, ненаправленно сканирует ее какое-то время, а потом соскакивает и вновь связывается в совершенно другом месте, совсем как некоторые читатели искали опечатку в нашем тексте. За прошедшие годы такой способ поиска мишеней в ДНК был продемонстрирован для нескольких десятков самых разных белков. И не только в пробирке: например, удалось показать, что бактериальные белки репарации, которые ищут повреждения, вызванные облучением ультрафиолетом, таким же образом работают в живой клетке, несмотря на все помехи, вызванные внутриклеточным окружением ДНК.
В Москву? В Моркву? В М¤скву?
Итак, мы более-менее понимаем, как в ДНК можно что-то искать. А как это найти? Это совершенно другой вопрос: как белок понимает, что то, с чем он в данный момент связан, мишень? Она ведь часто очень мало отличается от не-мишени: поменяйте слово ААТТГТГАГЦГГАТААЦААТТ на ААТТГТГЦГЦГГАТААЦААТТ, и Lac-репрессор с такой ДНК перестанет связываться вовсе. Белки, узнающие последовательности, и белки, узнающие повреждения ДНК, решают эту задачу немного по-разному, хотя, если вдуматься, общие принципы все равно есть.

– «Узнавание» идет либо за счет образования множества слабых водородных связей, либо путем механического воздействия на ДНК, также с образованием нескольких ромежуточных связей. В этом смысле белок похож на незрячего человека, перебирающего четки и 
Белки, которые узнают последовательность, делают это за счет образования множества слабых водородных связей с несколькими критически важными основаниями ДНК в этой последовательности. Их движение по ДНК похоже на скольжение по смазке: между поверхностями белка и ДНК находится слой молекул воды, которые также образуют множество связей с какими угодно молекулярными остатками. Если сам белок нужных связей с ДНК образовать не может, он продолжает скользить туда-сюда. Но стоит нащупать две-три специфичные позиции, как происходит резкое торможение: вода изгоняется из области контакта, и есть время, чтобы попытаться закончить образование всех необходимых связей. Не получилось? Значит, перед нами не мишень; вода просачивается обратно и поиск продолжается.
Белки, патрулирующие ДНК в поисках повреждений, решают более сложную задачу. Если нужно отличить, например, поврежденное основание от нормального – букву Ä от буквы A – есть не так много вариантов образования связей, которые были бы специфичны именно для неправильной буквы. Поэтому здесь приходится вспоминать о том, что ДНК не текст, а молекула. Например, очень часто поврежденные основания образуют не такие прочные связи со своими соседями, как нормальные. Многие белки репарации для узнавания повреждений сильно сгибают ДНК и пытаются втиснуть внутрь двойной спирали свои аминокислотные остатки. Такое внутреннее напряжение в молекуле ДНК находит выход в самом слабом месте – поврежденное основание выворачивается из ДНК наружу и попадает в активный центр белка, где дальше его, например, можно «отрезать» от остова ДНК, а потом другие белки репарации заменят его на правильное основание. По пути в активный центр белок еще, как правило, образует несколько промежуточных связей с выворачиваемым основанием, проверяя, действительно ли оно повреждено, словно перебирающий четки человек тщательно ощупывает подозрительную бусину с разных сторон.
Кстати, узнавание последовательностей тоже иногда требует механического воздействия на ДНК или хотя бы учета ее формы. Например, некоторые последовательности, узнаваемые факторами транскрипции, могут иметь уже существующий небольшой изгиб или же изменять свою структуру при связывании узнающего белка. Так что и тут чтение ДНК отличается от простого чтения букв в тексте.
Цена ошибки
Конечно, в природе нет совершенства, и наши белки могут делать ошибки. Важно понимать, что ошибки при узнавании могут быть двух сортов: принять за мишень то, что мишенью не является, и, наоборот, не узнать мишень. Ошибки второго рода не очень страшны как раз из-за механизма ненаправленного поиска: в самом деле, если на каком-то шаге белок не узнал свою мишень, с вероятностью ½ он к ней вернется через два шага поиска и получит еще один шанс. И так неоднократно.
Гораздо хуже ошибки первого рода: если расщепить ДНК или запустить работу гена не там, где надо, это может иметь печальные последствия для клетки. Поэтому эволюция белков, узнающих ДНК, шла по пути уменьшения вероятности ошибок первого рода, а на ошибки второго рода особого внимания не обращала. Это привело к тому, что, например, белки репарации, как ни странно, узнают повреждения в ДНК очень неэффективно – в лучшем случае, к узнаванию приводит каждая вторая встреча с повреждением, а гораздо чаще – каждая десятая-двадцатая. Это дает ученым надежду на то, что такие белки можно улучшить разными генноинженерными путями, хотя бы для использования в качестве лабораторных инструментов.
Зато отличают поврежденную ДНК от нормальной системы репарации сверхнадежно: даже при избытке нормальных оснований в миллионы раз они не узнаются ошибочно как повреждения.
Казалось бы, изучение того, как белки движутся по ДНК и узнают разные ее участки, пример чистого научного любопытства, никак не способного помочь в реальной жизни. Однако, как это часто случается в науке, в попытках ответа на совершенно отвлеченные вопросы неожиданно рождается что-то интересное для практики. Недавно, например, выяснилось, что некоторые вирусы – в том числе такие опасные, как вирус оспы, – используют видоизмененный белок репарации урацил-ДНК-гликозилазу в процессе копирования своей ДНК. При этом вирусу нужна именно способность этого фермента скользить по ДНК: белок прикрепляется к вирусному комплексу, который синтезирует ДНК, и помогает ему двигаться, не выпуская цепи. Зацепившись за это наблюдение, сотрудники лаборатории геномной и белковой инженерии Института химической биологии и фундаментальной медицины СО РАН разработали ингибиторы скольжения вирусного белка по ДНК, которые могут дать начало принципиально новому классу противовирусных лекарств.

А вот другой модный сегодня белок – недавно открытая нуклеаза Cas9, которая стала основой большинства современных технологий редактирования генома, – ищет свои мишени, используя только обычную диффузию в трехмерном пространстве. В той же лаборатории сейчас работают над созданием улучшенных вариантов этого белка, способных все-таки передвигаться вдоль ДНК и быстрее находить цель. Знание того, как работают молекулярные машины в деталях, всегда необходимо для осмысленных попыток обратить их на пользу человеку.
Литература
Жарков Д. О. Загадки «ржавой» ДНК // Наука из первых рук. 2006. Т. 12. № 6. С. 24–35.
Жарков Д. О. Часовые генома // Наука из первых рук. 2009. Т. 28. № 4. С. 160–169.
Мечетин Г. В., Жарков Д. О. Механизмы диффузионного поиска специфичных мишеней ДНК-зависимыми белками // Биохимия. 2014. Т. 79. № 6. С. 633–644.
Zharkov D. O., Grollman A. P. The DNA trackwalkers: Principles of lesion search and recognition by DNA glycosylases // Mutat. Res. 2005. V. 577. N. 1–2. P. 24–54.
: 20 авг 2019 , История «певчей» хромосомы , том 83,
№3
И транскрипция, и трансляция относятся к матричным биосинтезам. Матричным биосинтезом называется синтез
биополимеров (нуклеиновых кислот, белков) на матрице — нуклеиновой кислоте ДНК или РНК. Процессы матричного биосинтеза относятся к пластическому обмену: клетка расходует энергию АТФ.
Матричный синтез можно представить как создание копии исходной информации на несколько другом или новом
«генетическом языке». Скоро вы все поймете — мы научимся достраивать по одной цепи ДНК другую, переводить РНК в ДНК
и наоборот, синтезировать белок с иРНК на рибосоме. В данной статье вас ждут подробные примеры решения задач, генетический словарик пригодится — перерисуйте его себе 

Возьмем 3 абстрактных нуклеотида ДНК (триплет) — АТЦ. На иРНК этим нуклеотидам будут соответствовать — УАГ (кодон иРНК).
тРНК, комплементарная иРНК, будет иметь запись — АУЦ (антикодон тРНК). Три нуклеотида в зависимости от своего расположения
будут называться по-разному: триплет, кодон и антикодон. Обратите на это особое внимание.
Репликация ДНК — удвоение, дупликация (лат. replicatio — возобновление, лат. duplicatio — удвоение)
Процесс синтеза дочерней молекулы ДНК по матрице родительской ДНК. Нуклеотиды достраивает фермент ДНК-полимераза по
принципу комплементарности. Переводя действия данного фермента на наш язык, он следует следующему правилу: А (аденин) переводит в Т (тимин), Г (гуанин) — в Ц (цитозин).
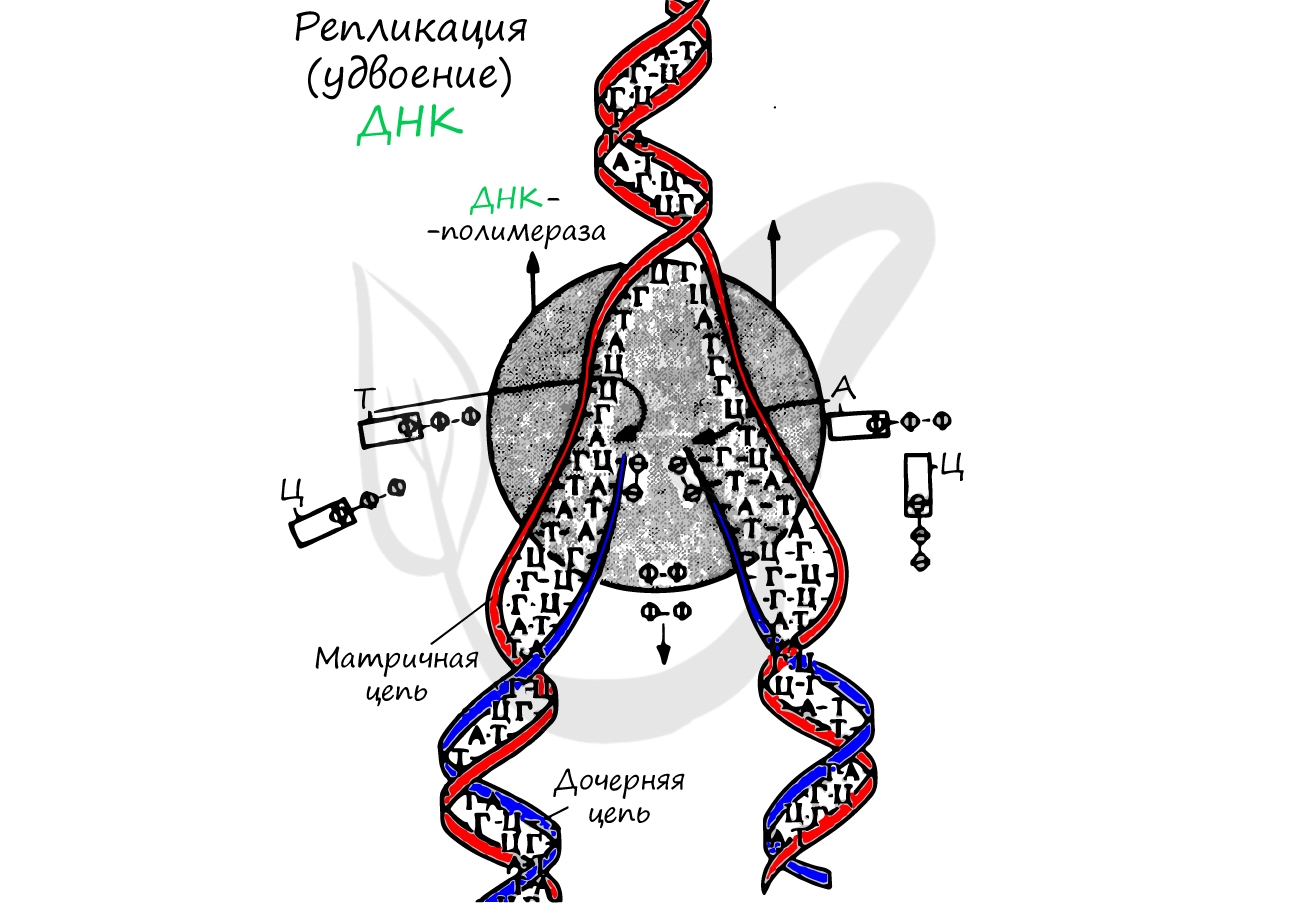
Удвоение ДНК происходит в синтетическом периоде интерфазы. При этом общее число хромосом не меняется, однако каждая из них
содержит к началу деления две молекулы ДНК: это необходимо для равномерного распределения генетического материала между
дочерними клетками.
Транскрипция (лат. transcriptio — переписывание)
Транскрипция представляет собой синтез информационной РНК (иРНК) по матрице ДНК. Несомненно, транскрипция происходит
в соответствии с принципом комплементарности азотистых оснований: А — У, Т — А, Г — Ц, Ц — Г (загляните в «генетический словарик»
выше).
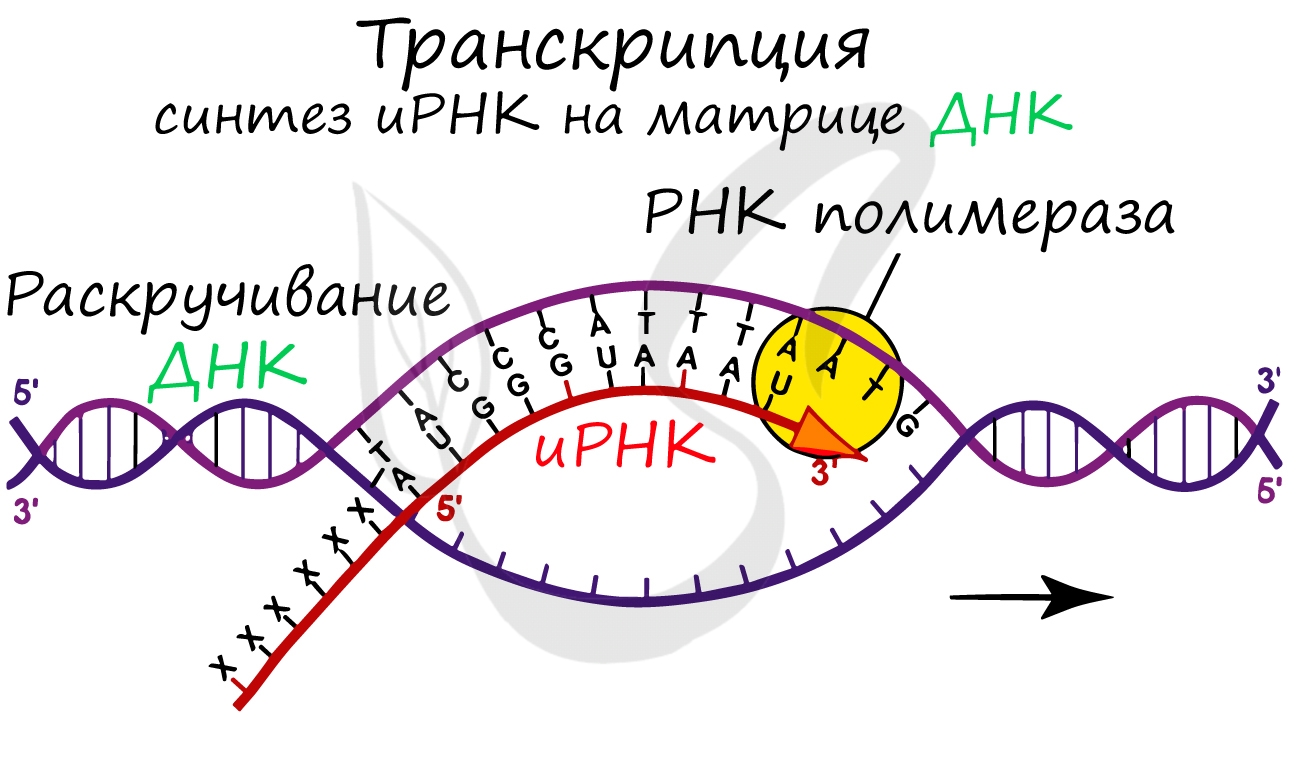
До начала непосредственно транскрипции происходит подготовительный этап: фермент РНК-полимераза узнает особый участок молекулы ДНК — промотор и связывается с ним. После связывания с промотором происходит раскручивание молекулы ДНК, состоящей из двух
цепей: транскрибируемой и смысловой. В процессе транскрипции принимает участие только транскрибируемая цепь ДНК.
Транскрипция осуществляется в несколько этапов:
- Инициация (лат. injicere — вызывать)
- Элонгация (лат. elongare — удлинять)
- Терминация (лат. terminalis — заключительный)
Образуется несколько начальных кодонов иРНК.
Нити ДНК последовательно расплетаются, освобождая место для передвигающейся РНК-полимеразы. Молекула иРНК
быстро растет.
Достигая особого участка цепи ДНК — терминатора, РНК-полимераза получает сигнал к прекращению синтеза иРНК. Транскрипция завершается. Синтезированная иРНК направляется из ядра в цитоплазму.
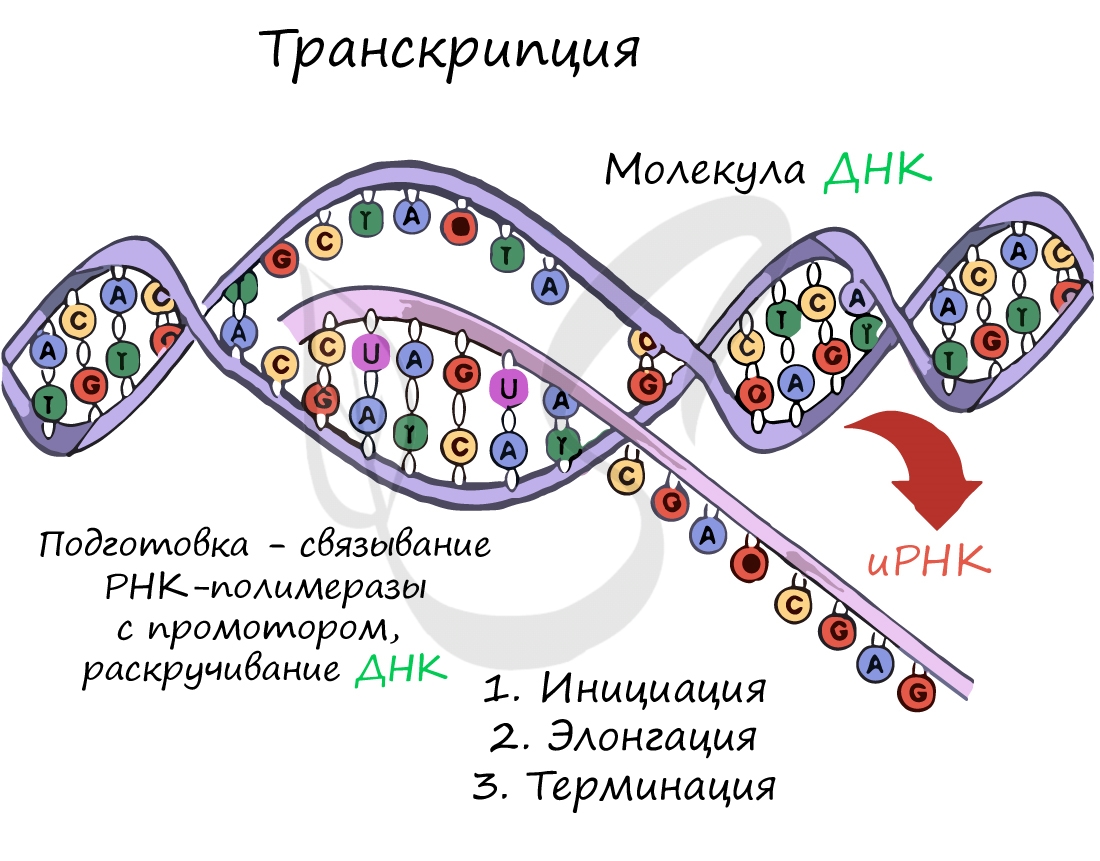
Трансляция (от лат. translatio — перенос, перемещение)
Куда же отправляется новосинтезированная иРНК в процессе транскрипции? На следующую ступень — в процесс трансляции.
Он заключается в синтезе белка на рибосоме по матрице иРНК. Последовательность кодонов иРНК переводится в последовательность
аминокислот.
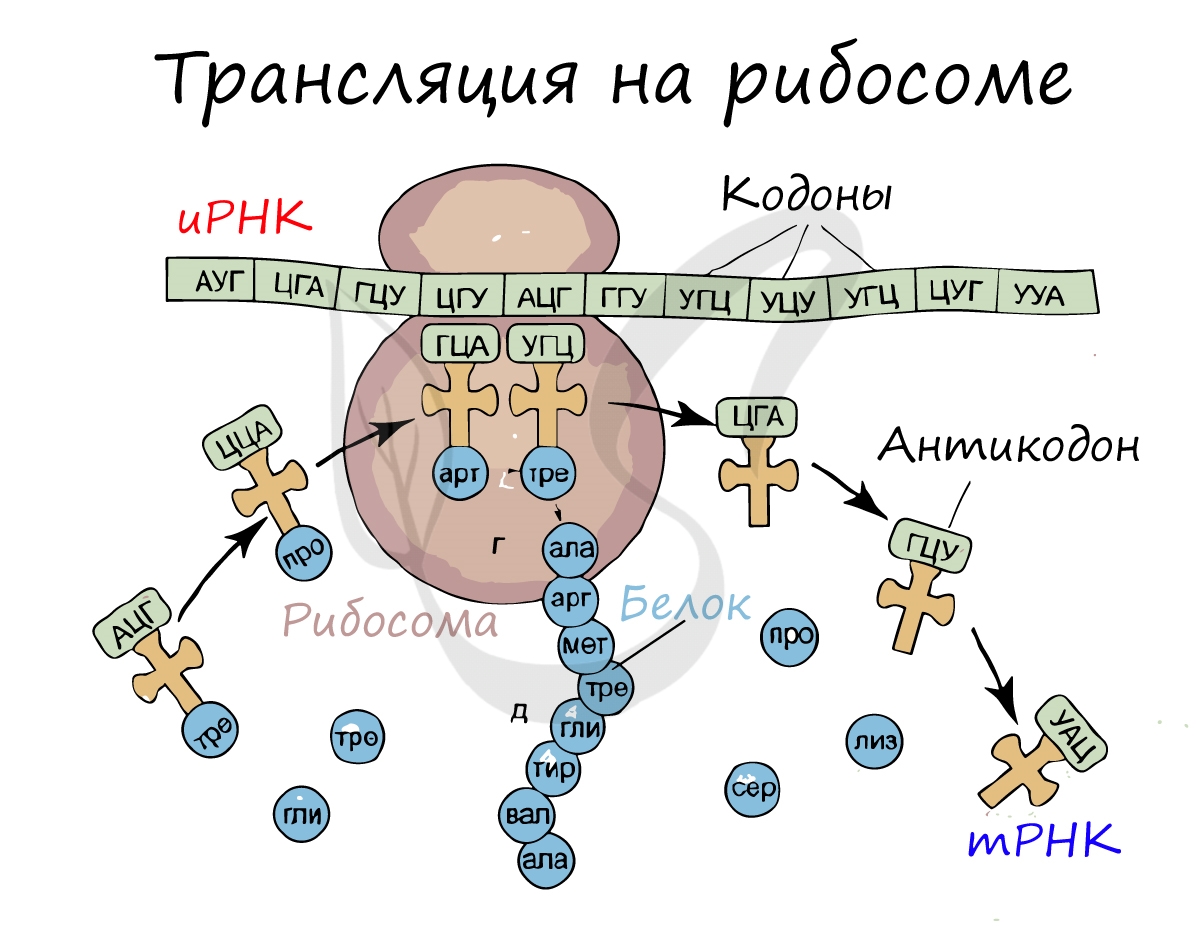
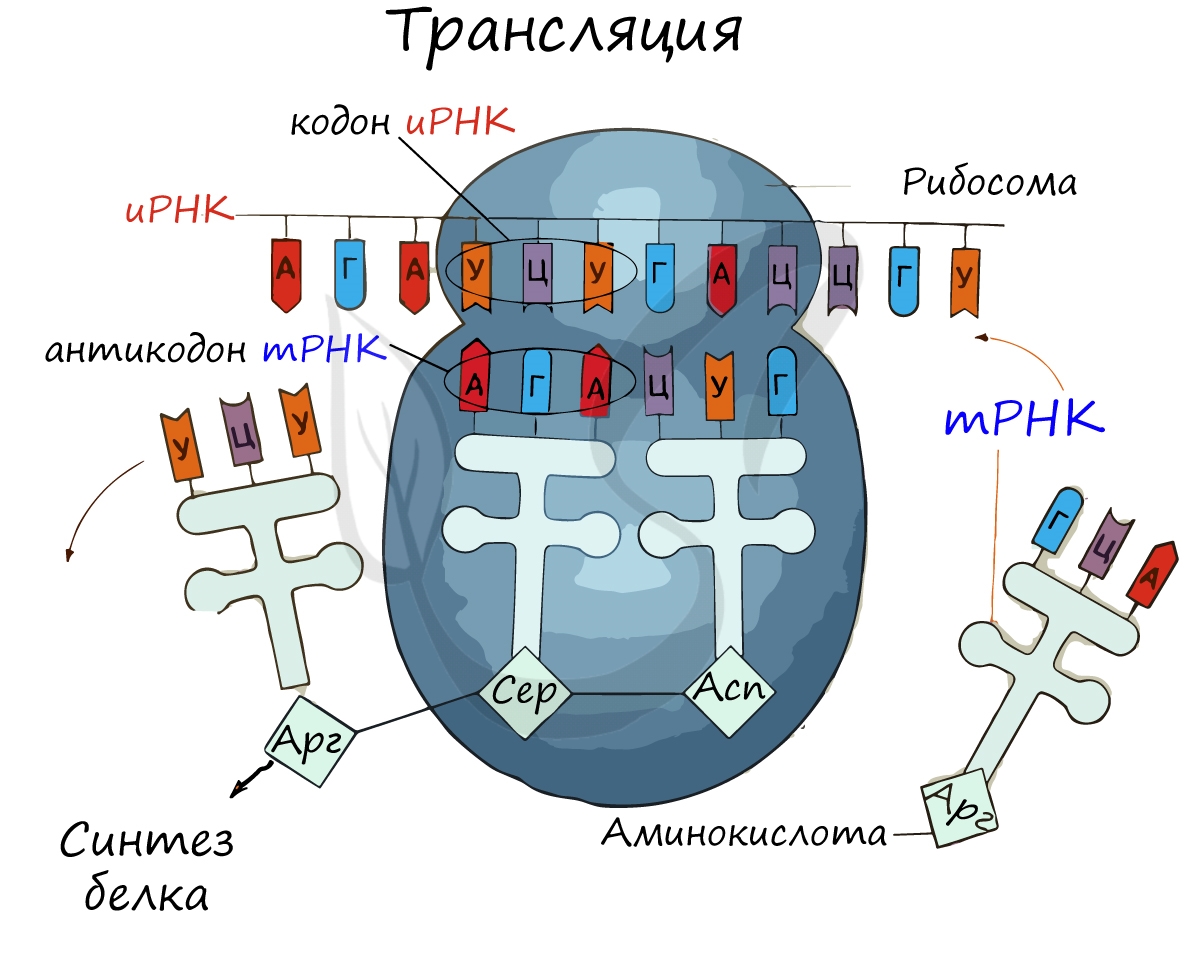
Перед процессом трансляции происходит подготовительный этап, на котором аминокислоты присоединяются к соответствующим молекулам тРНК. Трансляцию можно разделить на несколько стадий:
- Инициация
- Элонгация
- Терминация
Информационная РНК (иРНК, синоним — мРНК (матричная РНК)) присоединяется к рибосоме, состоящей из двух субъединиц.
Замечу, что вне процесса трансляции субъединицы рибосом находятся в разобранном состоянии.
Первый кодон иРНК, старт-кодон, АУГ оказывается в центре рибосомы, после чего тРНК приносит аминокислоту,
соответствующую кодону АУГ — метионин.
Рибосома делает шаг, и иРНК продвигается на один кодон: такое в фазу элонгации происходит десятки тысяч раз.
Молекулы тРНК приносят новые аминокислоты, соответствующие кодонам иРНК. Аминокислоты соединяются друг с другом: между ними образуются пептидные связи, молекула белка растет.
Доставка нужных аминокислот осуществляется благодаря точному соответствию 3 нуклеотидов (кодона) иРНК 3 нуклеотидам (антикодону) тРНК. Язык перевода между иРНК и тРНК выглядит как: А (аденин) — У (урацил), Г (гуанин) — Ц (цитозин).
В основе этого также лежит принцип комплементарности.
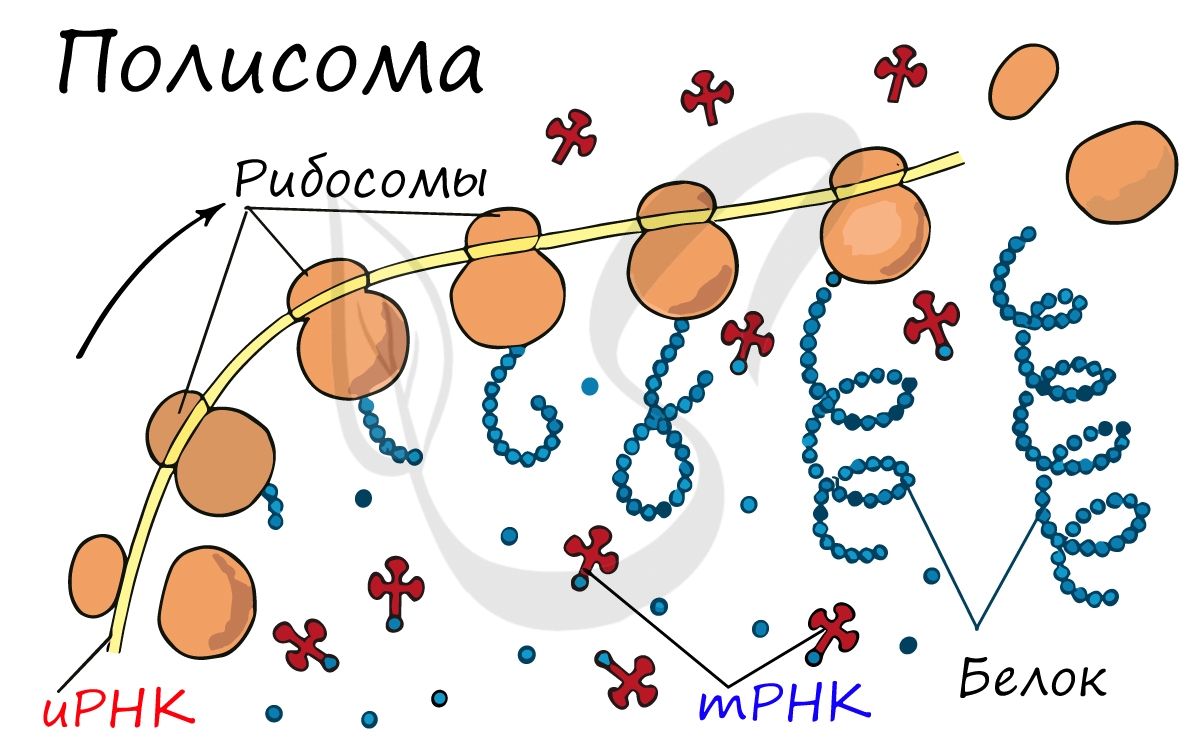
Движение рибосомы вдоль молекулы иРНК называется транслокация. Нередко в клетке множество рибосом садятся на одну молекулу
иРНК одновременно — образующаяся при этом структура называется полирибосома (полисома). В результате происходит одновременный синтез множества одинаковых белков.
Синтез белка — полипептидной цепи из аминокислот — в определенный момент завершатся. Сигналом к этому служит попадание
в центр рибосомы одного из так называемых стоп-кодонов: УАГ, УГА, УАА. Они относятся к нонсенс-кодонам (бессмысленным), которые не кодируют ни одну аминокислоту. Их функция — завершить синтез белка.
Существует специальная таблица для перевода кодонов иРНК в аминокислоты. Пользоваться ей очень просто, если вы запомните, что
кодон состоит из 3 нуклеотидов. Первый нуклеотид берется из левого вертикального столбика, второй — из верхнего горизонтального,
третий — из правого вертикального столбика. На пересечении всех линий, идущих от них, и находится нужная вам аминокислота 
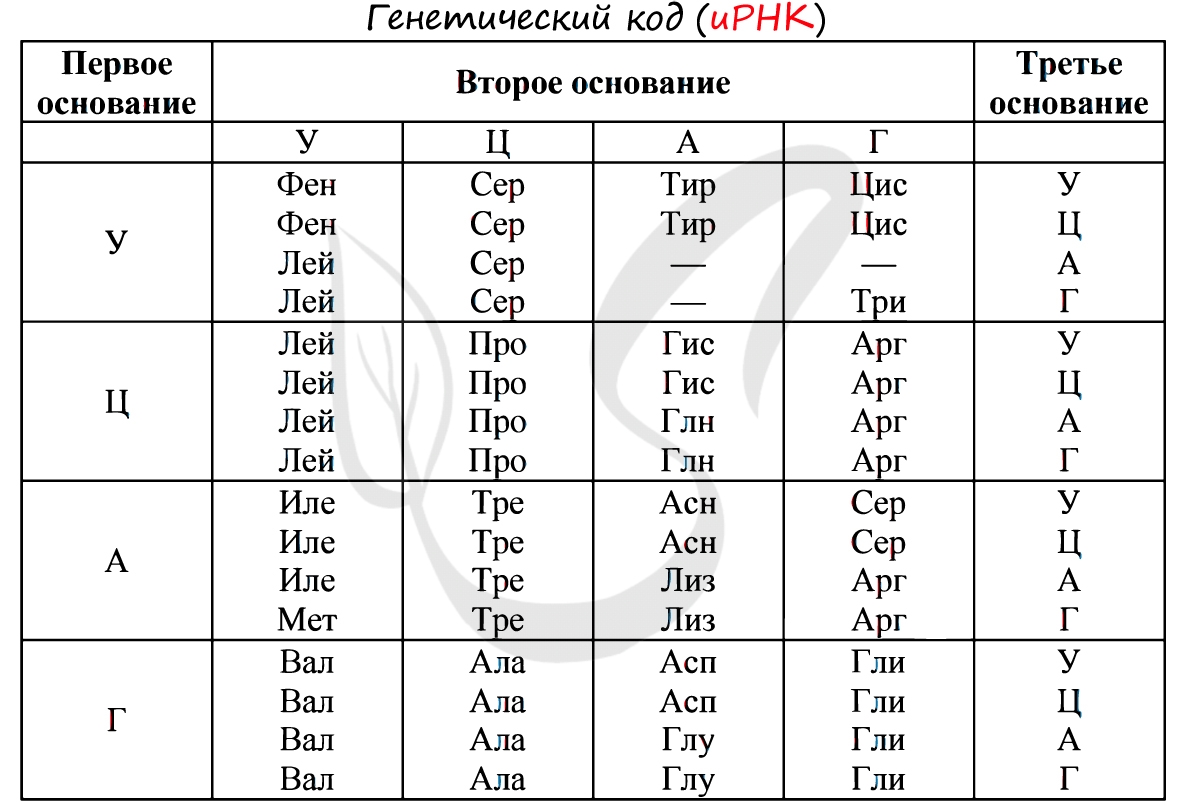
Давайте потренируемся: кодону ЦАЦ соответствует аминокислота Гис, кодону ЦАА — Глн. Попробуйте самостоятельно найти
аминокислоты, которые кодируют кодоны ГЦУ, ААА, УАА.
Кодону ГЦУ соответствует аминокислота — Ала, ААА — Лиз. Напротив кодона УАА в таблице вы должны были обнаружить прочерк:
это один из трех нонсенс-кодонов, завершающих синтез белка.
Примеры решения задачи №1
Без практики теория мертва, так что скорее решим задачи! В первых двух задачах будем пользоваться таблицей генетического кода (по иРНК),
приведенной вверху.
«Фрагмент цепи ДНК имеет следующую последовательность нуклеотидов: ЦГА-ТГГ-ТЦЦ-ГАЦ. Определите последовательность нуклеотидов
во второй цепочке ДНК, последовательность нуклеотидов на иРНК, антикодоны
соответствующих тРНК и аминокислотную последовательность соответствующего фрагмента молекулы белка, используя таблицу генетического кода»

Объяснение:
По принципу комплементарности мы нашли вторую цепочку ДНК: ГЦТ-АЦЦ-АГГ-ЦТГ. Мы использовали следующие правила при нахождении второй нити
ДНК: А-Т, Т-А, Г-Ц, Ц-Г.
Вернемся к первой цепочке, и именно от нее пойдем к иРНК: ГЦУ-АЦЦ-АГГ-ЦУГ. Мы использовали следующие правила при переводе ДНК в иРНК:
А-У, Т-А, Г-Ц, Ц-Г.
Зная последовательность нуклеотидов иРНК, легко найдем тРНК: ЦГА, УГГ, УЦЦ, ГАЦ. Мы использовали следующие правила перевода иРНК в тРНК:
А-У, У-А, Г-Ц, Ц-Г. Обратите внимание, что антикодоны тРНК мы разделяем запятыми, в отличие кодонов иРНК. Это связано с тем, что
тРНК представляют собой отдельные молекулы (в виде клеверного листа), а не линейную структуру (как ДНК, иРНК).
Пример решения задачи №2
«Известно, что все виды РНК синтезируются на ДНК-матрице. Фрагмент цепи ДНК, на которой синтезируется участок центральной петли тРНК, имеет
следующую последовательность нуклеотидов: ТАГ-ЦАА-АЦГ-ГЦТ-АЦЦ. Установите нуклеотидную последовательность участка тРНК, который синтезируется
на данном фрагменте, и аминокислоту, которую будет переносить эта тРНК в процессе биосинтеза белка, если третий триплет соответствует антикодону
тРНК»

Обратите свое пристальное внимание на слова «Известно, что все виды РНК синтезируются на ДНК-матрице. Фрагмент цепи ДНК, на которой
синтезируется участок центральной петли тРНК «. Эта фраза кардинально меняет ход решения задачи: мы получаем право напрямую и сразу
синтезировать с ДНК фрагмент тРНК — другой подход здесь будет считаться ошибкой.
Итак, синтезируем напрямую с ДНК фрагмент молекулы тРНК: АУЦ-ГУУ-УГЦ-ЦГА-УГГ. Это не отдельные молекулы тРНК (как было
в предыдущей задаче), поэтому не следует разделять их запятой — мы записываем их линейно через тире.
Третий триплет ДНК — АЦГ соответствует антикодону тРНК — УГЦ. Однако мы пользуемся таблицей генетического кода по иРНК,
так что переведем антикодон тРНК — УГЦ в кодон иРНК — АЦГ. Теперь очевидно, что аминокислота кодируемая АЦГ — Тре.
Пример решения задачи №3
Длина фрагмента молекулы ДНК составляет 150 нуклеотидов. Найдите число триплетов ДНК, кодонов иРНК, антикодонов тРНК и
аминокислот, соответствующих данному фрагменту. Известно, что аденин составляет 20% в данном фрагменте (двухцепочечной
молекуле ДНК), найдите содержание в процентах остальных нуклеотидов.

Один триплет ДНК состоит из 3 нуклеотидов, следовательно, 150 нуклеотидов составляют 50 триплетов ДНК (150 / 3). Каждый триплет ДНК
соответствует одному кодону иРНК, который в свою очередь соответствует одному антикодону тРНК — так что их тоже по 50.
По правилу Чаргаффа: количество аденина = количеству тимина, цитозина = гуанина. Аденина 20%, значит и тимина также 20%.
100% — (20%+20%) = 60% — столько приходится на оставшиеся цитозин и гуанин. Поскольку их процент содержания равен, то
на каждый приходится по 30%.
Теперь мы украсили теорию практикой. Что может быть лучше при изучении новой темы? 
© Беллевич Юрий Сергеевич 2018-2023
Данная статья написана Беллевичем Юрием Сергеевичем и является его интеллектуальной собственностью. Копирование, распространение
(в том числе путем копирования на другие сайты и ресурсы в Интернете) или любое иное использование информации и объектов
без предварительного согласия правообладателя преследуется по закону. Для получения материалов статьи и разрешения их использования,
обратитесь, пожалуйста, к Беллевичу Юрию.
Секвенирование — методы расшифровки нуклеотидной последовательности ДНК. Сообщение 1
Костюк С.А.
Белорусская медицинская академия последипломного образования, Минск
Kostiuk S.A.
Belarusian Medical Academy of Post-Graduate Education, Minsk
Sequencing — methods of DNA nucleotide sequence decryption. Report 1
Резюме. Описаны методы, позволяющие идентифицировать нуклеотидную последовательность ДНК. Подробно рассмотрены свойства флуо-рофоров и полимераз, используемых в секвенировании ДНК, представлены особенности секвенирующего гель-электрофореза. Ключевые слова: секвенирование, ферментативный метод, метод химической деградации, флуорофор, секвенатор.
Медицинские новости. — 2017. — №6. — С. 18-21. Summary. Methods of DNA nucleotide sequence identification are described in this article. Characteristics of phosphors and polymerases using in DNA sequencing are contemplated in detail, gel electrophoresis peculiarities are described. Keywords: sequencing, enzymatic method, method of chemical degradation, fluorophor, sequenator. Meditsinskie novosti. — 2017. — N6. — P. 18-21.
В 50-е годы прошлого века были разработаны методы, позволяющие определять последовательность аминокислот в полипептидной цепи. Теоретически это несложно, поскольку все аминокислоты, встречающиеся в природных белках, имеют разные свойства. После расшифровки генетического кода появилась возможность восстанавливать нуклеотидную последовательность транскрибируемой ДНК по аминокислотной последовательности соответствующего белка. Однако генетический код является вырожденным, следовательно, первичная структура ДНК, полученная на основе анализа последовательности аминокислот, не является однозначной [5, 14, 16].
Безусловно, определение нуклео-тидной последовательности ДНК крайне важно для множества фундаментальных и прикладных задач. Особое место оно занимает в науке: для анализа результатов секвенирования геномов была, фактически, создана новая наука — биоинформатика. Секвенирование сейчас используют молекулярные биологи, генетики, биохимики, микробиологи, ботаники, зоологи, и, конечно же, эволюционисты: практически вся современная систематика основана на его результатах. Секвенирование широко применяется в медицине как метод поиска наследственных заболеваний и изучения инфекций [3, 8].
В настоящее время определение точной нуклеотидной последовательности любого сегмента ДНК умеренной длины — вполне разрешимая задача. Уже определены последовательности нескольких сотен генов про- и эукариот. Зная последовательность гена и генетический
код, легко определить аминокислотную последовательность кодируемого им белка. Раньше для определения структуры белка приходилось делать тщательный и весьма трудоемкий анализ выделенного и очищенного белка, в то время как сейчас бывает проще определить структуру белка через нуклеотидную последовательность, чем с помощью прямого секвенирования. Если секвенирование белка занимает месяцы и даже годы, то ДНК удается секвенировать за несколько недель. Так в 2000 году был секвенирован геном человека, однако в данном случае речь идет только об установлении последовательности нукле-отидов, так как генетическая структура и функции отдельных участков генома еще не идентифицированы, что представляет собой более сложную задачу: прочесть -не значит понять [2, 9, 11, 15].
С чего все начиналось?
К концу 60-х годов прошлого века Ф. Сэнгером был разработан метод секвенирования РНК, получаемой с ДНК-матрицы при помощи РНК-полимеразы [13]. Применив этот способ, Ш. Вейссман и У. Фирс смогли к концу 1976 года определить последовательность более половины молекулы ДНК SV40, длина которой превышает 5200 нуклеотидных пар [1, 2]. Следующим шагом должна была стать разработка методов прямого секвенирования ДНК.
Первым методом прямого ферментативного секвенирования ДНК стал метод, предложенный Ф. Сэнгером и Д. Коулсоном в 1975 году («плюс-минус» метод). В качестве матрицы в реакции полимеразного копирования использовался одноцепочеч-
ный фрагмент ДНК, в качестве прайме-ров — синтетические олигонуклеотиды или природные субфрагменты, получаемые при гидролизе рестрикционными эндону-клеазами, а в качестве фермента — фрагмент Кленова ДНК полимеразы I (Poll) из Escherichia coli[14].
Метод включал два этапа. Сначала в ограниченных условиях проводили по-лимеразную реакцию в присутствии всех четырех типов дезоксинуклеозидтрифос-фатов (dNTP) (один из них был мечен по а-положению фосфата), получая на выходе набор продуктов неполного копирования матричного фрагмента. Смесь очищали от несвязавшихся dNTP и делили на восемь частей. После чего в «плюс»-системе проводили четыре реакции в присутствии каждого из четырех типов нуклеотидов, в «минус»-системе — в отсутствии каждого из них. В результате в «минус»-системе терминация происходила перед dNTP данного типа, а в «плюс»-системе — после него (рис. 1).
Полученные таким образом восемь образцов разделяли с помощью электрофореза, «считывали» сигнал и определяли последовательность исходной ДНК. Этим способом была секвенирована короткая ДНК фага фХ174, состоящая из 5386 ну-клеотидных пар [13].
В 1977 году Ф. Сэнгер предложил еще один способ ферментативного секвени-рования, получивший название метода терминирующих аналогов трифосфатов (метод терминаторов). Этот способ, более мощный и технологичный, несколько модифицированный, применяется до сих пор [13].
В основе метода также лежало ферментативное копирование с помощью фрагмента Кленова ДНК полимеразы I из Escherichia coli. В качестве праймеров использовали синтетические олигону-клеотиды. Специфическую терминацию синтеза обеспечивали добавлением в реакционную смесь помимо четырех
типов dNTP (один из которых был радиоактивно мечен по а-положению фосфата) еще и одного из 2′,3′-дидезоксинуклео-зидтрифосфатов (ddATP ddTTPddCTP или ddGTP), который способен включаться в растущую цепь ДНК, но не обеспечивает дальнейшее копирование из-за отсутствия 3′-ОН группы. Отношение концентраций dNTP/ddNTP авторы подбирали экспериментально, так, чтобы в итоге получить набор копий ДНК различной длины.
Таким образом, для определения первичной структуры исследуемого фрагмента ДНК требовалось провести четыре реакции копирования: по одному типу терминаторов в каждой из реакций. После этого полученные продукты разгонялись в полиакриламидном геле на соседних дорожках и по расположению полос определялась последовательность нуклеотидов (рис. 2) [14].
На выходе из сэнге-ровского секвенатора получаются короткие участки ДНК, так называемые риды (reads). Для биоинформатики принципиальны две вещи: во-первых, какой длины получаются риды, во-вторых, какие в них могут быть ошибки и как часто. Сэнгеровские риды по этим критериям очень хороши: длина -около тысячи нуклеотидов, причем качество начинает заметно падать только после 700-800 нуклеотидов.
Сам процесс секве-нирования по Ф. Сэнгеру предопределяет и эффект падения качества: труднее отличить молекулу массой 700 от таковой с массой 701, чем массой 5 от 6. Другой неприятный эффект: если в геноме встречается длинная последовательность, состоящая из одного вида нуклеотида, то трудно бывает точно определить, какой она длины (все промежуточные массы попадут в одну и ту же пробирку, не-
которые из них могут не встретиться, слиться друг с другом и т. д.). Но все же сэнгеровское секвенирование дает отличные результаты с достаточно длинными ридами.
Дешевизна, точность, а также сравнительная простота автоматизации делает этот метод своеобразным «золотым стандартом» определения последовательности нуклеотидных остатков ДНК. Именно при помощи сэнгеровского секвенирования был впервые расшифрован геном человека. Секвенирование по Ф. Сэнгеру применяется и сегодня, но его все активнее вытесняют другие методы.
В 1976 году А. Максамом и У. Гилбертом был разработан метод секвени-рования, основанный на специфической химической деградации фрагмента ДНК, радиоактивно меченого с одного конца — метод химической деградации [6]. Препарат меченой ДНК разделяли на четыре аликвоты и каждую обрабатывали реагентом, модифицирующим одно или два из четырех оснований. Авторы предложили модифицировать пуриновые основания диметилсульфа-том. При этом происходит метилирование адениновых остатков по азоту в положении 3, гуаниновых — по азоту в положении 7. Обработка образца ДНК соляной кислотой при 0°С приводит к выщеплению метиладенина. Последующая инкубация при температуре 90°С в щелочной среде вызывает разрыв сахарно-фосфатной цепи ДНК в местах выщепления оснований. Обработка пиперидином приводит к гидролизу образца по остаткам метилгуанина.
Пиримидиновые основания модифицируют гидразином. Если реакцию вести в бессолевой среде, то модифицируются как цитозин, так и тимидин; если обработку вести в присутствии 2М NaCl, то модифицируется лишь цитозин. Расщепление цепи ДНК на фрагменты и в этом случае осуществляется пиперидином. Условия реакций авторы подбирали таким образом, чтобы в итоге получить полный набор субфрагментов разной длины. Последующий электрофорез в полиакриламидном геле позволяет восстановить полную структуру исследуемого фрагмента (рис. 3) [6].
Важно отметить, что научное сообщество высоко оценило данные разработки, и в 1980 году Нобелевская премия по химии была присуждена У. Гилберту — за разработку методов секвенирования ДНК путем химической деградации, Ф. Сэн-геру — за разработку методов секвени-рования ДНК путем ферментативного построения.
Рисунок 1
Секвенирование ДНК ферментативным методом по Ф. Сэнгеру («плюс-минус» метод)
Рисунок 2
Секвенирование ДНК ферментативным методом по Ф. Сэнгеру (метод терминаторов)
Автоматическое секвенирование ДНК
В основе автоматического секвени-рования лежит уже упоминавшийся выше метод ферментативного секвенирова-ния с использованием терминирующих ddNTP. Как и классический вариант метода Ф. Сэнгера, автоматическое секвенирование включает две стадии: проведение терминирующих реакций и разделение продуктов этих реакций с помощью электрофореза. Как правило, автоматизирована лишь вторая стадия, то есть разделение меченых фрагментов ДНК в ПАА17 получение спектра эмиссии флуорофоров и последующий подсчет собранных данных [4, 10, 12].
Современные автоматизированные секвенаторы разделяют эти фрагменты, пропуская всю смесь через тончайшие капилляры, наполненные гелем. Чем короче фрагмент, тем быстрее он движется в геле по капилляру под действием электрического поля, поскольку фрагменты ДНК — по сути, ионы, движущиеся в электрическом поле от «минуса» к «плюсу». Процесс, называемый капиллярным электрофорезом, настолько эффективен, что фрагмент, только что вышедший из капилляра, оказывается ровно на один нуклеотид длиннее, чем предшествующий ему. По мере того как фрагмент появляется, он освещается лазером, что заставляет светиться меченый нуклеотид на его конце. Компьютер определяет разновидность этих нуклеотидов и регистрирует последовательность их появления, складывая «буквы» (нуклеотиды) в «текст» (последовательность ДНК). В случае расшифровки целого генома так нарабатываются миллиарды коротких «текстов», которые поступают в специальную программу, запускаемую на суперкомпьютерах. Программа находит места перекрывания «текстов» и, располагая их в нужном порядке, выстраивает полную последовательность генома.
Автоматическое секвенирование идеологически отличается от современного ему ручного секвенирования только типом используемой метки. Флуоресцентную метку включают либо в праймер, либо в терминатор транскрипции согласно следующим схемам: меченый праймер (четыре разных красителя) и немеченые терминаторы; меченый праймер (один краситель) и немеченые терминаторы; меченые терминаторы (каждый тип терминатора своим красителем) и немеченый праймер. Использование меченых праймеров предполагает проведение четырех независимых реакций (отдельно с каждым из терминаторов) для каждого секвенируемого образца. Использование
Рисунок 3
меченых терминаторов позволяет совместить все четыре реакции в одной пробирке. Если используется единственный краситель, то разделение продуктов сиквенсовой реакции в геле проводят на четырех разных дорожках. Использование четырех разных красок позволяет разгонять продукты реакции на одной дорожке [7, 17].
Флуоресценция — это излучение, сопровождающее переход электрона из возбужденного состояния в основное без изменения мультиплетности (то есть переход синглет — синглет, либо триплет — триплет). Типичное время излучения для флуоресценции -10-8с. Флуоресценции должен предшествовать обратный процесс — переход электрона в возбужденное состояние в результате поглощения (абсорбции) кванта света. Для флуоресценции доказана справедливость следующих правил: спектр испускания (эмиссии) не зависит от длины волны возбуждения; испускание сдвинуто относительно поглощения в сторону больших длин волн из-за энергетических потерь (сдвиг Стокса); спектр испускания представляет собой зеркальное отражение спектра поглощения. Для измерения флуоресценции используют время затухания флуоресценции и квантовый выход флуоресценции (отношение числа испущенных фотонов к числу поглощенных) [8].
Спектр абсорбции, равно как и спектр эмиссии, зависят от химической структуры флуорофора, а также от условий в которые молекула флуорофора помещена (рН, температура, среда и т.д.). В автоматическом секвенировании используют флуорофоры, абсорбция и излучение у которых происходит в диапазоне длин волн 450-650 нм (видимая область спектра; секвенаторы ABI и Pharmacia) и 650-825 нм (ближняя инфракрасная область спектра; секвенаторы фирмы LI-COR).
Секвенирование ДНК методом химической деградации по Максаму — Гилберту
Рисунок 4
Структурные формулы флуоресцеиновых красителей FAM и JOE
К настоящему времени синтезировано большое число разнообразных флуоресцентных красителей и постоянно продолжается работа над новыми, с улучшенными характеристиками. Помимо высокого квантового выхода флуоресценции красители должны обладать еще рядом свойств. Присоединение молекулы флуорофора способно изменять подвижность меченого фрагмента ДНК в геле. Поэтому, если используются одновременно несколько красителей, то необходимо, чтобы влияние каждого из них на подвижность было либо минимальным, либо одинаковым у всех. Выравнивание электрофоретической подвижности (когда это нужно) проводят
с помощью линкерных молекул, встраиваемых между красителем и, например, праймером. Еще одним важным моментом является условие минимального перекрывания спектров эмиссии. К сожалению, полностью избежать перекрывания спектров до сих пор не удалось. В качестве альтернативы синтезу все новых соединений недавно был предложен подход, основанный на определении не спектра, а времени флуоресценции. Правда, какого-либо практического выхода эта идея пока не получила [1, 3, 7, 10, 14].
Первыми флуорофорами, адаптированными к нуждам секвенирования, стали соединения из семейства флуоресцеино-вых (FAM, JOE) (рис. 4) и родаминовых (TAMRA, ROX, R110, R6G) красителей.
Следующее поколение флуорофоров этого семейства (dTAMRA, dROX, dR110, dR6G) получило довесок из двух остатков хлора. Это позволило несколько снизить перекрывание спектров испускания и значительно повысить интенсивность флуоресценции, значит, и чувствительность. Еще более высоким выходом флуоресценции характеризуются «трехкомпонентные» красители класса BigDye™ (Applied Biosystems) и DYEnamic™ ET (Amersham-Pharmacia-Biotech), при конструировании которых был использован принцип переноса энергии. Под переносом энергии понимают явление безизлучательного переноса энергии возбужденного состояния от донора к акцептору. Донором в BigDye™ является 4′-аминометил-5(или 6)-карбоксифлуо-ресцеин (5CF или 6CF), который связан с акцептором (представителем семейства d-родаминов) через остаток 4-аминометил-бензойной кислоты (рис. 5).
Все перечисленные красители флуоресцируют в видимой области спектра. В секвенаторах, детектирующих флуоресценцию в инфракрасной области спектра, используются красители цианинового
ряда с рабочим диапазоном 650-715 нм и 765-825 нм [1, 2, 9, 12, 16, 17].
Полимеразы, используемые при сек-венировании ДНК
В оригинальной работе Ф. Сэнгера для проведения сиквенсовых реакций был использован Кленовский фрагмент ДНК-полимеразы I из E.coli. В настоящее время для секвенирования используют рекомби-нантные ДНК-полимеразы, отвечающие следующим требованиям: отсутствие 3′- и 5′-экзонуклеазной активности, отсутствие дискриминации по включению в растущую цепь как обычных dNTP так и модифицированных (меченных) ddNTP. Выбор конкретной полимеразы зависит от условий проведения сиквенсовой реакции. Существует два разных подхода. В первом случае копирование осуществляется при 37°С высокопроцес-сивными термолабильными полимеразами (например, T7 DNA polymerase — Sequenase v1.0 и v2.0, Amersham Pharmacia Biotech). Во втором — реализуется циклический процесс, который включает денатурацию, отжиг и элонгацию, и предполагает использование термостабильных полимераз (например, ThermoSequenase™, Amersham Pharmacia Biotech и AmpliTaq FS™, PE Biosystems) [1, 2, 5, 8, 10, 15].
В настоящее время нет ни одного метода секвенирования, который работал бы для молекулы ДНК целиком; все они устроены так: сначала готовится большое число небольших участков ДНК (клонируется молекула ДНК многократно и «разрезается» в случайных местах), а потом читается каждый участок по отдельности.
Секвенирующий гель и электрофорез
Полученные в реакции секвенирования радиоактивно/флуоресцентно меченые одноцепочечные фрагменты ДНК разделяют с помощью электрофореза в полиакриламидном геле. Гели, используемые в секвенировании, должны уметь разделять фрагменты, отличаю-
щиеся друг от друга на один нуклеотид в широком диапазоне длин. Разделение должно проходить в денатурирующих условиях, препятствующих ренатурации и возникновению вторичных структур у разделяемых фрагментов. Этим требованиям удовлетворяют 5-8% полиакриламидных гелей, содержащих 7M мочевину. Обычно электрофорез проводится в трис-боратном буфере (89 тМТрис-HCl, 8,9 mM борной кислоты, 2mM ЭДТА, рН 8,0-8,5).
Для эффективного разделения напряжение должно составлять 30-50 В на см длины геля. Важным условием при проведении электрофореза является однородность температуры по всей поверхности геля. Неравномерный нагрев геля и стекол во время электрофореза способен привести к разнице в скоростях движения фрагментов ДНК по ширине геля и, как следствие, к искажению результатов. Для борьбы с этим явлением используют тонкие (обычно 0,4-0,1 мм) гели и принудительный подогрев стекол.
В автоматическом секвенировании помимо электрофореза в пластинах полиакри-ламида чрезвычайно популярен капиллярный электрофорез в линейном полиакриламиде. Капилляры представляют собой стеклянную трубку длинной 30-100 см, закатанную в полимерный пластификатор. Небольшой диаметр капилляра (50-100 мкм) позволяет проводить разделение значительно быстрее, чем в обычных гелях. Кроме того, капиллярные секвена-торы позволяют обеспечивать гораздо более высокую чувствительность за счет отсутствия горизонтальной диффузии [1, 5, 7, 8, 9, 17].
Л И Т Е Р А Т У Р А
1. Костюк С.А. Молекулярно-биологические методы в медицине: Монография. — Минск, 2013. — 327 с.
2. Bergot B.J., Chakeian M., Connell C.R., et al. — Bio-conjug Chem. — 1999. — Vol.58. — P.313-327.
3. Fiers W., Contreras R., Haegemann G, et al. — Nature. — 1978. — Vol.273. — P.113-120.
4. Ju J, Ruan C, Fuller CM, et al. // Anal. Chem. USA. — 1995. — Vol.92, N10. — P.4347-4351.
5. Lee L.G., Spurgeon S.L., HeinerC.R., et al. // Nucleic Acids Res. — 1997. — Vol.25. — P.2816-2822.
6. Maxam A.M., Gilbert W. // Proc. Natl. Acad. Sci. USA. — 1977. — Vol.74. — P.560-564.
7. MenchenS.M., LeeL.G., ConnellC.R., et al. // Nucleic Acids Res. — 1993. — Vol.51. — P.216-242.
8. Mujumdar R.B., Ernst L.A., Mujumdar S.R., et al. // Bioconjug Chem. — 1993. — Vol.4. — P.105-121.
9. Narayanan N., Little G., Lugade A., et al. — Nature. -1998. — Vol.128. — P.141-168.
10. Nunnally B.K., He H, LiL.C., et al. // Anal. Chem. USA. — 1997. — Vol.69, N13. — P.2392-2407.
11. Reddy V.B., Thimmappaya B., DharR, et al. // Science. — 1998. — Vol.200, N4341. — P.494-522.
12. Rosenblum B.B., Lee L..G., Spurgeon S.L., et al. // Nucleic Acids Res. — 1997. — Vol.25, N22. — P.4500-4504.
13. Sanger F, Coulson A.R. // J. Mol. Biol. — 1975. -Vol.94. — P.444-448.
14. Sanger F, Niclein S., Coulson A.R. // Proc. Natl. Acad. Sci. USA. — 1977. — Vol.74. — P.5463-5467.
15. Shealy D.B., Lipowska M., Lipowski J, et al. // Anal. Chem. — 1995. — Vol.67. — P.247-251.
16. Tabor S., Richardson C.C. // PNAS USA. — 1995. -Vol.92, N14. — P.6339-6343.
17. Tu 0., Knott T., Marsh M., et al. // Nucleic Acids Res. — 1998. — Vol.26, N11. — P.2797-2802.
Поступила 10.03.2017 г.