И транскрипция, и трансляция относятся к матричным биосинтезам. Матричным биосинтезом называется синтез
биополимеров (нуклеиновых кислот, белков) на матрице — нуклеиновой кислоте ДНК или РНК. Процессы матричного биосинтеза относятся к пластическому обмену: клетка расходует энергию АТФ.
Матричный синтез можно представить как создание копии исходной информации на несколько другом или новом
«генетическом языке». Скоро вы все поймете — мы научимся достраивать по одной цепи ДНК другую, переводить РНК в ДНК
и наоборот, синтезировать белок с иРНК на рибосоме. В данной статье вас ждут подробные примеры решения задач, генетический словарик пригодится — перерисуйте его себе 

Возьмем 3 абстрактных нуклеотида ДНК (триплет) — АТЦ. На иРНК этим нуклеотидам будут соответствовать — УАГ (кодон иРНК).
тРНК, комплементарная иРНК, будет иметь запись — АУЦ (антикодон тРНК). Три нуклеотида в зависимости от своего расположения
будут называться по-разному: триплет, кодон и антикодон. Обратите на это особое внимание.
Репликация ДНК — удвоение, дупликация (лат. replicatio — возобновление, лат. duplicatio — удвоение)
Процесс синтеза дочерней молекулы ДНК по матрице родительской ДНК. Нуклеотиды достраивает фермент ДНК-полимераза по
принципу комплементарности. Переводя действия данного фермента на наш язык, он следует следующему правилу: А (аденин) переводит в Т (тимин), Г (гуанин) — в Ц (цитозин).
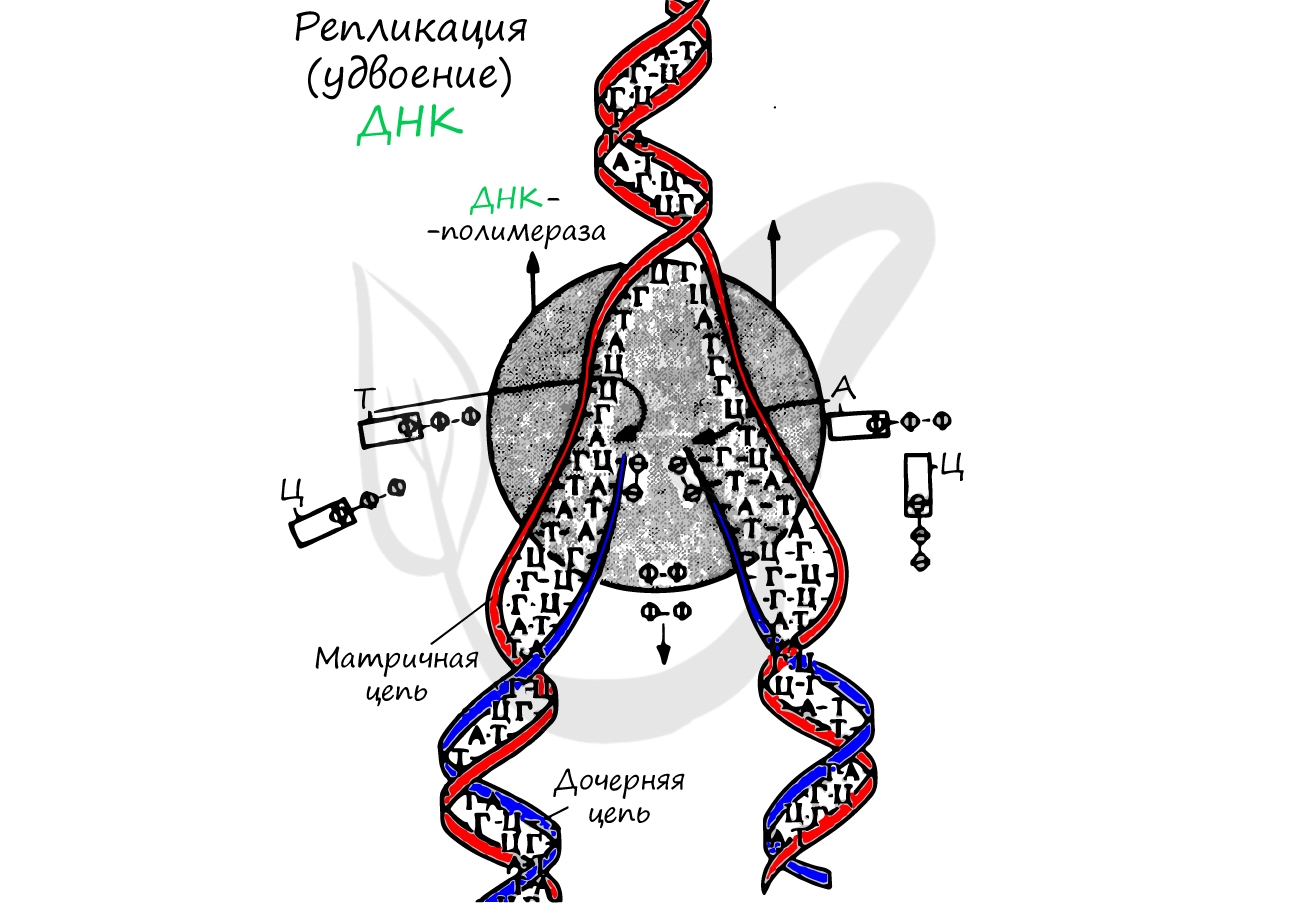
Удвоение ДНК происходит в синтетическом периоде интерфазы. При этом общее число хромосом не меняется, однако каждая из них
содержит к началу деления две молекулы ДНК: это необходимо для равномерного распределения генетического материала между
дочерними клетками.
Транскрипция (лат. transcriptio — переписывание)
Транскрипция представляет собой синтез информационной РНК (иРНК) по матрице ДНК. Несомненно, транскрипция происходит
в соответствии с принципом комплементарности азотистых оснований: А — У, Т — А, Г — Ц, Ц — Г (загляните в «генетический словарик»
выше).
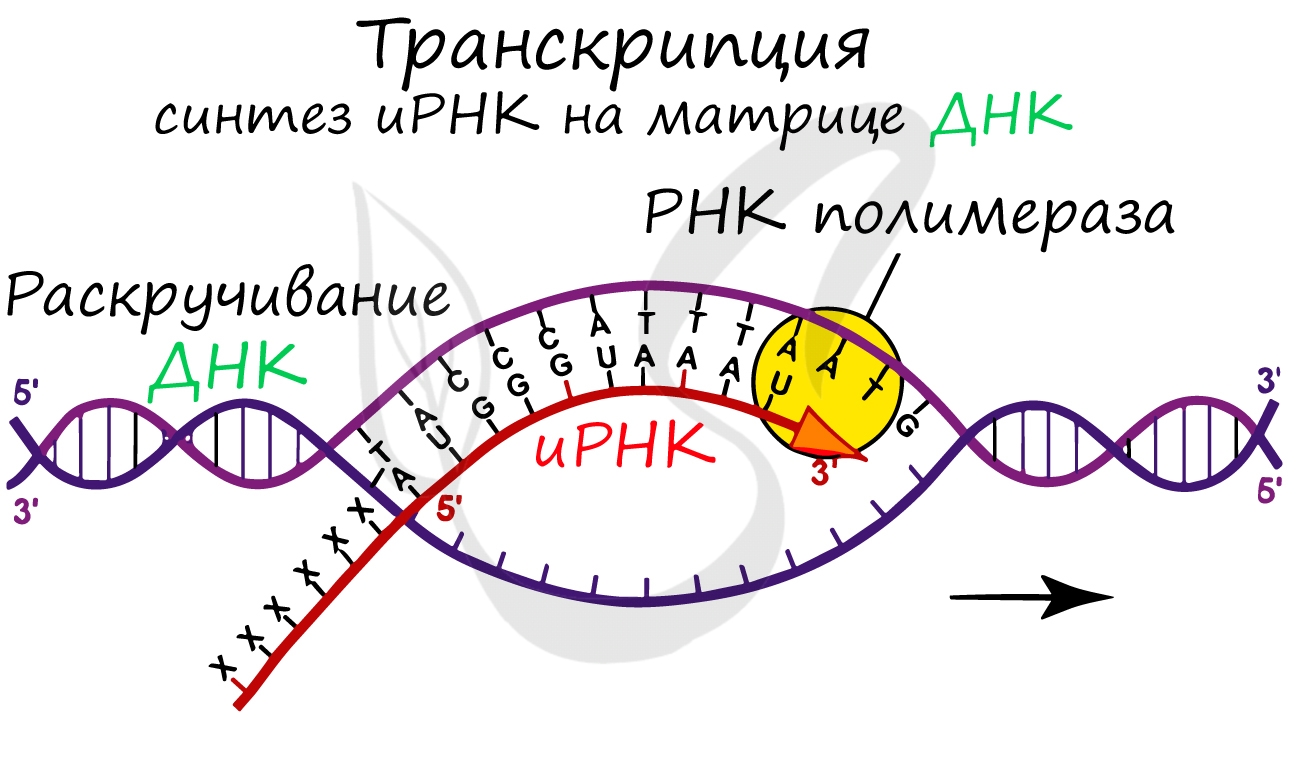
До начала непосредственно транскрипции происходит подготовительный этап: фермент РНК-полимераза узнает особый участок молекулы ДНК — промотор и связывается с ним. После связывания с промотором происходит раскручивание молекулы ДНК, состоящей из двух
цепей: транскрибируемой и смысловой. В процессе транскрипции принимает участие только транскрибируемая цепь ДНК.
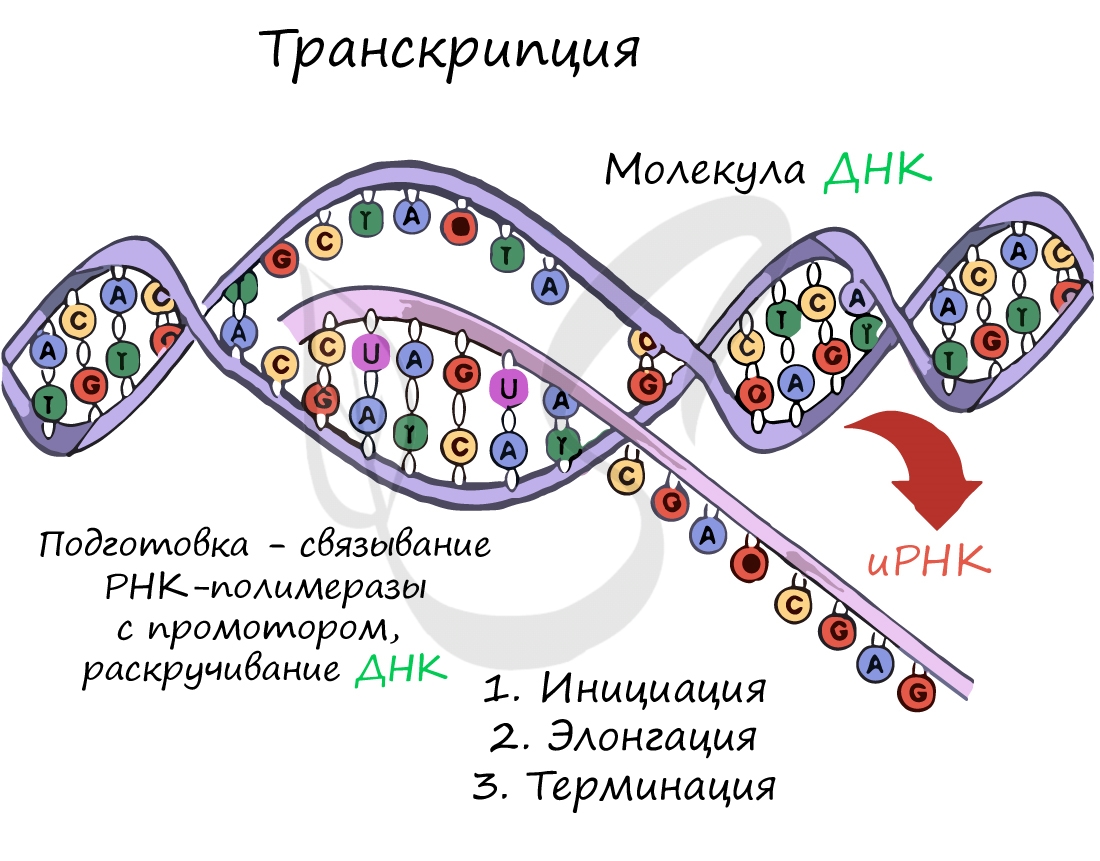
Транскрипция осуществляется в несколько этапов:
- Инициация (лат. injicere — вызывать)
- Элонгация (лат. elongare — удлинять)
- Терминация (лат. terminalis — заключительный)
Образуется несколько начальных кодонов иРНК.
Нити ДНК последовательно расплетаются, освобождая место для передвигающейся РНК-полимеразы. Молекула иРНК
быстро растет.
Достигая особого участка цепи ДНК — терминатора, РНК-полимераза получает сигнал к прекращению синтеза иРНК. Транскрипция завершается. Синтезированная иРНК направляется из ядра в цитоплазму.
Трансляция (от лат. translatio — перенос, перемещение)
Куда же отправляется новосинтезированная иРНК в процессе транскрипции? На следующую ступень — в процесс трансляции.
Он заключается в синтезе белка на рибосоме по матрице иРНК. Последовательность кодонов иРНК переводится в последовательность
аминокислот.
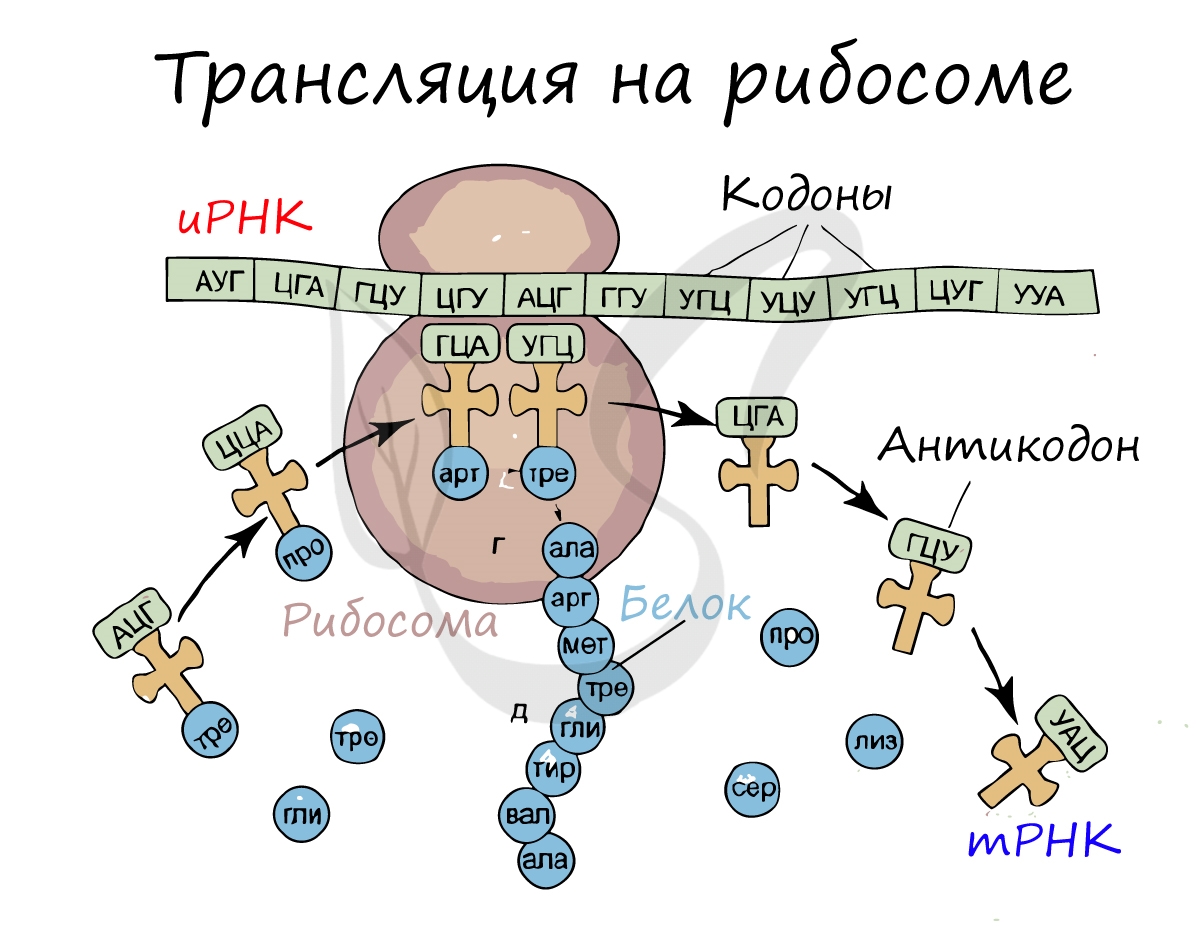
Перед процессом трансляции происходит подготовительный этап, на котором аминокислоты присоединяются к соответствующим молекулам тРНК. Трансляцию можно разделить на несколько стадий:
- Инициация
- Элонгация
- Терминация
Информационная РНК (иРНК, синоним — мРНК (матричная РНК)) присоединяется к рибосоме, состоящей из двух субъединиц.
Замечу, что вне процесса трансляции субъединицы рибосом находятся в разобранном состоянии.
Первый кодон иРНК, старт-кодон, АУГ оказывается в центре рибосомы, после чего тРНК приносит аминокислоту,
соответствующую кодону АУГ — метионин.
Рибосома делает шаг, и иРНК продвигается на один кодон: такое в фазу элонгации происходит десятки тысяч раз.
Молекулы тРНК приносят новые аминокислоты, соответствующие кодонам иРНК. Аминокислоты соединяются друг с другом: между ними образуются пептидные связи, молекула белка растет.
Доставка нужных аминокислот осуществляется благодаря точному соответствию 3 нуклеотидов (кодона) иРНК 3 нуклеотидам (антикодону) тРНК. Язык перевода между иРНК и тРНК выглядит как: А (аденин) — У (урацил), Г (гуанин) — Ц (цитозин).
В основе этого также лежит принцип комплементарности.
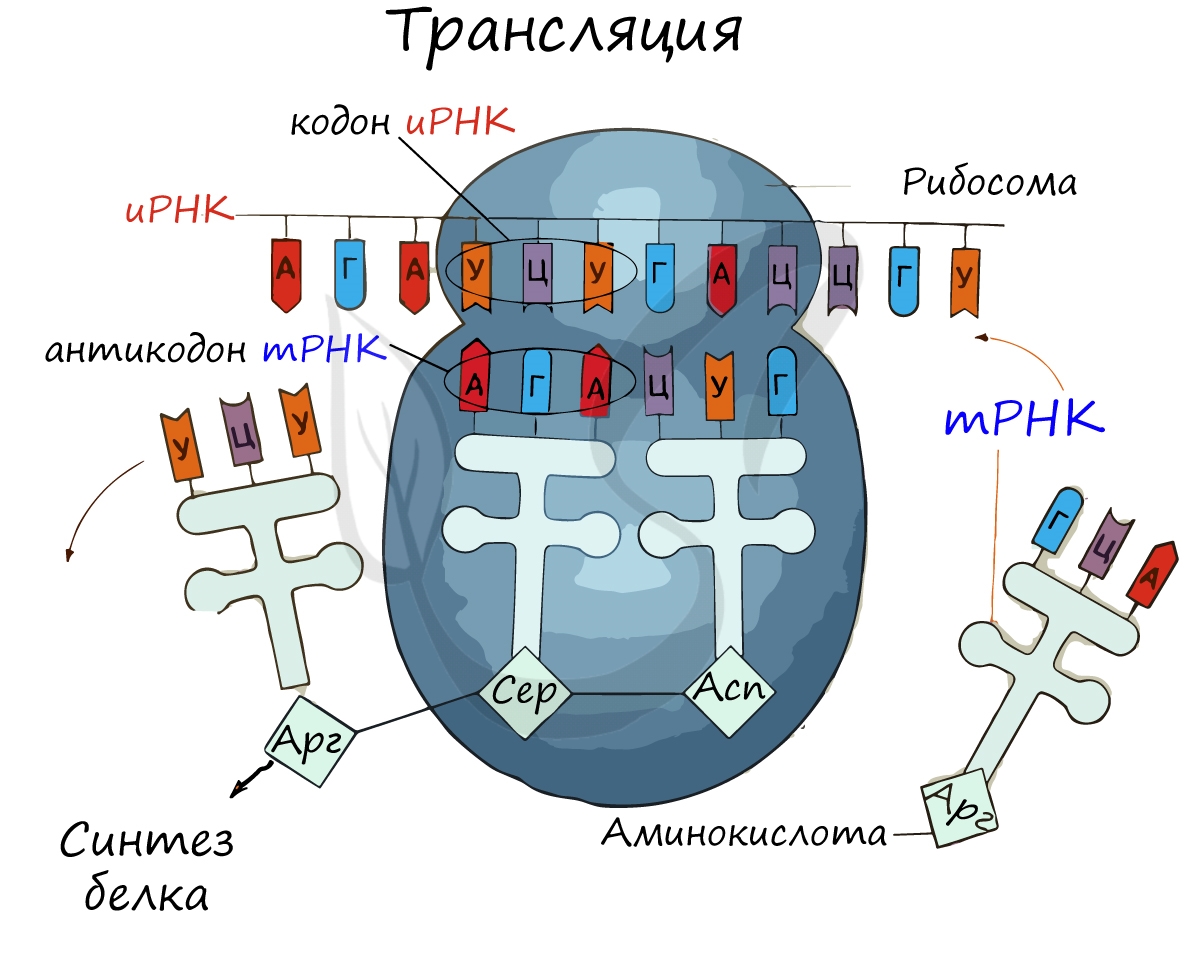
Движение рибосомы вдоль молекулы иРНК называется транслокация. Нередко в клетке множество рибосом садятся на одну молекулу
иРНК одновременно — образующаяся при этом структура называется полирибосома (полисома). В результате происходит одновременный синтез множества одинаковых белков.
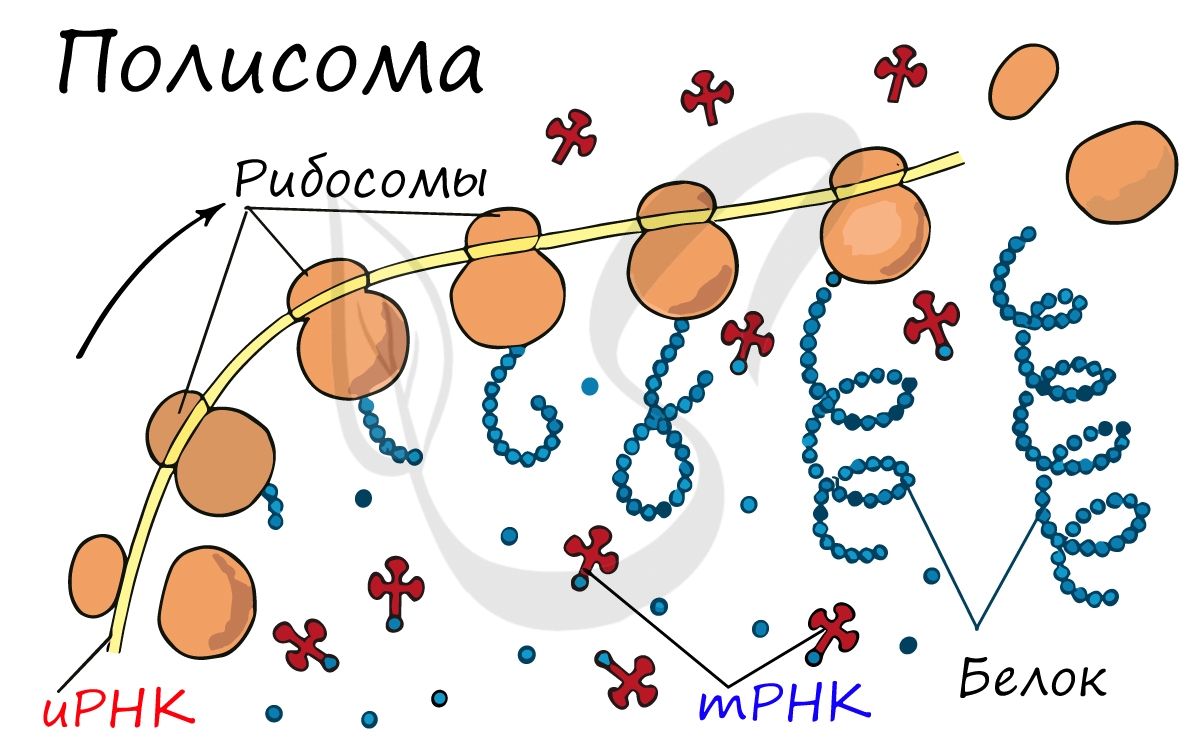
Синтез белка — полипептидной цепи из аминокислот — в определенный момент завершатся. Сигналом к этому служит попадание
в центр рибосомы одного из так называемых стоп-кодонов: УАГ, УГА, УАА. Они относятся к нонсенс-кодонам (бессмысленным), которые не кодируют ни одну аминокислоту. Их функция — завершить синтез белка.
Существует специальная таблица для перевода кодонов иРНК в аминокислоты. Пользоваться ей очень просто, если вы запомните, что
кодон состоит из 3 нуклеотидов. Первый нуклеотид берется из левого вертикального столбика, второй — из верхнего горизонтального,
третий — из правого вертикального столбика. На пересечении всех линий, идущих от них, и находится нужная вам аминокислота 
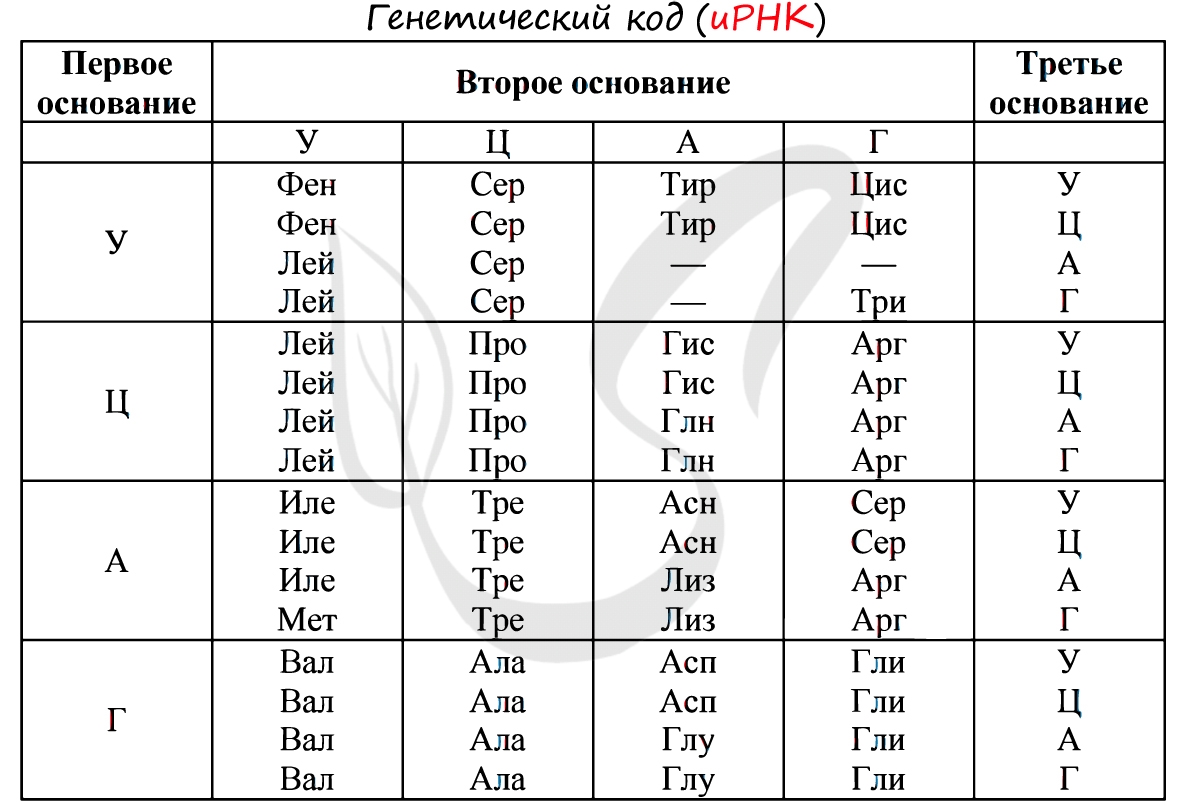
Давайте потренируемся: кодону ЦАЦ соответствует аминокислота Гис, кодону ЦАА — Глн. Попробуйте самостоятельно найти
аминокислоты, которые кодируют кодоны ГЦУ, ААА, УАА.
Кодону ГЦУ соответствует аминокислота — Ала, ААА — Лиз. Напротив кодона УАА в таблице вы должны были обнаружить прочерк:
это один из трех нонсенс-кодонов, завершающих синтез белка.
Примеры решения задачи №1
Без практики теория мертва, так что скорее решим задачи! В первых двух задачах будем пользоваться таблицей генетического кода (по иРНК),
приведенной вверху.
«Фрагмент цепи ДНК имеет следующую последовательность нуклеотидов: ЦГА-ТГГ-ТЦЦ-ГАЦ. Определите последовательность нуклеотидов
во второй цепочке ДНК, последовательность нуклеотидов на иРНК, антикодоны
соответствующих тРНК и аминокислотную последовательность соответствующего фрагмента молекулы белка, используя таблицу генетического кода»

Объяснение:
По принципу комплементарности мы нашли вторую цепочку ДНК: ГЦТ-АЦЦ-АГГ-ЦТГ. Мы использовали следующие правила при нахождении второй нити
ДНК: А-Т, Т-А, Г-Ц, Ц-Г.
Вернемся к первой цепочке, и именно от нее пойдем к иРНК: ГЦУ-АЦЦ-АГГ-ЦУГ. Мы использовали следующие правила при переводе ДНК в иРНК:
А-У, Т-А, Г-Ц, Ц-Г.
Зная последовательность нуклеотидов иРНК, легко найдем тРНК: ЦГА, УГГ, УЦЦ, ГАЦ. Мы использовали следующие правила перевода иРНК в тРНК:
А-У, У-А, Г-Ц, Ц-Г. Обратите внимание, что антикодоны тРНК мы разделяем запятыми, в отличие кодонов иРНК. Это связано с тем, что
тРНК представляют собой отдельные молекулы (в виде клеверного листа), а не линейную структуру (как ДНК, иРНК).
Пример решения задачи №2
«Известно, что все виды РНК синтезируются на ДНК-матрице. Фрагмент цепи ДНК, на которой синтезируется участок центральной петли тРНК, имеет
следующую последовательность нуклеотидов: ТАГ-ЦАА-АЦГ-ГЦТ-АЦЦ. Установите нуклеотидную последовательность участка тРНК, который синтезируется
на данном фрагменте, и аминокислоту, которую будет переносить эта тРНК в процессе биосинтеза белка, если третий триплет соответствует антикодону
тРНК»

Обратите свое пристальное внимание на слова «Известно, что все виды РНК синтезируются на ДНК-матрице. Фрагмент цепи ДНК, на которой
синтезируется участок центральной петли тРНК «. Эта фраза кардинально меняет ход решения задачи: мы получаем право напрямую и сразу
синтезировать с ДНК фрагмент тРНК — другой подход здесь будет считаться ошибкой.
Итак, синтезируем напрямую с ДНК фрагмент молекулы тРНК: АУЦ-ГУУ-УГЦ-ЦГА-УГГ. Это не отдельные молекулы тРНК (как было
в предыдущей задаче), поэтому не следует разделять их запятой — мы записываем их линейно через тире.
Третий триплет ДНК — АЦГ соответствует антикодону тРНК — УГЦ. Однако мы пользуемся таблицей генетического кода по иРНК,
так что переведем антикодон тРНК — УГЦ в кодон иРНК — АЦГ. Теперь очевидно, что аминокислота кодируемая АЦГ — Тре.
Пример решения задачи №3
Длина фрагмента молекулы ДНК составляет 150 нуклеотидов. Найдите число триплетов ДНК, кодонов иРНК, антикодонов тРНК и
аминокислот, соответствующих данному фрагменту. Известно, что аденин составляет 20% в данном фрагменте (двухцепочечной
молекуле ДНК), найдите содержание в процентах остальных нуклеотидов.

Один триплет ДНК состоит из 3 нуклеотидов, следовательно, 150 нуклеотидов составляют 50 триплетов ДНК (150 / 3). Каждый триплет ДНК
соответствует одному кодону иРНК, который в свою очередь соответствует одному антикодону тРНК — так что их тоже по 50.
По правилу Чаргаффа: количество аденина = количеству тимина, цитозина = гуанина. Аденина 20%, значит и тимина также 20%.
100% — (20%+20%) = 60% — столько приходится на оставшиеся цитозин и гуанин. Поскольку их процент содержания равен, то
на каждый приходится по 30%.
Теперь мы украсили теорию практикой. Что может быть лучше при изучении новой темы? 
© Беллевич Юрий Сергеевич 2018-2023
Данная статья написана Беллевичем Юрием Сергеевичем и является его интеллектуальной собственностью. Копирование, распространение
(в том числе путем копирования на другие сайты и ресурсы в Интернете) или любое иное использование информации и объектов
без предварительного согласия правообладателя преследуется по закону. Для получения материалов статьи и разрешения их использования,
обратитесь, пожалуйста, к Беллевичу Юрию.
From Wikipedia, the free encyclopedia
In genetics, complementary DNA (cDNA) is DNA synthesized from a single-stranded RNA (e.g., messenger RNA (mRNA) or microRNA (miRNA)) template in a reaction catalyzed by the enzyme reverse transcriptase.[1] cDNA is often used to express a specific protein in a cell that does not normally express that protein (i.e., heterologous expression), or to sequence or quantify mRNA molecules using DNA based methods (qPCR, RNA-seq). cDNA that codes for a specific protein can be transferred to a recipient cell for expression, often bacterial or yeast expression systems. cDNA is also generated to analyze transcriptomic profiles in bulk tissue, single cells, or single nuclei in assays such as microarrays, qPCR, and RNA-seq.
cDNA is also produced naturally by retroviruses (such as HIV-1, HIV-2, simian immunodeficiency virus, etc.) and then integrated into the host’s genome, where it creates a provirus.[2]
The term cDNA is also used, typically in a bioinformatics context, to refer to an mRNA transcript’s sequence, expressed as DNA bases (deoxy-GCAT) rather than RNA bases (GCAU).
Synthesis[edit]
RNA serves as a template for cDNA synthesis.[3] In cellular life, cDNA is generated by viruses and retrotransposons for integration of RNA into target genomic DNA. In molecular biology, RNA is purified from source material after genomic DNA, proteins and other cellular components are removed. cDNA is then synthesized through in vitro reverse transcription.[4]
RNA Purification[edit]
RNA is transcribed from genomic DNA in host cells and is extracted by first lysing cells then purifying RNA utilizing widely-used methods such as phenol-chloroform, silica column, and bead-based RNA extraction methods.[5] Extraction methods vary depending on the source material. For example, extracting RNA from plant tissue requires additional reagents, such as polyvinylpyrrolidone (PVP), to remove phenolic compounds, carbohydrates, and other compounds that will otherwise render RNA unusable.[6] To remove DNA and proteins, enzymes such as DNase and Proteinase K are used for degradation.[7] Importantly, RNA integrity is maintained by inactivating RNases with chaotropic agents such as guanidinium isothiocyanate, sodium dodecyl sulphate (SDS), phenol or chloroform. Total RNA is then separated from other cellular components and precipitated with alcohol. Various commercial kits exist for simple and rapid RNA extractions for specific applications.[8] Additional bead-based methods can be used to isolate specific sub-types of RNA ……(e.g. mRNA and microRNA) based on size or unique RNA regions.[9][10]
Reverse Transcription[edit]
First-strand synthesis[edit]
Using a reverse transcriptase enzyme and purified RNA templates, one strand of cDNA is produced (first-strand cDNA synthesis). The M-MLV reverse transcriptase from the Moloney murine leukemia virus is commonly used due to its reduced RNase H activity suited for transcription of longer RNAs.[11] The AMV reverse transcriptase from the avian myeloblastosis virus may also be used for RNA templates with strong secondary structures (i.e. high melting temperature).[12] cDNA is commonly generated from mRNA for gene expression analyses such as RT-qPCR and RNA-seq.[13] mRNA is selectively reverse transcribed using oligo-dT primers that are the reverse complement of the poly-adenylated tail on the 3′ end of all mRNA. An optimized mixture of oligo-dT and random hexamer primers increases the chance of obtaining full-length cDNA while reducing 5′ or 3′ bias.[14] Ribosomal RNA may also be depleted to enrich both mRNA and non-poly-adenylated transcripts such as some non-coding RNA.[15]
Second-strand synthesis[edit]
The result of first-strand syntheses, RNA-DNA hybrids, can be processed through multiple second-strand synthesis methods or processed directly in downstream assays.[16][17] An early method known as hairpin-primed synthesis relied on hairpin formation on the 3′ end of the first-strand cDNA to prime second-strand synthesis. However, priming is random and hairpin hydrolysis leads to loss of information. The Gubler and Hoffman Procedure uses E. Coli RNase H to nick mRNA that is replaced with E. Coli DNA Polymerase I and sealed with E. Coli DNA Ligase. An optimization of this procedure relies on low RNase H activity of M-MLV to nick mRNA with remaining RNA later removed by adding RNase H after DNA Polymerase translation of the second-strand cDNA. This prevents lost sequence information at the 5′ end of the mRNA.
Applications[edit]
Complementary DNA is often used in gene cloning or as gene probes or in the creation of a cDNA library. When scientists transfer a gene from one cell into another cell in order to express the new genetic material as a protein in the recipient cell, the cDNA will be added to the recipient (rather than the entire gene), because the DNA for an entire gene may include DNA that does not code for the protein or that interrupts the coding sequence of the protein (e.g., introns). Partial sequences of cDNAs are often obtained as expressed sequence tags.
With amplification of DNA sequences via polymerase chain reaction (PCR) now commonplace, one will typically conduct reverse transcription as an initial step, followed by PCR to obtain an exact sequence of cDNA for intra-cellular expression. This is achieved by designing sequence-specific DNA primers that hybridize to the 5′ and 3′ ends of a cDNA region coding for a protein. Once amplified, the sequence can be cut at each end with nucleases and inserted into one of many small circular DNA sequences known as expression vectors. Such vectors allow for self-replication, inside the cells, and potentially integration in the host DNA. They typically also contain a strong promoter to drive transcription of the target cDNA into mRNA, which is then translated into protein.
cDNA is also used to study gene expression via methods such as RNA-seq or RT-qPCR.[18][19][20] For sequencing, RNA must be fragmented due to sequencing platform size limitations. Additionally, second-strand synthesized cDNA must be ligated with adapters that allow cDNA fragments to be PCR amplified and bind to sequencing flow cells. Gene-specific analysis methods commonly use microarrays and RT-qPCR to quantify cDNA levels via fluorometric and other methods.
On 13 June 2013, the United States Supreme Court ruled in the case of Association for Molecular Pathology v. Myriad Genetics that while naturally occurring genes cannot be patented, cDNA is patent-eligible because it does not occur naturally.[21]
Viruses and retrotransposons[edit]
Some viruses also use cDNA to turn their viral RNA into mRNA (viral RNA → cDNA → mRNA). The mRNA is used to make viral proteins to take over the host cell.
An example of this first step from viral RNA to cDNA can be seen in the HIV cycle of infection. Here, the host cell membrane becomes attached to the virus’ lipid envelope which allows the viral capsid with two copies of viral genome RNA to enter the host. The cDNA copy is then made through reverse transcription of the viral RNA, a process facilitated by the chaperone CypA and a viral capsid associated reverse transcriptase.[22]
cDNA is also generated by retrotransposons in eukaryotic genomes. Retrotransposons are mobile genetic elements that move themselves within, and sometimes between, genomes via RNA intermediates. This mechanism is shared with viruses with the exclusion of the generation of infectious particles.[23][24]
See also[edit]
- cDNA library – Type of DNA library
- cDNA microarray – Collection of microscopic DNA spots attached to a solid surface
- RNA-Seq – Lab technique in cellular biology
- Real-time polymerase chain reaction – Laboratory technique of molecular biology (RT-qPCR)
References[edit]
Mark D. Adams et al. “Complementary DNA Sequencing: Expressed Sequence Tags and Human Genome Project.” Science (American Association for the Advancement of Science) 252.5013 (1991): 1651–1656. Web.
Philip M. Murphy, and H. Lee Tiffany. “Cloning of Complementary DNA Encoding a Functional Human Interleukin-8 Receptor.” Science (American Association for the Advancement of Science) 253.5025 (1991): 1280–1283. Web.
- ^ Hastings, P. J. (1 January 2001), «Complementary DNA (cDNA)», in Brenner, Sydney; Miller, Jefferey H. (eds.), Encyclopedia of Genetics, New York: Academic Press, p. 433, ISBN 978-0-12-227080-2, retrieved 29 November 2022
- ^ Croy, Ron. «Molecular Genetics II — Genetic Engineering Course (Supplementary notes)». Durham University durham.ac.uk; 20 April 1998. Archived from the original on 24 August 2002. Retrieved 4 February 2015.
- ^ Ying, Shao-Yao (1 July 2004). «Complementary DNA libraries». Molecular Biotechnology. 27 (3): 245–252. doi:10.1385/MB:27:3:245. ISSN 1559-0305. PMID 15247497. S2CID 25600775.
- ^ «5 Steps to Optimal cDNA Synthesis — US». www.thermofisher.com. Retrieved 12 May 2020.
- ^ Tavares, Lucélia; Alves, Paula M.; Ferreira, Ricardo B.; Santos, Claudia N. (6 January 2011). «Comparison of different methods for DNA-free RNA isolation from SK-N-MC neuroblastoma». BMC Research Notes. 4 (1): 3. doi:10.1186/1756-0500-4-3. ISSN 1756-0500. PMC 3050700. PMID 21211020.
- ^ R, Kansal; K, Kuhar; I, Verma; Rn, Gupta; Vk, Gupta; Kr, Koundal (December 2008). «Improved and Convenient Method of RNA Isolation From Polyphenols and Polysaccharide Rich Plant Tissues». Indian Journal of Experimental Biology. 46 (12): 842–5. PMID 19245182.
- ^ I, Vomelová; Z, Vanícková; A, Sedo (2009). «Methods of RNA Purification. All Ways (Should) Lead to Rome». Folia Biologica. 55 (6): 243–51. PMID 20163774.
- ^ Sellin Jeffries, Marlo K.; Kiss, Andor J.; Smith, Austin W.; Oris, James T. (14 November 2014). «A comparison of commercially-available automated and manual extraction kits for the isolation of total RNA from small tissue samples». BMC Biotechnology. 14 (1): 94. doi:10.1186/s12896-014-0094-8. ISSN 1472-6750. PMC 4239376. PMID 25394494.
- ^ «mRNA Isolation with Dynabeads in 15 minutes — US». www.thermofisher.com. Retrieved 20 May 2020.
- ^ Gaarz, Andrea; Debey-Pascher, Svenja; Classen, Sabine; Eggle, Daniela; Gathof, Birgit; Chen, Jing; Fan, Jian-Bing; Voss, Thorsten; Schultze, Joachim L.; Staratschek-Jox, Andrea (May 2010). «Bead Array–Based microRNA Expression Profiling of Peripheral Blood and the Impact of Different RNA Isolation Approaches». The Journal of Molecular Diagnostics. 12 (3): 335–344. doi:10.2353/jmoldx.2010.090116. ISSN 1525-1578. PMC 2860470. PMID 20228267.
- ^ Haddad, Fadia; Baldwin, Kenneth M. (2010), King, Nicola (ed.), «Reverse Transcription of the Ribonucleic Acid: The First Step in RT-PCR Assay», RT-PCR Protocols: Second Edition, Methods in Molecular Biology, Humana Press, vol. 630, pp. 261–270, doi:10.1007/978-1-60761-629-0_17, ISBN 978-1-60761-629-0, PMID 20301003
- ^ Martin, Karen. «Reverse Transcriptase & cDNA Overview & Applications». Gold Biotechnology. Retrieved 20 May 2020.
- ^ «qPCR, Microarrays or RNA Sequencing — What to Choose?». BioSistemika. 10 August 2017. Retrieved 20 May 2020.
- ^ «cDNA Synthesis | Bio-Rad». www.bio-rad.com. Retrieved 28 May 2020.
- ^ Herbert, Zachary T.; Kershner, Jamie P.; Butty, Vincent L.; Thimmapuram, Jyothi; Choudhari, Sulbha; Alekseyev, Yuriy O.; Fan, Jun; Podnar, Jessica W.; Wilcox, Edward; Gipson, Jenny; Gillaspy, Allison (15 March 2018). «Cross-site comparison of ribosomal depletion kits for Illumina RNAseq library construction». BMC Genomics. 19 (1): 199. doi:10.1186/s12864-018-4585-1. ISSN 1471-2164. PMC 6389247. PMID 29703133.
- ^ Invitrogen. «cDNA Synthesis System» (PDF). Thermofisher. Archived (PDF) from the original on 22 December 2018. Retrieved 27 May 2020.
- ^ Agarwal, Saurabh; Macfarlan, Todd S.; Sartor, Maureen A.; Iwase, Shigeki (21 January 2015). «Sequencing of first-strand cDNA library reveals full-length transcriptomes». Nature Communications. 6 (1): 6002. Bibcode:2015NatCo…6.6002A. doi:10.1038/ncomms7002. ISSN 2041-1723. PMC 5054741. PMID 25607527.
- ^ Derisi, J.; Penland, L.; Brown, P. O.; Bittner, M. L.; Meltzer, P. S.; Ray, M.; Chen, Y.; Su, Y. A.; Trent, J. M. (December 1996). «Use of a cDNA microarray to analyse gene expression patterns in human cancer». Nature Genetics. 14 (4): 457–460. doi:10.1038/ng1296-457. ISSN 1546-1718. PMID 8944026. S2CID 23091561.
- ^ White, Adam K.; VanInsberghe, Michael; Petriv, Oleh I.; Hamidi, Mani; Sikorski, Darek; Marra, Marco A.; Piret, James; Aparicio, Samuel; Hansen, Carl L. (23 August 2011). «High-throughput microfluidic single-cell RT-qPCR». Proceedings of the National Academy of Sciences. 108 (34): 13999–14004. Bibcode:2011PNAS..10813999W. doi:10.1073/pnas.1019446108. ISSN 0027-8424. PMC 3161570. PMID 21808033.
- ^ Hrdlickova, Radmila; Toloue, Masoud; Tian, Bin (January 2017). «RNA-Seq methods for transcriptome analysis». Wiley Interdisciplinary Reviews. RNA. 8 (1): e1364. doi:10.1002/wrna.1364. ISSN 1757-7004. PMC 5717752. PMID 27198714.
- ^ Liptak, Adam (13 June 2013). «Supreme Court Rules Human Genes May Not Be Patented». The New York Times. Archived from the original on 1 January 2022. Retrieved 14 June 2013.
- ^ Altfeld, Marcus; Gale, Michael Jr. (1 June 2015). «Innate immunity against HIV-1 infection». Nature Immunology. 16 (6): 554–562. doi:10.1038/ni.3157. ISSN 1529-2908. PMID 25988887. S2CID 1577651.
- ^ Havecker, Ericka R.; Gao, Xiang; Voytas, Daniel F. (18 May 2004). «The diversity of LTR retrotransposons». Genome Biology. 5 (6): 225. doi:10.1186/gb-2004-5-6-225. ISSN 1474-760X. PMC 463057. PMID 15186483.
- ^ Cordaux, Richard; Batzer, Mark A. (October 2009). «The impact of retrotransposons on human genome evolution». Nature Reviews Genetics. 10 (10): 691–703. doi:10.1038/nrg2640. ISSN 1471-0064. PMC 2884099. PMID 19763152.
External links[edit]
- H-Invitational Database
- Functional Annotation of the Mouse database
- Complementary DNA tool
- http://news.icecric.com/today-match-prediction/

В молекулярной биологии, комплементарность описывает взаимосвязь между двумя структурами, каждая из которых следует принципу блокировки и ключа. В природе комплементарность является основным принципом репликации и транскрипции ДНК, поскольку это свойство, разделяемое двумя последовательностями ДНК или РНК, так что когда они выровнены антипараллельно друг другу, нуклеотидных оснований в каждой позиции в последовательностях будут комплементарными, как если бы вы смотрели в зеркало и видели обратное. Эта комплементарная пара оснований позволяет ячейкам копировать информацию из одного поколения в другое и даже находить и устранять повреждения информации, хранящейся в последовательностях.
Степень комплементарности между двумя цепями нуклеиновой кислоты может варьироваться от полной комплементарности (каждый нуклеотид находится напротив своей противоположности) до отсутствия комплементарности (каждый нуклеотид не находится напротив своей противоположности) и определяет стабильность последовательностей, которые должны быть вместе. Кроме того, различные функции репарации ДНК, а также регуляторные функции основаны на комплементарности пар оснований. В биотехнологии принцип комплементарности пар оснований позволяет создавать ДНК-гибриды между РНК и ДНК и открывает двери для современных инструментов, таких как библиотеки кДНК. Хотя наибольшая комплементарность наблюдается между двумя отдельными цепочками ДНК или РНК, также возможно, что последовательность обладает внутренней комплементарностью, что приводит к связыванию последовательности с самой собой в свернутой конфигурации.
Содержание
- 1 Комплементарность пары оснований ДНК и РНК
- 2 Самокомплементарность и петли в виде шпилек
- 3 Регуляторные функции
- 3.1 Антисмысловые транскрипты
- 3,2 миРНК и миРНК
- 3.3 Целующиеся шпильки
- 4 Биоинформатика
- 4.1 Библиотека кДНК
- 4.2 Коды неоднозначности
- 4.3 Амбиграммы
- 5 См. Также
- 6 Ссылки
- 7 Внешние ссылки
Комплементарность пар оснований ДНК и РНК


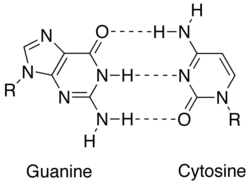
Комплементарность достигается за счет различных взаимодействий между азотистыми основаниями : аденином, тимин (урацил в РНК ), гуанин и цитозин. Аденин и гуанин — это пурины, а тимин, цитозин и урацил — пиримидины. Пурины крупнее пиримидинов. Оба типа молекул дополняют друг друга и могут образовывать пары оснований только с противоположным типом азотистых оснований. В нуклеиновой кислоте нуклеиновые основания удерживаются вместе посредством водородной связи, которая эффективно работает только между аденином и тимином и между гуанином и цитозином. Комплемент оснований A = T имеет две водородные связи, а пара оснований G≡C имеет три водородные связи. Все другие конфигурации между азотистыми основаниями будут препятствовать образованию двойной спирали. Нити ДНК ориентированы в противоположных направлениях, они называются антипараллельными.
Комплементарная цепь ДНК или РНК может быть сконструирована на основе комплементарности азотистых оснований. Каждая пара оснований, A = T по сравнению с G≡C, занимает примерно одинаковое пространство, что позволяет формировать скрученную двойную спираль ДНК без каких-либо пространственных искажений. Водородная связь между азотистыми основаниями также стабилизирует двойную спираль ДНК.
Комплементарность цепей ДНК в двойной спирали позволяет использовать одну цепочку в качестве матрицы для построения другой. Этот принцип играет важную роль в репликации ДНК, закладывая основу, объясняя, как генетическая информация может быть передана следующему поколению. Комплементарность также используется в транскрипции ДНК, которая генерирует цепь РНК из матрицы ДНК. Кроме того, вирус иммунодефицита человека, одноцепочечный РНК-вирус, кодирует РНК-зависимую ДНК-полимеразу (обратная транскриптаза ), которая использует комплементарность для катализа репликации генома.. Обратная транскриптаза может переключаться между двумя родительскими РНК геномами посредством выборочной рекомбинации во время репликации.
механизмы репарации ДНК, такие как контрольное считывание основаны на комплементарности и позволяют исправлять ошибки во время репликации ДНК путем удаления несовпадающих азотистых оснований. В общем, повреждения в одной цепи ДНК могут быть восстановлены путем удаления поврежденного участка и его замены с использованием комплементарности для копирования информации из другой цепи, как это происходит в процессах исправления несоответствия, эксцизионная репарация нуклеотидов и эксцизионная репарация оснований.
Нити нуклеиновых кислот также могут образовывать гибриды, в которых одноцепочечная ДНК может легко отжигаться с комплементарной ДНК или РНК. Этот принцип лежит в основе широко используемых лабораторных методов, таких как полимеразная цепная реакция, ПЦР.
Две цепи комплементарной последовательности обозначаются как смысловая и антисмысл. Смысловая цепь, как правило, представляет собой транскрибируемую последовательность ДНК или РНК, которая была образована при транскрипции. В то время как антисмысловая цепь — это цепь, которая комплементарна смысловой последовательности.
Самокомплементарность и шпильки

Само-комплементарность относится к тому факту, что последовательность ДНК или РНК может складываться обратно на себя, создавая двойную структуру, подобную структуре. В зависимости от того, насколько близко друг к другу находятся части последовательности, которые дополняют друг друга, нить может образовывать петли шпильки, соединения, выпуклости или внутренние петли. РНК с большей вероятностью образует такие структуры из-за связывания пар оснований, не наблюдаемого в ДНК, такого как связывание гуанина с урацилом.

Регуляторные функции
Комплементарность может быть обнаружена между короткими отрезками нуклеиновой кислоты и кодирующей областью или транскрибируемым геном и приводит к спариванию оснований. Эти короткие последовательности нуклеиновых кислот обычно встречаются в природе и выполняют регуляторные функции, такие как подавление гена.
Антисмысловые транскрипты
Антисмысловые транскрипты представляют собой участки некодирующей мРНК, которые комплементарны кодирующей последовательности. Полногеномные исследования показали, что антисмысловые транскрипты РНК обычно встречаются в природе. Обычно считается, что они увеличивают кодирующий потенциал генетического кода и добавляют общий уровень сложности к регуляции генов. На данный момент известно, что 40% генома человека транскрибируется в обоих направлениях, что подчеркивает потенциальное значение обратной транскрипции. Было высказано предположение, что комплементарные области между смысловыми и антисмысловыми транскриптами позволят генерировать двухцепочечные гибриды РНК, которые могут играть важную роль в регуляции генов. Например, индуцированная гипоксией мРНК фактора 1α и мРНК β-секретазы транскрибируются двунаправленно, и было показано, что антисмысловой транскрипт действует как стабилизатор смыслового сценария.
миРНК и миРНК

миРНК, микроРНК, представляют собой короткие последовательности РНК, которые комплементарны участкам транскрибируемого гена и выполняют регуляторные функции. Текущие исследования показывают, что циркулирующая миРНК может быть использована в качестве новых биомаркеров, следовательно, есть многообещающие доказательства для использования в диагностике заболеваний. MiRNA образуются из более длинных последовательностей РНК, которые освобождаются ферментом Dicer от последовательности РНК, происходящей из гена-регулятора. Эти короткие цепи связываются с комплексом RISC. Они совпадают с последовательностями в вышестоящей области транскрибируемого гена благодаря своей комплементарности, чтобы действовать как глушитель для гена тремя способами. Один из них заключается в предотвращении связывания рибосомы и инициации трансляции. Два — путем разрушения мРНК, с которой связан комплекс. И третья — предоставление новой последовательности двухцепочечной РНК (дцРНК), на которую Дайсер может действовать, чтобы создать больше миРНК, чтобы найти и разрушить больше копий гена. Малые интерферирующие РНК (миРНК) аналогичны функциям миРНК; они происходят из других источников РНК, но служат той же цели, что и miRNA. Учитывая их небольшую длину, правила взаимодополняемости означают, что они все еще могут быть очень разборчивыми в своих целях выбора. Учитывая, что существует четыре варианта для каждого основания в цепи и длина 20–22 пар оснований для ми / миРНК, это приводит к более чем 1 × 10 возможных комбинаций. Учитывая, что геном человека составляет ~ 3,1 миллиарда оснований в длину, это означает, что каждая миРНК должна случайно найти совпадение во всем геноме человека только один раз.
Шпильки для поцелуев
Шпильки для поцелуев образуются, когда одна цепь нуклеиновой кислоты дополняет сама себя, создавая петли РНК в форме шпильки. Когда две шпильки входят в контакт друг с другом in vivo, комплементарные основания двух нитей образуются и начинают раскручивать шпильки до тех пор, пока не образуется комплекс двухцепочечной РНК (дцРНК) или комплекс не разматывается обратно на две отдельные цепи из-за несоответствия в шпильках. Вторичная структура шпильки до поцелуя обеспечивает стабильную структуру с относительно фиксированным изменением энергии. Целью этих структур является баланс между стабильностью петли шпильки и силой связывания с дополнительной цепью. Слишком сильная начальная привязка к плохому месту и пряди не размотаются достаточно быстро; слишком слабое начальное связывание, и нити никогда не будут полностью образовывать желаемый комплекс. Эти шпильки позволяют обнажить достаточно оснований, чтобы обеспечить достаточно сильную проверку начального связывания, и достаточно слабое внутреннее связывание, чтобы позволить разворачиваться, как только будет найдено подходящее совпадение. — CG — C G — UACGGCUACGGCAGCGAAAGC UAAU CUU — CCUGCAACUUAGGCAGG — A GAA — GGACGUUGAAUCCGUCC — GAUUUUUCUCGCGCCGCGAUA UGCGC — G C — — G C — Поцелуй шпильки сходясь вверху петель. Взаимодополняемость двух головок побуждает шпильку разворачиваться и выпрямляться, превращаясь в одну плоскую последовательность из двух прядей, а не из двух шпилек.
Биоинформатика
Комплементарность позволяет хранить информацию, содержащуюся в ДНК или РНК, в одной цепи. Комплементарная цепь может быть определена из матрицы и наоборот, как в библиотеках кДНК. Это также позволяет проводить анализ, например сравнивать последовательности двух разных видов. Сокращения были разработаны для записи последовательностей при наличии несоответствий (коды неоднозначности) или для ускорения чтения противоположной последовательности в дополнении (амбиграммы).
Библиотека кДНК
A Библиотека кДНК представляет собой набор экспрессируемых генов ДНК, которые рассматриваются как полезный справочный инструмент в процессах идентификации и клонирования генов. Библиотеки кДНК конструируются из мРНК с использованием обратной транскриптазы (ОТ) РНК-зависимой ДНК-полимеразы, которая транскрибирует матрицу мРНК в ДНК. Следовательно, библиотека кДНК может содержать только вставки, которые предназначены для транскрибирования в мРНК. Этот процесс основан на принципе комплементарности ДНК / РНК. Конечным продуктом библиотек является двухцепочечная ДНК, которая может быть вставлена в плазмиды. Следовательно, библиотеки кДНК являются мощным инструментом в современных исследованиях.
Коды неоднозначности
При написании последовательностей для систематической биологии может потребоваться коды ИЮПАК, что означает «любой из двух» или «любой из трех». Код IUPAC R (любой пурин ) комплементарен Y (любой пиримидин ) и M (амино) к K (кето). W (слабый) и S (сильный) обычно не меняются местами, но в прошлом их меняли местами некоторые инструменты. W и S обозначают «слабый» и «сильный», соответственно, и указывают количество водородных связей, которые нуклеотид использует для образования пары со своим комплементарным партнером. Партнер использует такое же количество связей, чтобы создать дополняющую пару.
Код IUPAC, который специально исключает один из трех нуклеотидов, может быть комплементарным коду IUPAC, исключающему комплементарный нуклеотид. Например, V (A, C или G — «не T») может быть дополнительным к B (C, G или T — «не A»).
| Символ | Описание | Основания представлены | ||||
|---|---|---|---|---|---|---|
| A | aденин | A | 1 | |||
| C | cиттозин | C | ||||
| G | gуанин | G | ||||
| T | tгимин | T | ||||
| U | uрацил | U | ||||
| W | weak | A | T | 2 | ||
| S | strong | C | G | |||
| M | amino | A | C | |||
| K | keto | G | T | |||
| R | purine | A | G | |||
| Y | pyримидин | C | T | |||
| B | не A (B идет после A) | C | G | T | 3 | |
| D | not C (D идет после C) | A | G | T | ||
| H | не G (H идет после G) | A | C | T | ||
| V | не T (V идет после T и U) | A | C | G | ||
| Nили — | anоснование y (не пробел) | A | C | G | T | 4 |
Амбиграммы
Для создания подходящей (амбиграфической ) нотации нуклеиновой кислоты для комплементарных оснований можно использовать определенные символы (например, гуанин = b, цитозин = q, аденин = n и тимин = u), что позволяет дополнить целые последовательности ДНК, просто повернув текст «вверх ногами». Например, с предыдущим алфавитом buqn (GTCA) будет читаться как ubnq (TGAC, обратное дополнение), если перевернуть его вверх ногами.
- qqubqnnquunbbqnbb
- bbnqbuubnnuqqbuqq
Амбиграфические обозначения легко визуализируют комплементарные участки нуклеиновой кислоты, такие как палиндромные последовательности. Эта функция улучшается при использовании пользовательских шрифтов или символов вместо обычных символов ASCII или даже Unicode.
См. Также
- Базовая пара
Ссылки
Внешние ссылки
- Инструмент обратного дополнения
- Инструмент обратного дополнения @ DNA.UTAH.EDU

